Oczyszczanie i ocena genomowego DNA
Na tej stronie
Dostępność prostych metod oczyszczania DNA i RNA znacznie ułatwiła analizę i charakterystykę genomu i ekspresji genów. Istnieje zapotrzebowanie na szybką i wygodną izolację DNA i RNA z różnych źródeł komórkowych, w tym komórek i tkanek ssaków, roślin i kultur bakteryjnych. Konwencjonalne podejścia do izolacji i oczyszczania DNA opierają się na wieloetapowych procedurach obejmujących fenol/chloroform, wymianę anionową lub systemy wymiany żelu krzemionkowego. Oczyszczanie RNA zazwyczaj obejmuje sól chaotropową i fenol/chloroform z dodatkowym etapem oczyszczania oligo-dT, jeśli wymagane jest mRNA.
Jakość materiału matrycowego, który jest zawarty w reakcji PCR lub RT, ma głęboki wpływ na wiarygodność uzyskanych danych1. Bardzo ważne jest, aby jakość materiału docelowego była określana i raportowana2 i preferowane jest stosowanie kwasu nukleinowego o najwyższej możliwej jakości. Niniejszy rozdział zawiera dogłębną analizę wpływu jakości materiału docelowego na uzyskane dane qPCR i RT-qPCR z uwzględnieniem zarówno czystości, jak i integralności.
Oczyszczanie kwasu nukleinowego
Oczyszczanie kwasu nukleinowego jest pierwszym wymogiem dla większości eksperymentów wykorzystujących biologię molekularną. Próbki DNA i RNA są często uzyskiwane za pomocą "surowych" preparatów. Te metody oczyszczania mogą być szybkie i bezpośrednie i są szczególnie przydatne, gdy próbki są wykorzystywane w PCR/RT-PCR, gdzie wymagana jest tylko niewielka ilość DNA lub RNA. Niektóre zastosowania wymagają próbki oczyszczonej konwencjonalnymi metodami; ostateczne zastosowanie podyktuje najlepszą metodę do wyboru. Gdy kwas nukleinowy z materiału biologicznego musi być nienaruszony, czysty, skoncentrowany i bez inhibitorów dalszych reakcji enzymatycznych, procedury ekstrakcji wymagają dodania czynników chaotroficznych.
Środki chaotropowe
Środek chaotropowy to substancja, która denaturuje białka, DNA lub RNA poprzez zakłócenie trójwymiarowej struktury cząsteczki. Czynniki chaotropowe zakłócają stabilizujące interakcje wewnątrzcząsteczkowe, w których pośredniczą siły niekowalencyjne, takie jak wiązania wodorowe, siły van der Waalsa i efekty hydrofobowe. Powszechnie stosowane środki chaotropowe obejmują mocznik (6-8 M), guanidynę HCL (6 M) i nadchloran litu (4,5 M). Tiocyjanian guanidyny jest silnym denaturantem białek, który jest silniejszy niż HCl guanidyny i jest często stosowany podczas izolacji nienaruszonego kwasu rybonukleinowego w celu wyeliminowania aktywności RNazy. Wiele protokołów izolacji RNA obejmuje dodanie merkaptoetanolu, oprócz chaotropu, w celu zapewnienia środowiska redukującego, które denaturuje cztery mostki dwusiarczkowe utworzone między ośmioma resztami cysteiny rybonukleazy A (RNazy A). Połączenie czynnika chaotropowego i czynnika redukującego, takiego jak merkaptoetanol, powoduje rozpad rybonukleazy i całkowitą utratę jej aktywności.
Ekstrakcja genomowego DNA
Preparat surowy
Zestawy Extract-N-Amp™ mogą być używane do szybkiej i wydajnej ekstrakcji genomowego DNA (gDNA) gotowego do PCR z prawie każdego rodzaju próbki, przy użyciu prostego protokołu jednopróbkowego, który zajmuje 15 minut lub mniej, w zależności od próbki. Zestawy Extract-N-Amp zostały wykorzystane do ekstrakcji gDNA z próbek takich jak krew pełna, mieszki włosowe, skrawki ogonów myszy, komórki hodowli tkankowych, komórki wymazów z policzka, Drosophila melanogaster, różne rośliny i nasiona. W przeciwieństwie do pełnych procedur oczyszczania, podejście to pozwala uniknąć mechanicznego rozerwania, ekstrakcji organicznej, oczyszczania kolumnowego lub wytrącania DNA. Zestawy zawierają odczynniki do ekstrakcji i amplifikacji DNA, które mogą być wykorzystywane w wielu dalszych zastosowaniach, w tym w analizie PCR/qPCR.
Oczyszczanie genomowego DNA
DNA można wyizolować z niemal każdego źródła komórkowego lub tkankowego. Oczyszczanie gDNA wymaga usunięcia go z komórki przy jednoczesnej ochronie przed degradacją. Procedury izolacji muszą być również wystarczająco delikatne, aby chronić długie nici DNA przed naprężeniami mechanicznymi. Próbka jest najpierw zbierana w buforze, takim jak sól fizjologiczna buforowana fosforanami (PBS), zwykle zawierającym detergent, taki jak Triton™ X-100 lub dodecylosiarczan sodu (SDS) w celu lizy komórek i solubilizacji białek i lipidów. Proteinaza K jest dodawana w celu oddzielenia zbrylonych komórek i tkanek oraz inaktywacji enzymów poprzez trawienie proteolityczne. Kwas etylenodiaminotetraoctowy (EDTA) jest chelatorem, który działa jako zmiatacz jonów metali w roztworze i jest dodawany do buforów do oczyszczania DNA w celu usunięcia jonów magnezu (Mg2+). Mg2+ jest niezbędnym kofaktorem dla DNaz, dlatego usunięcie Mg2+ inaktywuje DNazy, zapobiegając degradacji DNA. RNaza jest często dodawana w celu degradacji RNA obecnego w komórkach.
Po lizie komórek i degradacji RNA, białek i lipidów, DNA musi zostać oddzielone od pozostałych resztek tych materiałów. Klasyczne metody oczyszczania wykorzystują fenol/chloroform3 do usuwania białek, pozostawiając DNA i inne materiały rozpuszczalne w wodzie. TRI Reagent® wykorzystuje adaptację tej metody. Alternatywnie, przy użyciu systemów takich jak zestawy do oczyszczania gDNA GenElute™, DNA jest adsorbowane na membranach na bazie krzemionki (w formatach jednokolumnowych i 96-dołkowych), w obecności soli chaotropowych, które zaburzają strukturę białka. Podczas oczyszczania DNA z materiału roślinnego, tkanka musi być najpierw mechanicznie rozbita przez mielenie w ciekłym azocie, podczas gdy pełna krew lub bakterie są wstępnie inkubowane z enzymami, aby zapewnić skuteczną lizę komórek i uwolnienie DNA. DNA jest płukane, gdy jest związane z błonami, a następnie wymywane przez zmianę stężenia soli, a następnie uwalniane za pomocą detergentu i środka chaotropowego. Białka, polisacharydy i resztki komórek są eliminowane za pomocą procedury wytrącania, po której następuje odwirowanie przez kolumnę filtracyjną.
Oczyszczanie całkowitego RNA
Oczyszczanie komórkowego RNA jest wymagane do badań ekspresji genów lub innych funkcji komórkowych, które są regulowane przez RNA. Całkowity RNA to RNA, który jest transkrybowany z komórkowego DNA (gDNA i mitochondrialnego DNA, mtDNA) i ogólnie odnosi się do próbki zawierającej: Rybosomalny, transferowy i informacyjny RNA (rRNA, tRNA i mRNA) i nie obejmuje mikroRNA (miRNA) ani mniejszych niekodujących RNA (ncRNA). Całkowity RNA jest frakcją zainteresowania, która jest izolowana do analizy ekspresji genów.
Izolacja RNA jest trudna ze względu na wszechobecność enzymów rybonukleazy (RNazy) w komórkach i tkankach, które szybko degradują RNA. Dlatego też inhibitory RNazy są włączane do procesu oczyszczania. Najpopularniejszą metodą oczyszczania jest ekstrakcja tiocyjanianem guanidyny/chloroformem. Jest to zasada działania TRI Reagent®, który jest jednofazowym roztworem zawierającym fenol i tiocyjanian guanidyny. Próbka tkanki lub komórki jest homogenizowana lub rozbijana w odczynniku TRI, chloroform jest mieszany z lizatem, a następnie mieszanina jest rozdzielana na trzy fazy przez odwirowanie. Faza wodna zawierająca RNA jest usuwana, a RNA jest wytrącane przy użyciu izopropanolu. Zestawy do izolacji RNA GenElute wychwytują RNA na żywicy krzemionkowej po lizie komórek. Zestawy GenElute wykorzystują tiocyjanian guanidyny i 2-merkaptoetanol do inaktywacji RNaz podczas izolacji całkowitego jądrowego i cytoplazmatycznego RNA. W obecności tiocyjanianu guanidyny białka łatwo rozpuszczają się, struktury komórkowe rozpadają się, a nukleoproteiny dysocjują od kwasów nukleinowych, ponieważ struktura drugorzędowa białka zostaje utracona. Ponadto, ponieważ tiocyjanian guanidyny jest silniejszy niż HCL guanidyny, RNazy są trwale denaturowane. Lizaty są odwirowywane przez kolumnę filtracyjną w celu usunięcia resztek komórkowych i ścinanego DNA. Filtrat jest następnie nakładany na kolumnę krzemionkową o dużej pojemności w celu związania całkowitego RNA, po czym następuje płukanie i elucja wodą wolną od RNazy lub buforem.
Z całkowitej próbki RNA większość stanowi rRNA (~80%), podczas gdy frakcja mRNA stanowi tylko 2-5%. W przypadku niektórych procedur może być pożądane oczyszczenie mRNA ze znacznie większej ilości rRNA i tRNA. Zestawy GenElute mRNA zapewniają wygodne procedury izolacji poliadenylowanego mRNA z wcześniej przygotowanego całkowitego RNA lub bezpośrednio z komórek i tkanek ssaków. W przypadku bezpośredniego przygotowania mRNA, komórki lub tkanki są rozbijane za pomocą trawienia SDS/proteinazą K w celu uwolnienia RNA i wyeliminowania RNaz. Oligo dT30 kowalencyjnie połączony z kulkami polistyrenowymi o średnicy 1 μm jest następnie używany do wychwytywania poliadenylowanego mRNA przez hybrydyzację. Kulki polistyrenowe pozostają zawieszone podczas hybrydyzacji, eliminując potrzebę mieszania lub kołysania. Kompleksy mRNA-kulki są przemywane na filtrze wirówkowym mikrowirówki, a następnie oczyszczone mRNA jest eluowane.
Wiele metod ekstrakcji stosowanych do oczyszczania "całkowitego RNA" specyficznie wybiera większe cząsteczki i dlatego nie nadaje się do izolacji mniejszych ncRNA, w tym miRNA. Istnieją specjalnie zaprojektowane zestawy do oczyszczania frakcji mniejszych gatunków RNA, które można wybrać, gdy są one szablonem zainteresowania.
Oczyszczanie mikro RNA (miRNA) i niekodującego RNA (ncRNA)
Oczyszczanie miRNA i innych, mniejszych cząsteczek ncRNA jest trudniejsze ze względu na ich krótkie długości. Ważne jest, aby wybrać odpowiednią metodę izolacji RNA, która zachowuje małe RNA i unikać stosowania zestawu do oczyszczania całkowitego RNA, chyba że producent wyraźnie stwierdza, że całkowite RNA obejmuje małe RNA <100 zasad. Metody ekstrakcji ncRNA są ukierunkowane na różnice między cząsteczkami RNA i wszelkimi innymi czynnikami zanieczyszczającymi, wykorzystując specyficzne wiązanie w kolumnie lub wytrącanie. RNAzol® to kwaśna mieszanina tiocyjanianu guanidyny i fenolu, która niszczy komórki i tkanki oraz, z dodatkiem chloroformu lub bromochloropropanu, oddziela wszystkie gatunki RNA od białek i DNA. Metody oczyszczania pozwalają następnie na oddzielne przygotowanie próbek małego RNA, mRNA lub całkowitego RNA, dzięki czemu każda z tych frakcji może być analizowana równolegle w dalszych aplikacjach. Zestawy miRPremier® microRNA Isolation to oparte na krzemionce zestawy bind-wash-elute zaprojektowane specjalnie do odzyskiwania małego RNA. Zestaw miRPremier wykorzystuje metodę solenia w celu usunięcia większych cząsteczek przed związaniem małego RNA z kolumną, ponieważ ilość etanolu potrzebna do związania małego RNA z krzemionką (dwutlenkiem krzemu) wytrąca również białka i inne makrocząsteczki, tworząc lepką zawiesinę, która może zatykać kolumny i uniemożliwiać skuteczne odzyskiwanie RNA. Alternatywnym podejściem do obejścia tego problemu jest zastosowanie etapu wstępnego oczyszczania w celu usunięcia makrocząsteczek, takich jak ekstrakcja za pomocą RNAzolu lub kolumny wstępnie wiążącej, tak aby można było dodać wystarczającą ilość alkoholu do związania małego RNA bez zatykania kolumny wiążącej RNA.
Ocena jakości próbki kwasu nukleinowego
.Procedury obsługi próbek i ekstrakcji kwasów nukleinowych są zmienne, dlatego kluczowe znaczenie ma zdefiniowanie wiarygodnego protokołu analizy jakości i ilości próbki oraz uwzględnienie szczegółów tej analizy w publikacjach2. Ilość RNA jest w dużej mierze określana przez wydajność rRNA, podczas gdy jakość RNA jest określana przez czystość (brak inhibitorów) i integralność cząsteczek RNA. Jednak podczas pomiaru jakości próbki czystość i integralność wpływają na wyniki; trudno jest określić czystość i integralność próbki o niskim stężeniu, a obecność zanieczyszczeń może zakłócać kwantyfikację. Istnieje wiele technik, które można wykorzystać do oceny ilości i jakości kwasów nukleinowych, ale żadna z nich nie dostarcza wszystkich informacji wymaganych do pełnego opisu próbki. Dlatego ważne jest, aby rozważyć zestaw opcji, aby upewnić się, że jakość próbki nie wpłynie na ostateczne dane analizy1.
Kwasy nukleinowe kwantyfikacji
Spektroskopia UV
Kwasy nukleinowe tradycyjnie oznaczane są ilościowo poprzez pomiar absorpcji UV za pomocą spektrofotometru. Absorbancja rozcieńczonej próbki DNA lub RNA jest odczytywana przy 260 nm i 280 nm. Gęstość optyczna (OD) przy 260 nm jest wykorzystywana do określenia stężenia RNA lub DNA w roztworze przy użyciu prawa Beera-Lamberta, które przewiduje liniową korelację między absorbancją a stężeniem.
Prawo Beera-Lamberta
A = εbc
A = absorbancja
b = długość drogi kuwety w cm (typowo 1 cm)
c = stężenie analitu
ε = współczynnik ekstynkcji
Dla RNA, ε = 40 μg/ml
Dla DNA, ε = 50 μg/ml
Odczyt OD przy 260 nm (A260) wynoszący 1.0 odpowiada w przybliżeniu 40 μg/ml czystego RNA i 50 μg/ml czystego dwuniciowego DNA. Jednak prawo Beera-Lamberta jest ważne tylko dla odczytów OD do 2, a podana zależność OD/stężenie zależy od czystości próbek. Najwyraźniej substancje zanieczyszczające o absorpcji przy 260 nm lub 280 nm wpłyną na szacowany odczyt OD. Czyste RNA ma A260/A280 równe 2,1, podczas gdy czyste DNA będzie miało A260/A280 równe 1,8.
.
Dodatkowo pH i siła jonowa buforu również zakłócają odczyt OD. Wykazano, że istnieje znaczna zmienność w stosunku A260/A280 gdy różne źródła wody są używane do wykonywania oznaczeń spektrofotometrycznych. Na przykład, zmiana pH wody od pH 5,4 do 7,5-8,5 używanej do zawieszania RNA znacznie zwiększyła stosunek RNA A260/A280 od około 1,5 do 2,05.
Ważne jest, aby zarówno stosunek A260/A280 jak i stosunek A260/A230 były bliskie 2,0 (Tabela 7.1).
Zanieczyszczenie TRIS, etanolem i izopropanolem
Efekt zanieczyszczenia TRIS: Zanieczyszczenie TRIS nie wpływa znacząco na widmo absorbancji RNA/DNA.
Efekt zanieczyszczenia etanolem: Etanol nie ma znaczącego wpływu na widmo absorbancji RNA/DNA, gdy jest mierzony w TE pH 8,0. Jednakże, gdy pomiar jest wykonywany w czystej wodzie, stosunek A260/A280 może być zmniejszony, a zatem stosunek niższy niż 2,0 może wskazywać na zanieczyszczenie etanolem.
Wpływ zanieczyszczenia izopropanolem: Izopropanol nie ma znaczącego wpływu na widmo absorbancji RNA/DNA, gdy jest mierzony w TE pH 8,0. Jednakże, gdy pomiar jest wykonywany w czystej wodzie, stosunek A260/A280 może być zmniejszony, a zatem stosunek niższy niż 2,0 może wskazywać na zanieczyszczenie izopropanolem.
Zanieczyszczenie białkiem, izotiocyjanianem guanidyny i fenolem
Efekt zanieczyszczenia białkiem: Próbka zawierająca zanieczyszczenie 0.01% BSA może wykazywać prawie normalne widmo absorbancji, chociaż stosunek A260/A280 może spaść poniżej 1,9 (RNA) lub 1,7 (DNA), co jest ostrzeżeniem, że coś zanieczyszcza próbkę. Przy 0,5% BSA widma absorbancji wyglądają nieprawidłowo i jest to wyraźne wskazanie, że w próbce występuje znaczny poziom zanieczyszczenia. Dlatego wysokie stężenia białka mogą być wykrywane przez nieprawidłowe stosunki A260/A280 ale niskie i umiarkowane ilości nie są.
Efekt zanieczyszczenia izotiocyjanianem guanidyny: Izotiocyjanian guanidyny ma niewielki wpływ na stosunek A260/A280 dlatego izotiocyjanian guanidyny ma niewielki wpływ na kwantyfikację RNA. Jednak obecność izotiocyjanianu guanidyny ma bardzo silny wpływ na stosunek A260/A230 przy zanieczyszczeniu 0,5% stosunek A260/A230 spada poniżej 0,5. Należy unikać próbek z podejrzeniem zanieczyszczenia guanidyną, ponieważ może to mieć szkodliwy wpływ na dalsze reakcje enzymatyczne.
Efekt zanieczyszczenia fenolem: Fenol ma bardzo silny wpływ na kwantyfikację RNA. Przy 0,5% zanieczyszczeniu fenolem próbki RNA o stężeniu 50 ng/μL, zmierzone stężenie jest ponad trzykrotnie wyższe. Ten silny zakłócający wpływ fenolu na kwantyfikację RNA wynika z zanieczyszczającego piku absorpcji przy 270 nm. Przy bardzo niskich stężeniach RNA, poniżej 10 ng/μL, ten zanieczyszczający pik jest często mylony z RNA. Jednak absorbancja RNA zawsze wynosi 260 nm, a nigdy 270 nm, dlatego jeśli pik występuje przy 270 nm, jest to spowodowane zanieczyszczeniem. Po wyizolowaniu RNA przy użyciu metody opartej na fenolu, błędne przeszacowanie stężenia RNA stanowi poważny problem. Dzieje się tak szczególnie w przypadku niskiego stężenia RNA.
Automatyzowana spektrofotometria UV
Podczas gdy konwencjonalna spektroforeza UV wymagała pomiarów przy użyciu stosunkowo dużych objętości próbki, obecnie dostępne są systemy takie jak NanoDrop® 2000 UV-Vis Spectrophotometer, które są zautomatyzowane i obsługują bardzo dokładne analizy próbek o bardzo małych rozmiarach. Systemy retencji próbek eliminują potrzebę stosowania kuwet i kapilar, co zmniejsza ilość analizowanej próbki do od 0,5 μL do 2 μL. NanoDrop zapewnia skanowanie absorbancji w zakresie od około 200 nm do 350 nm, który jest odpowiednim obszarem do określania stężenia i czystości RNA/DNA.
Systemy kwantyfikacji kwasów nukleinowych oparte na fluorescencji
Zastosowanie barwników fluorescencyjnych do kwantyfikacji kwasów nukleinowych jest alternatywą dla spektrofotometrii absorpcyjnej6,7. Określanie stężenia kwasów nukleinowych przy użyciu metod fluorescencyjnych wykorzystuje wiązanie małych cząsteczek lub barwników z kwasami nukleinowymi i mierzy późniejsze zmiany charakterystyki fluorescencji. Chociaż jest to droższe niż spektrofotometria absorbancji, kwantyfikacja oparta na fluorescencji jest bardziej czuła, precyzyjna i może być specyficzna dla kwasu nukleinowego będącego przedmiotem zainteresowania.
Ponieważ fluorometry mierzą fluorescencję w jednostkach względnych, a nie bezwzględnych, pomiar jest najpierw kalibrowany za pomocą znanego stężenia standardowego roztworu kwasu nukleinowego o właściwościach podobnych do mierzonej próbki. Po kalibracji pojedynczy pomiar może ustalić stężenie kwasu nukleinowego w roztworze, ale zazwyczaj wymagana jest krzywa standardowa w celu ustalenia liniowości testu w mierzonym zakresie.
Zautomatyzowane systemy, takie jak fluorometr QuBit® 2.0 (Life Technologies) mogą być używane z szeregiem różnych testów kwantyfikacji opartych na fluorescencji do pomiaru stężenia kwasu nukleinowego w roztworze. Testy wykazują szeroki zakres dynamiczny wykrywania i są w stanie dokładnie analizować małe próbki.
Należy zauważyć, że odczyt OD jest miarą absorpcji i nie może być stosowany jako pełna ocena jakości próbki. Podobnie systemy oparte na fluorescencji zapewniają pomiar ilości, a nie jakości. Na przykład nie można ich używać jako testu integralności próbki i należy również przeprowadzić odpowiednią analizę, taką jak elektroforeza kapilarna, rozdzielczość żelu lub test stosunku 3'/5' (jak opisano poniżej).
Systemy elektroforezy żelowej i kapilarnej analizy jakości kwasów nukleinowych
.Elektroforeza żelowa
RNA, choć termodynamicznie stabilny, jest szybko trawiony w obecności niemal wszechobecnych enzymów RNazy. W rezultacie krótsze fragmenty RNA często występują w próbce, co może potencjalnie zagrozić wynikom dalszych zastosowań. Proces degradacji RNA jest tylko częściowo zrozumiały i zależy od rodzaju obecnej RNazy. Ponadto jakość RNA z danej ekstrakcji może się znacznie różnić i musi być pod stałym nadzorem. Dlatego ważne jest określenie integralności próbek RNA.
W celu oceny potencjalnej degradacji zastosowano metody elektroforetyczne, które oddzielają próbki zgodnie z rozmiarem złożonych cząsteczek. W przeszłości integralność RNA oceniano za pomocą elektroforezy w żelu agarozowym barwionym bromkiem etydyny, który tworzy określony wzór pasm. Całkowity RNA przeprowadzony na denaturującym żelu agarozowym wykazuje dwa odrębne fragmenty rybosomalne odpowiadające 18S lub 28S dla eukariotycznego RNA lub 16S i 23S dla prokariotycznego RNA oraz dodatkowe fragmenty mniejszych gatunków RNA. Tradycyjnie intensywność tych pasm rRNA na denaturujących żelach agarozowych była wykorzystywana do obliczania stosunku, który służył jako wskaźnik integralności RNA. RNA uważa się za wysokiej jakości, gdy stosunek intensywności pasm eukariotycznych 28S:18S wynosi około 2,0.
Rysunek 1 przedstawia przykład oceny całkowitego RNA za pomocą elektroforezy w żelu agarozowym barwionym bromkiem etydyny. Dla
dobrej jakości całkowitego RNA, dwa największe rRNA pojawiają się jako dyskretne pasma w przybliżeniu 5 kb i 2 kb, a górne pasmo powinno mieć około dwukrotnie większą intensywność niż dolne. MRNA może pojawić się jako lekki rozmaz lub może być ledwo widoczny, głównie między dwoma pasmami rRNA.

Rysunek 1.Ocena integralności RNA za pomocą analizy żelu agarozowego. Próbki całkowitego RNA (2 μg) frakcjonowano na 1% żelu agarozowym w buforze TBE i barwiono bromkiem etydyny. Pasy 1 i 2 pokazują RNA wysokiej jakości, podczas gdy pasy 3 i 4 pokazują zdegradowane RNA.
Elektroforeza żelowa jest jednak mało czułą metodą określania integralności próbki i wymaga dużych stężeń próbki. Podczas pracy z małymi próbkami rzadko jest wystarczająca ilość, aby przeprowadzić elektroforezę żelową, a także wykonać wszystkie pożądane eksperymenty. Ponieważ podejście to opiera się na ludzkiej interpretacji obrazów żelowych, jest subiektywne, trudno porównywalne między laboratoriami, a uzyskane dane nie mogą być przetwarzane cyfrowo.
Niektóre z tych kwestii zostały rozwiązane poprzez analizę całkowitego RNA za pomocą zautomatyzowanej elektroforezy kapilarnej.
Systemy kapilarnej analizy jakości kwasów nukleinowych
Próbki kwasów nukleinowych mogą być analizowane i porównywane przy użyciu oprzyrządowania takiego jak Agilent 2100 BioAnalyzer lub Bio-Rad Experion. Korzystając z tych systemów, ślady elektroforetyczne są wykorzystywane do przypisywania numerycznych wartości integralności lub kategorii integralności.
Urządzenia te wykorzystują podejście lab-on-a-chip, łącząc elektroforezę kapilarną z detekcją fluorescencyjną. Proces elektroforetyczny przeprowadzany na chipie opiera się na tradycyjnych zasadach elektroforezy żelowej, które zostały zminiaturyzowane, co zmniejsza zużycie próbki i czas separacji.
Chip mieści studzienki na próbki, żel i zewnętrzny standard (drabina wielkości fragmentów). Podczas produkcji mikrokanały są wytwarzane w szkle w celu utworzenia wzajemnie połączonych sieci między studzienkami. Te mikrokanały są następnie wypełniane polimerem przesiewającym i barwnikiem fluorescencyjnym. Elektrody są umieszczane w studzienkach, a chip staje się zintegrowanym obwodem elektrycznym.
Naładowane biocząsteczki, takie jak DNA lub RNA, są przenoszone przez matrycę w odpowiedzi na gradient napięcia. Ze względu na stały stosunek masy do ładunku i obecność przesiewającej matrycy polimerowej, cząsteczki są rozdzielane według wielkości, tak że mniejsze fragmenty migrują szybciej niż większe.
Cząsteczki barwnika interkalują do nici kwasu nukleinowego, a kompleksy te są wykrywane przez fluorescencję indukowaną laserem. Dane są następnie przekształcane w elektroniczne obrazy przypominające żel i elektroferogramy.
Analyzer Agilent 2100 BioAnalyzer jest używany w połączeniu z zestawami RNA 6000 Nano i RNA 6000 Pico LabChip. To urządzenie bioanalityczne opiera się na połączeniu chipów mikroprzepływowych, separacji wielkości indukowanej napięciem w kanałach wypełnionych żelem i detekcji fluorescencji indukowanej laserem (LIF) w zminiaturyzowanej skali. Dwanaście próbek może być przetwarzanych sekwencyjnie przy zużyciu bardzo małych ilości każdej próbki. Cząsteczki RNA są barwione barwnikiem interkalującym i wykrywane za pomocą LIF. Dane są archiwizowane automatycznie i dostępne jako elektroferogramy, obrazy żelopodobne oraz w formacie tabelarycznym.
BioAnalyzer ma dobry liniowy zakres dynamiczny, a test RNA 6000 Nano może być stosowany do analizy próbek RNA o stężeniach od 25 ng/μL do 500 ng/μL. Chociaż dolna granica ilościowa testu RNA 6000 Nano jest określona jako 25 ng/μL, zaleca się stosowanie co najmniej 50 ng/μL w celu uzyskania znaczącego obliczenia liczby integralności RNA (RIN). W przypadku stosowania niższych stężeń można zaobserwować większe odchylenia RIN między próbkami. Test Pico może być użyty do określenia integralności RNA dla próbek o stężeniach w zakresie od 50 pg/μL do 5 ng/μL.
Oprogramowanie Agilent 2100 BioAnalyzer służy do oceny proporcji RNA wykrytego przed, pomiędzy i po pikach rRNA w celu określenia względnej liczby integralności (RIN) dla próbki RNA. Nienaruszony RNA ma RIN równy 10, podczas gdy całkowicie zdegradowany RNA ma RIN równy 1. W ten sposób interpretacja elektroferogramu jest ułatwiona i możliwe jest porównanie próbek.
Częściowo zdegradowany RNA (RIN <9) może dawać zadowalające wyniki w RT-qPCR, ale jest prawdopodobne, że zależy to od analizowanych celów, wymaganego stopnia czułości i strategii RT. Strategia RT-qPCR, która wykorzystuje dwa etapy z oligo-dT do zagruntowania odwrotnej transkrypcji i starterów PCR w pobliżu 5'-końca długiego cDNA, będzie wymagać próbek o znacznie wyższej integralności niż strategia wykorzystująca jednoetapowy RT-qPCR ze starterami specyficznymi dla genu. Wpływ różnych strategii RT i ich tolerancji na zdegradowane szablony omówiono szczegółowo (patrz Odwrotna transkrypcja). Ponadto wyższa integralność próbki będzie wymagana do wykrycia rzadkiego, a nie obfitego docelowego mRNA. Dlatego należy również wziąć pod uwagę wpływ degradacji matrycy na stosunek celów o różnej liczebności (np. genów testowych i referencyjnych). Korelacja między RIN a sukcesem RT-qPCR powinna być określona empirycznie dla każdego testu. Chociaż RIN znacznie ułatwia ocenę integralności preparatów RNA i znacznie ułatwia porównywanie próbek lub procedur izolacji RNA, nie jest jeszcze możliwe określenie z góry, czy RNA może być odpowiedni dla danego eksperymentu. Rysunek 2 przedstawia ślady Agilent BioAnalyzer dla dwóch próbek RNA. Nie wykryto różnicy w wydajności kilku rzadkich celów mRNA w cDNA wygenerowanym za pomocą jednoetapowego RT-qPCR przy użyciu starterów specyficznych dla genów. Jednak te same docelowe mRNA zostały wykryte do 2 cykli później, wskazując 4-krotnie mniej mRNA, w próbce o niższej integralności (11, RIN = 7,0) niż w próbce o wyższej jakości (9, RIN = 9,8) przy użyciu dwuetapowego RT-qPCR z oligo-dT do gruntowania RT.
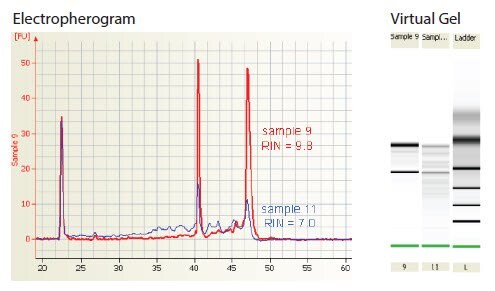
Rysunek 2Elektrofereogram pokazujący ocenę integralności RNA przy użyciu Agilent 2100 BioAnalyzer. Próbki całkowitych preparatów RNA (~150 ng) frakcjonowano na RNA 6000 Nanochip i rozdzielano za pomocą Agilent 2100. Oceniono integralność i przypisano RIN
Jakość próbek ma krytyczne znaczenie i dlatego musi zostać zweryfikowana przed użyciem próbek w testach qPCR. Wyraźnie wykazano, że analiza zdegradowanych próbek RNA może skutkować niską jakością danych1, chociaż nie zawsze tak jest8 i częściowo zależy od protokołu RT (Odwrotna transkrypcja)9.
Chociaż systemy kapilarne są znacznym ulepszeniem w stosunku do konwencjonalnej elektroforezy żelowej, są one wyspecjalizowanymi urządzeniami, drogimi i nie są przystosowane do wysokiej przepustowości. Ponadto zaobserwowano, że RIN może nie być panaceum, które byłoby pożądane. Rysunek 3 pokazuje próbki, które zostały poddane inkubacji na gołej ludzkiej skórze przez 1 minutę, nieoczekiwanie wykazując pozornie brak degradacji, gdy całkowity RNA został przeanalizowany przy użyciu Agilent BioAnalyzer. Korzystając z alternatywnej metody określania degradacji, zaobserwowano, że zawierają one zdegradowane transkrypty (patrz poniżej).

Rysunek 3.Elektroferogram całkowitego RNA (~150 ng) rozdzielonego za pomocą elektroforezy kapilarnej przy użyciu Agilent 2100 BioAnalyzer. RNA było inkubowane na nagiej ludzkiej dłoni przez 1 minutę przed analizą. Analiza RIN 10 wskazywała, że RNA było najwyraźniej wysokiej jakości i nienaruszone.
Z tego powodu ważne jest, aby zachować ostrożność przy interpretacji RIN i innych automatycznie generowanych miar integralności próbki i rozważyć dodatkowe, specyficzne dla transkryptu badanie. Jedno z takich podejść opiera się na pomiarze celu przy użyciu dwóch niezależnych testów. Test integralności polega na pomiarze stosunku ilości celu zlokalizowanego w kierunku 5' i 3' tego samego transkryptu (Rysunek 4)10. Zapewnia to względną miarę integralności RNA specyficznej dla transkryptu (protokół testu 3'/5', patrz Załącznik A).

Rysunek 4.Schemat przedstawiający test integralności 3'/5'. cDNA jest wytwarzany z całkowitego RNA przy użyciu RT z primerem oligodT. Dwa testy qPCR są zaprojektowane tak, aby celować w ten sam transkrypt, a sondy są znakowane różnymi fluoroforami, aby ułatwić wykrywanie multipleksowe. Jeśli RNA jest nienaruszony, wykrywalność testów 5' i 3' powinna być równa, natomiast jeśli RNA jest zdegradowany, wykrywalność testu 3' będzie wyższa niż testu 5'.
W ten sposób test stosunku 3'/5' jest wykorzystywany do oceny jakości próbek RNA. Zastosowanie tego podejścia zademonstrowano na Rysunku 5, testując stan transkryptu GAPDH (konkretne badane cele powinny być testowane w każdym eksperymencie) w próbce RNA, która została pokazana na Rysunku 3. Po syntezie cDNA z primerem oligo-dT, transkrypt GAPDH jest oznaczany ilościowo przy użyciu testu 5' i 3'. Jeśli RNA jest nienaruszony, oczekuje się równego stężenia przy użyciu każdego testu (stosunek = 1), natomiast jeśli RNA jest zdegradowany, oczekuje się wyższej liczby kopii dla testu 3' w stosunku do testu 5'.
Rysunek 5. Demonstracja testu 3'/5' do identyfikacji zdegradowanych próbek RNA. Chociaż analiza Agilent 2100 BioAnalyzer zwróciła RIN 10 dla obu tych próbek RNA, test 3'/5' został użyty do wykazania utraty 9 Cq (około 1000-krotnej) w teście 5' dla próbki po inkubacji przez 1 minutę na ludzkiej dłoni (degradacja 1'), podczas gdy 3' pozostała stała.
Analiza RNA przy użyciu Agilent 2100 BioAnalyzer doprowadziła do wniosku, że RNA pokazane na Rysunku 3 było nienaruszone, rejestrując RIN 10 zarówno dla RNA po 1' inkubacji na nagiej ludzkiej dłoni, jak i tej samej oczyszczonej próbki przed degradacją. Przy zastosowaniu testu 3'/5', test 3' wykazał takie samo Cq dla obu próbek, podczas gdy test 5' wykazał utratę 9 cykli, co wskazuje na 1000-krotną utratę GAPDH 5' i że transkrypt jest degradowany. Obserwacja ta potwierdza wykazanie, że status rRNA (istotny składnik algorytmu RIN) nie jest wiarygodnym wskaźnikiem integralności mRNA. Jednak wiarygodność testu 3'/5' jako miary ogólnej jakości próbki zależy od szybkości degradacji poszczególnych transkryptów11. Aby to zbadać, RNA wyekstrahowane z komórek HT29 poddano degradacji poprzez inkubację przez 72 godziny w temperaturze pokojowej lub alternatywnie przez 2 godziny w temperaturze 62 °C. Jakość wszystkich próbek analizowano przy użyciu Agilent 2100 BioAnalyzer. Testy zostały również zaprojektowane dla regionów 3' i 5' 13 wszechobecnie wyrażanych genów, a ilość tych celów została określona w oczyszczonej, wysokiej jakości niezdegradowanej próbce RNA (HT29) przy użyciu obu testów dla każdego genu (Rysunek 6A).

Rysunek 6A.Określenie jakości RNA przy użyciu testu 3'/5' a) Próbki RNA z komórek HT29 z RIN około 10 poddano odwrotnej transkrypcji przy użyciu oligo-dT i określono stosunek 3'/5' (oś y) dla każdego z 13 genów (oś X). Istnieje ogólne odchylenie 3' w kwantyfikacji, gdy oba testy są używane dla tego samego celu, a RNA jest wysokiej jakości. Stopień odchylenia różni się między genami, wpływając w ten sposób na określenie proporcji między genami, na przykład podczas normalizacji do genu referencyjnego. (Dane uprzejmie dostarczone przez prof. Stephena Bustina, Wielka Brytania).

Rysunek 6B.Próbki RNA z komórek HT29 z RIN około 7 poddano odwrotnej transkrypcji przy użyciu oligo-dT i określono stosunek 3'/5' (oś y) dla każdego z 13 genów (oś X). Istnieje silne odchylenie 3' w kwantyfikacji, gdy oba testy są używane dla tego samego celu, a RNA jest wysokiej jakości i wyraźne przesunięcie 3' w stosunku pokazanym na rysunku 7.6A. Stopień przesunięcia różni się znacznie między genami, co wskazuje, że różne geny ulegają degradacji w różnym tempie. (Dane uprzejmie dostarczone przez prof. Stephena Bustina, Wielka Brytania).

Rysunek 6C.Próbki RNA z komórek HT29 z RIN około 5 poddano odwrotnej transkrypcji przy użyciu oligo-dT i określono stosunek 3'/5' (oś y) dla każdego z 13 genów (oś X). Istnieje szeroki zakres danych dla powtórzeń. (Dane uprzejmie dostarczone przez prof. Stephena Bustina, Wielka Brytania).
Przy użyciu wysokiej jakości RNA dane dla replikacji są bardzo zbliżone, ale jasne jest, że dla wielu genów występuje odchylenie w teście do 10 razy, zwykle w kierunku 3' z bardziej dramatycznym 100-krotnym odchyleniem obserwowanym dla UBC. Może to odzwierciedlać degradację lub zróżnicowane wykrywanie przez testy z powodu składania matrycy lub innego czynnika wpływającego na RT-PCR. Niektóre cele wydają się mieć odchylenie 5' w wykrywaniu, co może być spowodowane różnicami w wydajności testu lub innymi składnikami testu RT-qPCR. Testy zostały następnie wykorzystane do ilościowego określenia tych samych celów przy użyciu cDNA z RNA o RIN 7,3 i 5,3 (odpowiednio Ryc. 6B i 6C). Wystąpiły wyraźne oznaki degradacji przy użyciu próbek RNA o umiarkowanej degradacji (RIN 7), przy czym większość genów wykazywała przesunięcie 3' od 10 do 1000 razy, wskazując na degradację tych transkryptów między testami 3' i 5'. Co ciekawe, trzy geny wykazały przesunięcie 5', co może wskazywać, że degradacja spowodowała zmianę konformacji, dzięki czemu RT lub 5' qPCR są bardziej wydajne. Ponieważ zmiany dla każdego genu są różne, oczywiste jest, że stosunek między genami mierzony przy użyciu RNA RIN 9 różniłby się znacznie od tego mierzonego przy użyciu RNA RIN 7. W przypadku stosowania RNA RIN 5 występuje wyższy stopień zmienności, a stosunek 3'/5' dla każdego genu wynosi średnio około 1. Ponieważ średnia 3'/5' = 1 byłaby również oczekiwana od wysokiej jakości nienaruszonego RNA, uznaje się, że system ten ma zastosowanie do określania integralności próbek RNA o umiarkowanym lub pozornie zerowym stopniu degradacji i jest ulepszeniem, gdy jest stosowany jako dodatek do elektroforezy żelowej lub kapilarnej.
Obserwacja odchylenia 5' w testach była intrygująca. Teoretycznie, w przypadku testów o identycznej wydajności, nie powinno być możliwe wygenerowanie większej ilości amplikonu 5' niż 3'. Najwyraźniej dodatkowe czynniki wpływają na zróżnicowaną wydajność tych testów. Kolejnym czynnikiem wpływającym na jakość próbki jest obecność zanieczyszczeń, które mogą hamować lub nawet zwiększać wydajność RT lub qPCR oraz to, czy mogą one być specyficzne dla danego celu genetycznego.
Zanieczyszczenia
.W kontekście testów PCR, istotnymi formami zanieczyszczeń są matryca kwasu nukleinowego, która jest niewłaściwie obecna w próbce, co może być również wykryte wraz z określonym celem i skutkować fałszywie dodatnim wynikiem, lub materiał, który może hamować reakcje dalszych etapów, powodując fałszywie ujemne wyniki lub niższe szacunki stężenia matrycy.
Zanieczyszczenie szablonu
Testy PCR są szczególnie podatne na zanieczyszczenie amplikonu specyficzną sekwencją docelową, ponieważ proces PCR generuje kopie amplikonu wykładniczo. W związku z tym proces przeprowadzania testu PCR wytwarza idealne zanieczyszczenie dla przyszłych testów PCR. W laboratorium biologii molekularnej konieczne jest oddzielenie przestrzeni, w której reakcja jest ustawiana i przeprowadzana, od obszaru analizy po PCR, w którym produkt PCR będzie analizowany i dalej przetwarzany. Tam, gdzie to możliwe, zaleca się korzystanie z oddzielnych pomieszczeń z dedykowanym sprzętem i fartuchami laboratoryjnymi, tak aby nic z przestrzeni analizy po PCR nigdy nie miało kontaktu z czystą przestrzenią (przed PCR). Wiele laboratoriów wprowadza również dodatkowe kontrole dostępu personelu do tych obszarów, aby zapobiec przenoszeniu reakcji. Ilościowe testy PCR są szczególnie podatne na zanieczyszczenie amplikonem, ponieważ są w stanie wykryć pojedynczą cząsteczkę matrycy, a zanieczyszczająca matryca zakłóci pomiary ilości.
Oprócz generowania określonego amplikonu metodą PCR, w konfiguracji laboratorium należy zachować ostrożność, aby zapewnić, że oryginalny materiał matrycy nie zostanie przeniesiony z jednej próbki do drugiej (zanieczyszczenie krzyżowe) lub, w przypadku analizy próbek ludzkich, że materiał nie zostanie wprowadzony przez personel wykonujący analizę.
Zaleca się przyjęcie standardowej procedury eksperymentalnej z odpowiednimi kontrolami w celu identyfikacji potencjalnych źródeł zanieczyszczenia.
Zanieczyszczenie RNA gDNA
Podczas analizy RNA ważne jest, aby upewnić się, że nie ma zanieczyszczenia gDNA. Enzymatyczne traktowanie próbek DNazą I w roztworze jest zalecane w celu usunięcia zanieczyszczającego gDNA i jest szczególnie ważne w przypadku badań genów bez intronów lub gdy projekt testu nie rozróżnia sekwencji gDNA i cDNA. Tam, gdzie to możliwe, zaleca się projektowanie testów na granicy intron/ekson, tak aby tylko przetworzone sekwencje mRNA mogły być amplifikowane i/lub wykrywane. Nie zawsze zapobiega to amplifikacji z gDNA, ponieważ istnieją sekwencje, które istnieją jako przetworzone pseudogeny, które są skutecznie genomowymi sekwencjami cDNA (patrz PCR/qPCR/dPCR Assay Design). Warto również wziąć pod uwagę, że DNA występuje w odczynnikach i potencjalnie może powodować zanieczyszczenie12.
.Inhibicja
Obecność inhibitorów w różny sposób wpływa na testy qPCR dla różnych celów13. Dlatego obecność inhibitorów w próbce może powodować złożone zakłócenia prawdziwych danych. Ponieważ każdy test ma inny wpływ, wynikowe stosunki między genem zainteresowania (GOI) a ilościami genów referencyjnych mogą nie odzwierciedlać rzeczywistej obfitości każdego genu, co prowadzi do nieprawidłowych szacunków względnych ilości docelowych lub trudności w interpretacji testów genotypowania.
Inhibitory PCR, które są powszechnie wprowadzane podczas procesów eksperymentalnych obejmują; tris, etanol, izopropanol, EDTA, enzym odwrotnej transkryptazy, izotiocyjanian guanidyny i fenol.
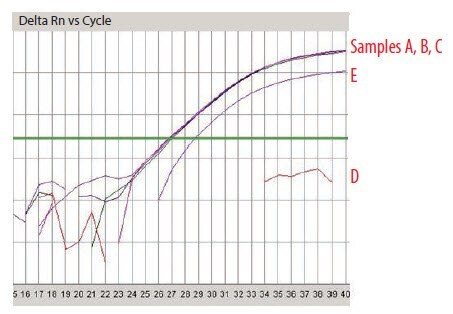
Jednym z systemów, który może być stosowany do wykrywania inhibitorów jest test SPUD14 (Rysunek 7). Stosując tę metodę, sztuczny amplikon (pochodzący z sekwencji kwasu nukleinowego ziemniaka pozbawionej homologii z jakąkolwiek inną znaną sekwencją) jest poddawany qPCR w obecności wody (kontrola amplifikacji; Rysunek 7 próbka a) lub próbki testowej (Rysunek 7 próbki b i c). Jeśli próbka testowa nie zawiera inhibitora testu SPUD, cykl kwantyfikacji (Cq) zarejestrowany dla kontroli amplifikacji i próbki testowej będzie taki sam. Jeśli jednak próbka testowa zawiera inhibitor, wartość Cq zostanie zwiększona (szczegółowy protokół testu jest dostępny jako SPUD Protocol, Appendix A).

Rysunek 7.7.Przedstawienie testu SPUD. W tym przypadku różnica w Cq między próbką c a próbką kontrolną a wskazuje na obecność inhibitora.
Zintegrowany przykład: Analiza jakości próbki RNA
Każda z opisanych wcześniej metod kontroli jakości próbki służy do określenia konkretnej cechy próbki. Ta sekcja zawiera opis przykładowego przepływu pracy, który można zastosować do próbek, wraz z opisem danych dostarczanych przez każdy test kontroli jakości. Przedstawione badanie nie jest ani wyczerpujące, ani nie stanowi ostatecznej standardowej procedury operacyjnej; jest jedynie przykładem możliwej procedury kontroli jakości.
Przygotowano pięć próbek RNA i oznaczono je kolejno od A do E. Zostały one przeanalizowane przy użyciu NanoDrop i stwierdzono, że mają bardzo podobne stężenie (około 100 ng/μL). Stosunek A260/A280 został zarejestrowany dwukrotnie przy użyciu dwóch niezależnych odczytów dla każdej próbki (Rysunek 8) i stwierdzono, że wynosi około 1.9 we wszystkich przypadkach, co sugeruje próbki wysokiej jakości i bez zanieczyszczeń, które zostałyby wykryte przy A280 nm, warto zauważyć, że odczyt A230 nm jest nieobecny w tej analizie. Analiza przy użyciu elektroforezy kapilarnej (Agilent 2100 BioAnalyzer) ujawnia inny obraz (Rysunek 9). Próbki A i B są wysokiej jakości (RIN 10 i stężenie 110 ng/μL zgodnie z odczytem NanoDrop), próbka C jest wysoce zdegradowana (RIN 2,4, stężenie 62 ng/μL jest o połowę niższe niż stężenie określone przez NanoDrop), a próbki D i E są wysokiej jakości (odpowiednio RIN 9,1 i 9,5), ale o niższym stężeniu niż próbki A i B (odpowiednio 30 ng/μL i 43 ng/μL). Informacje z dwóch metod pomiaru w pewnym stopniu się uzupełniają, ponieważ NanoDrop nie ujawnia degradacji. Jest to jednak również sprzeczne, ponieważ stężenia próbek D i E znacznie się różnią. Byłoby to powodem do niepokoju i powinno wywołać dalsze badania. Chociaż elektroforeza kapilarna jest potężnym narzędziem do oceny próbek, ma stosunkowo niską przepustowość i nie jest dostępna dla wszystkich badaczy. Alternatywnym testem integralności próbki jest analiza 3'/5'. Test ten został zastosowany do próbek A-E przy użyciu równych stężeń zgodnie z odczytem NanoDrop. Gdy próbki te zostały przetestowane przy użyciu tego podejścia, było jasne, że RNA próbek A (Rysunek 10A) i B (dane nie pokazane, identyczne z próbką A) były nienaruszone, podczas gdy próbka C była wysoce zdegradowana (Rysunek 10B). Należy zauważyć, że dla próbki C wartość Cq zarówno dla testów 5', jak i 3' przesunęła się znacząco do wyższych wartości, wykazując utratę określonego celu w stosunku do próbek A i B. Profil dla próbek D i E nie był zgodny z oczekiwaniami (Rysunek 10C pokazuje próbkę D, dane nie są pokazane dla próbki E, ale były podobne), ponieważ test 5' ma niższe Cq niż 3', co wskazuje na więcej sygnału 5' niż 3' w próbce cDNA wytworzonej z primingu oligo-dT. Alternatywnym wyjaśnieniem jest to, że próbki te zawierają inhibitor, który wpływa na test 3' bardziej niż 5'. Aby przetestować tę teorię, test SPUD został przeprowadzony na wszystkich próbkach (Rysunek 11). Nie ma dowodów na obecność inhibitora w próbkach od A do C, ale zarówno próbki D, jak i E wykazują inhibicję testu SPUD, przy czym próbka D powoduje całkowite niepowodzenie testu.
Wykorzystując wszystkie informacje, można stwierdzić, że w każdej próbce było równe stężenie materiału kwasu nukleinowego (NanoDrop), a próbki zawierały niewykrywalne zanieczyszczenia, które absorbują przy 280 nm (białko) (inne długości fal nie były testowane). Próbki A i B miały wysoką integralność bez wykrytych inhibitorów, próbka C była wysoce zdegradowana (elektroforeza kapilarna i test 3'/5') bez wykrytych inhibitorów, a próbki D i E były nienaruszone, ale zawierały inhibitory PCR (3'/5' i SPUD).

Rysunek 8.Ocena jakości NanoDrop (A260/A280).

Rysunek 9.Agilent 2100 BioAnalyzer Analiza próbek RNA A-E.
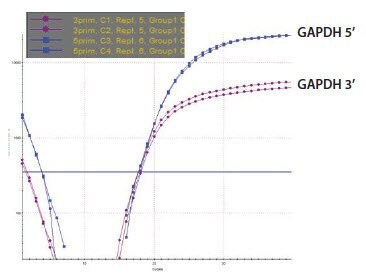
Rysunek 10A.GAPDH 3'/5' Multiplex Assay Próbka RNA.

Rysunek 10B.GAPDH 3'/5' Multiplex Assay Próbka C.

Rysunek 10C.GAPDH 3'/5' Multiplex Assay Próbka D.
Rysunek 11.Analiza SPUD próbek A-E.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?