Wpływ metody oczyszczania GenElute™-E na dokładność kwantyfikacji DNA i dalsze procesy enzymatyczne
Takie metody wymywania i elucji są żmudne i wymagają wielu etapów płukania i wirowania. Powtarzające się etapy wirowania mogą powodować znaczne ścinanie DNA. Dodatkowo, sole chaotropowe i inne zanieczyszczenia mogą łatwo przedostać się do eluowanego DNA lub RNA, wpływając na końcową czystość i oznaczenie ilościowe, a także na dalsze procesy enzymatyczne, takie jak PCR.
Oceniliśmy nowy system oczyszczania kwasów nukleinowych z pojedynczym wirowaniem, który eliminuje potrzebę wysokiego wiązania soli i etapów płukania etanolem. Zestawy do oczyszczania DNA i RNA GenElute™-E wykorzystują metodę chromatografii ujemnej zależnej od wykluczenia wielkości w celu oddzielenia dużych cząsteczek kwasu nukleinowego DNA i RNA od mniejszych białek, lipidów i składników jonowych w próbkach komórek, tkanek, krwi i innych (Rysunki 1-2).

Rysunek 1. Oczyszczanie kwasów nukleinowych przy użyciu technik chromatografii negatywnej (wykluczania wielkości).

Rysunek 2.Elucja oczyszczonego DNA i zatrzymanie białek, lipidów i innych cząsteczek przy użyciu zestawu GenElute™-E Single Spin Blood DNA High Yield Kit z próbkami krwi myszy. A) Widma absorbancji oczyszczonego genomowego DNA (czerwona linia). B) Widma absorbancji retentatu kolumny wirówkowej GenElute™-E (czarna linia) nałożone na widma oczyszczonego DNA (czerwona). Widmo retentatu pokazuje białko i inne zanieczyszczenia z OD przy ~280nm lub ≤240nm. Zwróć uwagę na zmianę skali na osi y.
Oceniliśmy wpływ metody oczyszczania kwasów nukleinowych na zanieczyszczenia zakłócające kwantyfikację i dalsze procesy enzymatyczne. Do tych badań porównawczych genomowe DNA przygotowano przy użyciu technologii oczyszczania jednoskrętnego GenElute™-E lub klasycznej technologii bind-wash-elute spin prep opartej na krzemionce od dostawcy X. Czystość kwasu nukleinowego oceniano przy użyciu trzech metod:
- spektrofotometria UV (stosunek gęstości optycznej mierzony przez A260nm/280nm i A260nm/230nm)
- Elektroforeza żelowa
- Quantitative PCR (qPCR)
Wyniki wskazują, że systemy oczyszczania GenElute™-E z pojedynczym wirowaniem DNA i RNA zapewniają wyższą czystość niż obecne techniki przygotowania wirowania krzemionkowego, co prowadzi do dokładniejszych oznaczeń ilościowych i zmniejszenia zakłóceń ze strony zanieczyszczeń w dalszych procesach enzymatycznych, takich jak PCR.
Evaluation of Impurities Introduced During Nucleic Acid Preparation
Interference with Quantitation by Ultraviolet (UV) Spectrophotometry
Wspólne zanieczyszczenia biologiczne, takie jak białka, pofragmentowane kwasy nukleinowe, ssDNA, RNA (w przypadku pomiaru DNA), startery i sole chaotropowe z protokołów ekstrakcji i oczyszczania mogą prowadzić do przeszacowania stężenia kwasu nukleinowego. Niektóre z tych zanieczyszczeń można zmierzyć za pomocą spektrofotometrii w ultrafiolecie (UV). W tej metodzie światło UV przepuszczane jest przez oczyszczoną próbkę DNA lub RNA. Absorbancja przy 260 nm (A260) jest używana do pomiaru kwasu nukleinowego. Absorbancja przy 280 nm (A280) jest używana do pomiaru zanieczyszczeń białkowych w próbce. Stosunek A260/A280 można wykorzystać do oszacowania czystości próbki w odniesieniu do zanieczyszczeń białkowych. DNA o wysokiej czystości będzie miało stosunek A260/A280 ≥ 1,8. Niższy stosunek wskazuje na zanieczyszczenie białkami w końcowym preparacie.
Absorbancja przy 230 nm (A230) może być wykorzystana do identyfikacji obecności zanieczyszczeń chemicznych, takich jak sole chaotroficzne. Stosunek A260/A230 powinien być idealnie większy niż 2,0 dla minimalnej interferencji chemicznej w procesach enzymatycznych, takich jak PCR. Ocena tego stosunku ma kluczowe znaczenie, gdy wymagane są próbki o wysokiej czystości.
Widmo absorbancji, stosunek A260/280 oraz A260/230 zostały ocenione dla systemów oczyszczania jednoniciowego DNA i RNA GenElute™-E przy użyciu próbek krwi. Wyniki porównano z wynikami uzyskanymi z oczyszczania tych samych próbek przy użyciu konkurencyjnego produktu (Rysunek 3 i Tabela 1). Dane sugerują, że systemy oczyszczania GenElute™-E zapewniają wyższą czystość genomowego DNA przy mniejszej ilości zanieczyszczeń biologicznych i chemicznych w końcowym przygotowaniu próbki w porównaniu z metodami przygotowania krzemionkowego bind-wash-elute.

Rysunek 3. Widma absorpcji UV genomowego DNA przygotowanego z krwi przy użyciu A) zestawu spin prep do oczyszczania DNA na bazie krzemionki od dostawcy X lub B) zestawów do oczyszczania DNA do chromatografii jednospinowej ujemnej GenElute™-E. Wstawiony wykres zmierzony przy 10-krotnym stężeniu eluowanej frakcji. Zanieczyszczenia są wykrywane wyłącznie we frakcjach krzemionkowych. Czarne linie przedstawiają widma dla próbki oczyszczonej przy użyciu wskazanej metody z jakimkolwiek wpływem zanieczyszczeń procesowych. Czerwone linie to widma dla oczyszczonej próbki bez zanieczyszczeń procesowych, używane jako kontrole bazowe. Pomarańczowe linie to widma dla metody oczyszczania bez użycia rzeczywistej próbki, pokazujące wpływ zanieczyszczeń procesowych na odczyt spektrofotometryczny.
Zanieczyszczenia DNA i kwasów nukleinowych o niskiej masie cząsteczkowej oceniane za pomocą elektroforezy żelowej
Rozdzielenie wielkości za pomocą elektroforezy żelowej zapewnia wizualną i ilościową ocenę czystości próbki. Większe genomowe DNA migruje wolniej niż mniejsze fragmenty DNA i RNA. Uzyskane pasma można obserwować lub określać ilościowo za pomocą barwnika fluorescencyjnego.
Oczyszczone próbki DNA przygotowane z tkanki wątroby myszy przy użyciu zestawów spin prep na bazie krzemionki (Dostawca X) lub chromatografii ujemnej przez wykluczenie wielkości (zestawy do oczyszczania pojedynczego spin DNA GenElute™-E) zostały rozdzielone na żelu agarozowym. SYBR green został użyty jako barwnik kwasu nukleinowego. Aby znormalizować wyniki, ta sama masa DNA z każdej oczyszczonej próbki została załadowana na każdy pas żelu. Wizualizacja żelu agarozowego na transiluminatorze wykazała, że ciemniejsze, pojedyncze pasma genomowego DNA uzyskano przy użyciu chromatografii ujemnej (wykluczenie wielkości) w porównaniu z metodą oczyszczania opartą na krzemionce (Ryc. 4).
Próbki eluowane z kolumn spinowych z membraną krzemionkową wykazywały słabsze pasma na dolnym końcu żelu, zgodne z zanieczyszczeniem małych cząsteczek RNA lub ściętego DNA w końcowym przygotowaniu. Ponieważ obciążenie żelu zostało znormalizowane względem DNA w próbce (określonego przez absorbancję przy 260 nm), dane te sugerują, że wydajność genomowego DNA jest zawyżona za pomocą tej metody, ponieważ zanieczyszczające mniejsze cząsteczki kwasu nukleinowego również przyczyniają się do pomiaru absorbancji. Pojedyncze pasmo DNA o większej intensywności uzyskane przy użyciu metody chromatografii negatywnej GenElute™-E (wykluczanie wielkości) sugeruje większą czystość genomowego DNA z tych próbek tkanek. Wyniki zostały określone ilościowo przy użyciu metod fluorometrycznych (Rysunek 5).
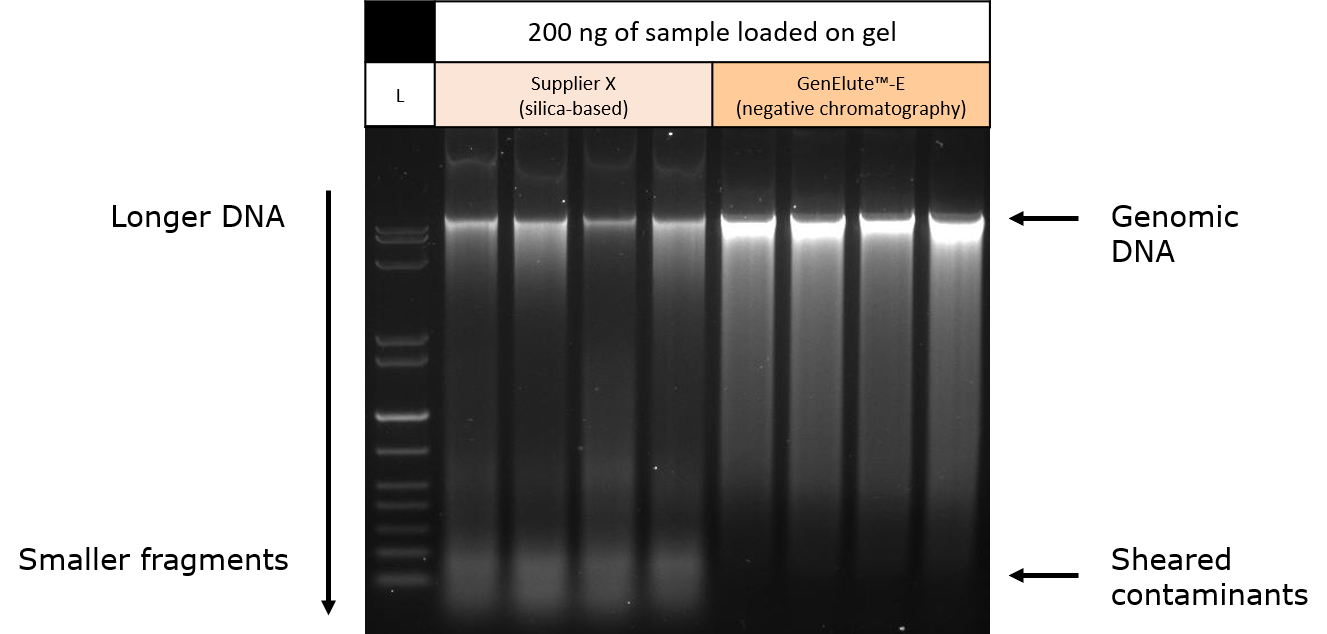
Rysunek 4. Elektroforeza żelowa oczyszczonego genomowego DNA z próbek tkanki wątroby myszy. Tkankę myszy oczyszczono przy użyciu metod spin prep opartych na krzemionce (Dostawca X) lub chromatografii ujemnej poprzez wykluczenie wielkości (zestawy do oczyszczania GenElute™-E single-spin DNA). Próbki dostosowano tak, aby ta sama ilość DNA została załadowana na każdy pas żelu agarozowego. Wyniki sugerują obecność zanieczyszczającego RNA lub ścinanego DNA w próbkach oczyszczonych metodą opartą na krzemionce.

Rysunek 5. Obliczona wydajność genomowego DNA (gDNA) i ilości zanieczyszczającego RNA lub ścinanego DNA z próbek tkanki wątroby myszy. Próbki oczyszczono przy użyciu metod spin prep opartych na krzemionce (Dostawca X) lub chromatografii ujemnej poprzez wykluczenie wielkości (zestawy do oczyszczania GenElute™-E single-spin DNA). Ilość DNA obliczono poprzez ilościowe oznaczenie sygnału fluorescencyjnego z zielonego barwnika SYBR. Wyniki wskazują na niewielkie, ale znaczące ilości zanieczyszczeń RNA lub ściętego DNA w próbkach przygotowanych przy użyciu kolumn spinowych do oczyszczania na bazie krzemionki.
Interferencje w qPCR
Metody oczyszczania oparte na krzemionce wprowadzają do oczyszczanej próbki sole denaturujące i rozpuszczalniki organiczne, takie jak etanol. Zanieczyszczenia te mogą łatwo przenosić się do dalszych zastosowań i prowadzić do hamowania reakcji enzymatycznych. Usunięcie takich zanieczyszczeń może sprawić, że metody enzymatyczne, takie jak qPCR, będą bardziej czułe i odporne.
Aby ocenić obecność zanieczyszczeń hamujących procesy enzymatyczne, genomowe DNA z próbek nerek myszy oczyszczono przy użyciu każdej metody oczyszczania spinowego. Próbki końcowe wykorzystano w eksperymentach analizy qPCR (Ryc. 6). Gen odpowiadający beta-aktynie był również amplifikowany jako kontrola endogenna. Dane pokazują, że krzywe amplifikacji dla próbek oczyszczonych krzemionką były przesunięte w prawo w porównaniu do próbek przygotowanych przy użyciu metody chromatografii negatywnej GenElute™-E (wykluczanie wielkości). Sugeruje to obecność zakłócających zanieczyszczeń lub niższą niż obliczona wydajność w próbkach oczyszczonych krzemionką.

Rysunek 6. Analiza qPCR w celu określenia zakłócających zanieczyszczeń w preparatach genomowego DNA. A) Krzywe amplifikacji dla genomowego DNA przygotowanego z tkanki nerki myszy przy użyciu zestawu spin prep na bazie krzemionki od dostawcy X (niebieskie krzywe) i zestawu spin prep GenElute™-E do chromatografii negatywnej (wykluczanie wielkości) (pomarańczowe krzywe). Krzywe dla próbek oczyszczonych krzemionką są przesunięte w prawo w porównaniu do próbek oczyszczonych GenElute™-E, co sugeruje obecność zakłócających zanieczyszczeń lub przeszacowanie stężenia początkowego w preparatach krzemionkowych. B) Obliczenia Cq przy użyciu genu β-aktyny jako endogennego odniesienia kontrolnego.
Wnioski
Zestawy do oczyszczania DNA i RNA z pojedynczym wirowaniem GenElute™-E zapewniają wygodną metodę chromatografii ujemnej do oczyszczania kwasów nukleinowych w oparciu o wykluczenie wielkości. Zestawy te stanowią alternatywę dla tradycyjnych preparatów spinowych opartych na krzemionce, o zwiększonej czystości, o czym świadczy spektrofotometria UV, elektroforeza żelowa i qPCR.
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?