Oczyszczanie oligonukleotydów

Znaczenie oczyszczania oligonukleotydów
Oczyszczanie oligonukleotydów to proces izolowania zsyntetyzowanych oligonukleotydów (krótkich sekwencji DNA lub RNA) od zanieczyszczeń, takich jak niekompletne sekwencje, sole lub organiczne produkty uboczne. Zanieczyszczenia te powstają podczas chemicznej syntezy oligonukleotydów i mogą wpływać na jakość produktu końcowego. Wybór optymalnej techniki oczyszczania oligonukleotydów zależy od rodzaju oligonukleotydu i jego zastosowania. Optymalne oczyszczanie ma znaczące korzyści w zakresie wydajności, w tym:
Poprawa dokładności
Zanieczyszczenia mogą zakłócać dalsze zastosowania, takie jak PCR, sekwencjonowanie lub edycja genów. Oczyszczone oligonukleotydy zapewniają wyższą specyficzność i dokładność wiązania z sekwencjami docelowymi.1
Zwiększona wydajność
Oligonukleotydy o wysokiej czystości poprawiają wydajność wiązania w diagnostyce molekularnej i testach badawczych, takich jak qPCR i hybrydyzacja.2
Zmniejszony szum tła
Usuwanie zanieczyszczeń z produktów ubocznych produkcji oligonukleotydów minimalizuje sygnały tła w eksperymentach molekularnych, prowadząc do bardziej precyzyjnych wyników.3
Spójność w eksperymentach
Oczyszczanie oligo zapewnia powtarzalność w badaniach, co ma kluczowe znaczenie dla generowania wiarygodnych, spójnych wyników w wielu testach.4
Wspólne metody oczyszczania obejmują odsalanie, wkłady, HPLC (wysokowydajna chromatografia cieczowa) i PAGE (elektroforeza w żelu poliakrylamidowym).
Źródła zanieczyszczeń oligo i opcje oczyszczania
Podczas produkcji DNA, każdy nukleotyd jest sekwencyjnie sprzężony z rosnącym łańcuchem poprzez chemii fosforoamidytów. W każdym cyklu sprzęgania niewielki procent łańcuchów oligonukleotydowych nie wydłuża się, co skutkuje mieszaniną sekwencji o pełnej długości (n) i skróconych (n-1, n-2 itd.) ("shortmers"). Ponadto, zanieczyszczenia drobnocząsteczkowe są produktem ubocznym procesu rozszczepiania i deprotekcji.
Po rozszczepieniu, deprotekcji i odsalaniu (usuwa zanieczyszczenia drobnocząsteczkowe), dodatkowe oczyszczanie może oddzielić pożądaną sekwencję pełnej długości od niepożądanych krótkich cząsteczek.
Czystość wymagana dla konkretnej techniki/aplikacji (Tabela 1) zależy od potencjalnych problemów spowodowanych obecnością shortmerów.
Ponadto, oligonukleotydy, które zawierają powszechne modyfikacje, np. biotynę, Amino C2 dT, itp. mogą być oczyszczane przy użyciu dowolnej metody opisanej poniżej. W przypadku oligonukleotydów zawierających bardziej złożone struktury barwników, np. ROX™, TxRd (Sulforhodamine 101-X) itp., HPLC jest metodą z wyboru; tylko ona może usunąć wolny barwnik, który w przeciwnym razie zakłócałby działanie zamierzonej techniki/aplikacji.
Wyjaśnienie metod oczyszczania oligo
Oczyszczanie oligo przez odsalanie
Desalanie usuwa zanieczyszczenia drobnocząsteczkowe, np.akrylonitryl powstający w wyniku deprotekcji szkieletu fosfodiestrowego, które są pozostałościami produktów ubocznych po rozszczepieniu i deprotekcji. W przypadku wielu technik/aplikacji, w tym PCR, odsalanie jest dopuszczalne dla oligonukleotydów ≤35 zasad, ponieważ przytłaczająca obfitość sekwencji o pełnej długości przeważa nad jakimkolwiek wkładem krótkich sekwencji (dowiedz się więcej o obliczaniu wydajności). Oligonukleotydy >35 zasad mogą wymagać dodatkowego oczyszczania, takiego jak Reverse-Phase Cartridge (Cartridge).
Nie ma gwarantowanego poziomu czystości w przypadku oczyszczania wyłącznie za pomocą soli, ponieważ proces ten nie usuwa krótkich cząsteczek. Każdy oligonukleotyd jest odsalany bez dodatkowej opłaty.
Oczyszczanie oligonukleotydów za pomocą kartridża
Separacja na kartridżu z odwróconą fazą oferuje kolejny poziom czystości (Rysunek 1). Podstawą separacji jest różnica w hydrofobowości między sekwencją o pełnej długości (posiada hydrofobową grupę 5'-DMT) a krótkimi sekwencjami (nie posiadają grupy 5'-DMT). Podczas gdy sekwencja o pełnej długości jest zatrzymywana na kolumnie, krótsze fragmenty są wymywane. Po rozszczepieniu 5'-DMT na kasecie, oczekiwana sekwencja pełnej długości jest eluowana i odzyskiwana. Ponadto, oligonukleotydy zmodyfikowane pewnymi barwnikami na końcu 5'-, np. cyjaniną lub WellRED, są kompatybilne z oczyszczaniem za pomocą kasety ze względu na zwiększoną hydrofobowość nadaną przez cząsteczkę barwnika.
Wraz ze wzrostem długości oligonukleotydu, odsetek shortmerów zawierających grupę 5'-DMT (produkt wygenerowany przez sekwencje nieosłonięte) ma tendencję do wzrostu. Te niepożądane sekwencje nie zostaną usunięte przez oczyszczanie za pomocą kasety, dlatego w przypadku dłuższych oligonukleotydów zaleca się wysokosprawną chromatografię cieczową (HPLC) lub elektroforezę w żelu poliakrylamidowym (PAGE). Oczyszczanie za pomocą wkładów nie zapewnia gwarantowanego poziomu czystości. Jednak wieloletnie doświadczenie wykazało, że 65-80% pełnej długości sekwencji (za pomocą analitycznej HPLC) jest powszechne.
Oczyszczanie oligo za pomocą RP-HPLC
Wysokosprawna chromatografia cieczowa w odwróconej fazie (RP-HPLC) działa na tej samej zasadzie, co kartridż w odwróconej fazie (Rysunek 1). Jednak wyższa rozdzielczość pozwala na uzyskanie wyższych poziomów czystości. RP-HPLC jest skuteczną metodą oczyszczania oligonukleotydów z barwnikami, ponieważ ich wewnętrzna hydrofobowość zapewnia doskonałe oddzielenie pożądanej sekwencji od tych, w których brakuje barwnika, krótkich i krótkich z delecjami. Co więcej, RP-HPLC jest metodą z wyboru do syntezy na dużą skalę ze względu na pojemność i właściwości rozdzielcze kolumny. Jednak rozdzielczość oparta na hydrofobowości zmniejsza się wraz z długością oligonukleotydu. Dlatego RP-HPLC zazwyczaj nie jest zalecana do oczyszczania oligonukleotydów o długości 50 zasad. Chociaż dłuższe oligonukleotydy (do 80 zasad, w niektórych przypadkach dłuższe) mogą być oczyszczane przy użyciu tej metody, może to mieć negatywny wpływ na czystość i wydajność.
Standardowa RP-HPLC zazwyczaj osiąga czystość > 85% sekwencji pełnej długości (poprzez analityczną HPLC). Wyższe poziomy czystości mogą być osiągalne w zależności od składu sekwencji. Dokładny charakter tego, co RP-HPLC zapewnia pod względem czystości, a także wszelkie związane z tym opłaty, należy sprawdzić u lokalnego sprzedawcy lub specjalisty ds. obsługi klienta.

Rysunek 1. Separacja przez fazę odwróconą: wkład i HPLC. Zarówno standardowe oczyszczanie za pomocą kartridża, jak i RP-HPLC odbywa się z 5'-DMT na sekwencji.
.
Oczyszczanie oligonukleotydów metodą IE-HPLC
Wysokosprawna chromatografia cieczowa z wymianą anionów (IE-HPLC) opiera się na liczbie naładowanych grup (fosforanowych) w szkielecie oligonukleotydu. Metoda anionowymienna polega na zastosowaniu fazy ruchomej z gradientem soli na czwartorzędowej amonowej fazie stacjonarnej (Rysunek 2). Rozdzielczość jest doskonała do oczyszczania mniejszych ilości. Jednak IE-HPLC jest ograniczona długością, zwykle do 40 zasad. Dłuższe oligonukleotydy prowadzą do niższej rozdzielczości między sekwencją o pełnej długości a krótszymi; a zatem niższej i mniej spójnej czystości.
Podstawowym powodem stosowania IE-HPLC w przeciwieństwie do RP-HPLC jest oczyszczanie oligonukleotydów o znaczącej strukturze drugorzędowej, zwykle występujących w sekwencjach o wysokiej zawartości GC. IE-HPLC jest skuteczna w przypadku takich oligonukleotydów, ponieważ faza ruchoma ma wysoce zasadowe pH, które zakłóca wiązania wodorowe, a tym samym strukturę drugorzędową. Po tej technice można zastosować RP-HPLC, aby dodać drugi wymiar do procesu separacji. Prosimy o zapytanie o gwarantowany procent sekwencji o pełnej długości.

Rysunek 2.Separacja za pomocą IE-HPLC. Oczyszczanie IE-HPLC odbywa się bez 5'-DMT na sekwencji.
.
Oczyszczanie oligonukleotydów metodą PAGE
Elektroforeza w żelu poliakrylamidowym (PAGE) wykorzystuje denaturujące środowisko do rozdzielania oligonukleotydów na podstawie masy cząsteczkowej, a nie ładunku (Rys. 3). W odpowiednich warunkach można rozdzielić oligonukleotydy różniące się pojedynczą zasadą. Ta doskonała rozdzielczość wielkości zazwyczaj skutkuje najwyższym osiągalnym poziomem czystości spośród wszystkich dostępnych metod oczyszczania. Minimalna wydajność PAGE jest niższa niż w przypadku innych metod ze względu na złożoną procedurę wymaganą do ekstrakcji oligonukleotydów. Technika ta jest zalecana, gdy wymagana jest wysoka czystość i dla sekwencji ≥50 zasad. PAGE zazwyczaj osiąga czystość na poziomie 95% pełnej długości sekwencji. Dokładny charakter tego, co PAGE zapewnia pod względem czystości, a także wszelkie związane z tym opłaty, należy sprawdzić u lokalnego sprzedawcy lub specjalisty ds. obsługi klienta.
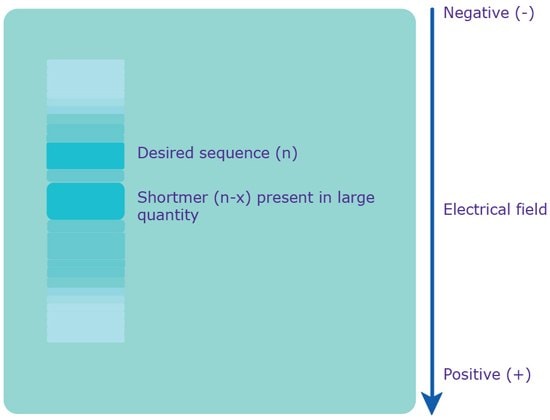
Rysunek 3.Separacja przez PAGE. Do żelu przykładane jest pole elektryczne, które powoduje, że polianionowy oligonukleotyd przemieszcza się do dodatnio naładowanej anody. Ponieważ ładunek jest równomiernie rozłożony w całej surowej mieszaninie, różne sekwencje są rozdzielane na podstawie masy cząsteczkowej, tj. długości. Krótsze sekwencje poruszają się szybciej, podczas gdy dłuższe, pożądane sekwencje poruszają się wolniej.
Oczyszczanie oligonukleotydów przez filtrację żelową
Filtracja żelowa jest zalecana w przypadku oligonukleotydów o mniejszej skali, przeznaczonych do eksperymentów in vivo (Rysunek 4). W zależności od miejsca produkcji, oligonukleotydy fosforotioanowe (S-Oligos), które są często używane do badań antysensownych in vivo, mogą być poddawane filtracji żelowej (prosimy o kontakt z lokalnym sprzedawcą lub specjalistą ds. obsługi klienta, aby sprawdzić, czy ta obróbka jest dostępna w danym regionie geograficznym). Oczyszczanie to usuwa śladowe ilości produktów ubocznych syntezy, rozszczepiania i deprotekcji (które nie zostały usunięte przez standardowe odsalanie), a także rozpuszczalniki oczyszczające. Te śladowe produkty uboczne, które są zazwyczaj nieszkodliwe in vitro, mogą prowadzić do efektów cytotoksycznych in vivo.
Dodatkowe opcje in vivo są dostępne z naszym produktem o większej ilości, iScale Oligos™.

Rysunek 4. Separacja poprzez filtrację żelową. Oczyszczanie przez filtrację żelową odbywa się bez 5'-DMT na sekwencji.
Wyzwania związane z oczyszczaniem oligonukleotydów do sekwencjonowania nowej generacji
Powyższe tradycyjne metody oczyszczania są skuteczne w usuwaniu zanieczyszczeń drobnocząsteczkowych, a także zwiększają procent sekwencji o pełnej długości do pożądanego poziomu. Dodatkowy rodzaj zanieczyszczeń, zanieczyszczenia krzyżowe, pojawił się jako problem ze względu na ciągły wzrost wykorzystania niezwykle czułych technik, takich jak sekwencjonowanie nowej generacji (NGS).5 Tradycyjne metody oczyszczania nie są skuteczne w usuwaniu oligonukleotydów zanieczyszczających krzyżowo. Niewielkie ilości zanieczyszczeń krzyżowych są rzeczywistością w każdym wysokowydajnym zakładzie produkcji oligonukleotydów. Zazwyczaj takie niewielkie ilości nie mają zauważalnego wpływu na tradycyjne techniki/aplikacje, takie jak PCR. Jednak większość platform NGS opiera się na adapterach indeksowych (kodów kreskowych) do multipleksowania. Jeśli niewielkie ilości niewłaściwego kodu kreskowego (z powodu zanieczyszczenia krzyżowego) zostaną dodane do niewłaściwego celu sekwencjonowania, problem jest niewykrywalny aż do etapu analizy danych, co sprawia, że problem ten jest kosztowny pod względem czasu i pieniędzy. Next-Gen Sequencing Oligos (NGSO) zostały opracowane w celu uniknięcia tego problemu. NGSO jest produkowany przy użyciu zastrzeżonych metod oczyszczania i jest zalecany dla każdego oligonukleotydu przeznaczonego do użycia jako adapter NGS.
Zalecenia dotyczące metod oczyszczania oligonukleotydów
Oligonukleotydy najlepiej oczyszczać różnymi metodami lub kombinacją metod w zależności od zamierzonego zastosowania. Aby rozpocząć, zapoznaj się z tabelą 1 dla zalecanych metod opartych na technice/zastosowaniu. Ponadto, aby zoptymalizować metodę oczyszczania oligo, należy skonsultować się z naszym działem usług technicznych pod adresem oligotechserv@sial.com. Mamy ponad 35-letnie doświadczenie w projektowaniu, produkcji, modyfikacji i oczyszczaniu oligos DNA i RNA.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?