Podstawy ilościowego PCR
A Technical Guide to PCR Technologies
Wykrywanie ilościowe PCR (formalnie ilościowe PCR w czasie rzeczywistym, qPCR) opiera się na podstawowej technice PCR i pozwala badaczom oszacować ilość materiału wyjściowego w próbce. Ponieważ produkty są wykrywane w miarę postępu reakcji, qPCR ma znacznie szerszy zakres dynamiczny analizy niż konwencjonalny, końcowy PCR; od pojedynczej kopii do około 1011 kopii można wykryć w jednym przebiegu. Ilościowa reakcja PCR w czasie rzeczywistym i późniejsze wykrywanie amplikonów jest przeprowadzane w formacie zamkniętej probówki, co eliminuje potrzebę manipulacji po PCR, takich jak elektroforeza żelowa i znacznie zmniejsza ryzyko zanieczyszczenia krzyżowego.
Podstawowe zasady qPCR zostały omówione na tej stronie, a mechanizmy powszechnie stosowanych chemikaliów detekcyjnych opisano w Quantitative PCR and Digital PCR Detection Methods.
Podstawowe zasady.
Podczas wykonywania qPCR, fluorescencyjny barwnik reporterowy jest używany jako pośredni pomiar ilości kwasu nukleinowego obecnego podczas każdego cyklu amplifikacji. Wzrost sygnału fluorescencyjnego jest wprost proporcjonalny do ilości wykładniczo gromadzących się cząsteczek produktu PCR (amplikonów) wytwarzanych podczas powtarzających się faz reakcji (patrz reakcja łańcuchowa polimerazy). Cząsteczki reporterowe są klasyfikowane jako; barwniki wiążące dwuniciowe DNA (dsDNA), barwniki sprzężone ze starterami lub dodatkowe oligonukleotydy sprzężone z barwnikami, określane jako sondy (Quantitative PCR and Digital PCR Detection Methods).
Zastosowanie barwnika wiążącego dsDNA, takiego jak SYBR® Green I, stanowi najprostszą formę chemii detekcyjnej. W stanie wolnym w roztworze lub przy obecności tylko jednoniciowego DNA (ssDNA), barwnik SYBR Green I emituje światło o niskiej intensywności sygnału. W miarę postępu PCR i wzrostu ilości dsDNA, więcej barwnika wiąże się z amplikonami, a tym samym intensywność sygnału wzrasta.
Alternatywnie, sonda (lub kombinacja dwóch w zależności od chemii wykrywania) może dodać poziom specyficzności wykrywania poza barwnikiem wiążącym dsDNA, ponieważ wiąże się z określonym regionem matrycy, który znajduje się między starterami. Najczęściej stosowanym formatem sondy jest sonda podwójnie znakowana (DLP; określana również jako sonda hydrolizy lub sonda TaqMan® Probe). DLP jest oligonukleotydem z etykietą fluorescencyjną 5', np. 6-FAM™ i cząsteczką wygaszającą 3', taką jak jeden z ciemnych wygaszaczy, np, BHQ®1 lub OQ™ (Quantitative PCR and Digital PCR Detection Methods). Sondy te są zaprojektowane do hybrydyzacji z matrycą między dwoma starterami i są używane w połączeniu z polimerazą DNA, która ma nieodłączną aktywność egzonukleazy od 5' do 3'. Gdy DLP jest wolna w roztworze, intensywność sygnału jest niska, ponieważ barwnik reporterowy znajduje się w pobliżu ugrupowania wygaszającego. W miarę wytwarzania większej ilości matrycy podczas reakcji, więcej sond hybrydyzuje z matrycą, która z kolei jest rozszczepiana przez aktywność egzonukleazy 5' do 3' postępującej polimerazy DNA. Intensywność sygnału fluorescencyjnego wzrasta wraz z uwalnianiem 5' barwnika reporterowego do roztworu. Zastosowanie sond znakowanych różnymi barwnikami reporterowymi pozwala na jednoczesne wykrywanie i kwantyfikację wielu celów w pojedynczej (multipleksowej) reakcji.
Typowy przebieg qPCR składa się z powtarzających się cykli inkubacji w zmiennej temperaturze, Procedura cykliczna 3.1. Ten profil jest często używany, gdy barwniki wiążące dsDNA, Molecular Beacons lub Scorpion® Probes są wybranymi chemikaliami wykrywającymi dla qPCR. Wydłużanie primerów jest najbardziej wydajne w temperaturze 72 °C, ponieważ jest to optymalna temperatura dla procesywności większości polimeraz DNA. W temperaturze 72 °C polimeryzacja zachodzi z szybkością około 100 zasad na sekundę. Amplikony qPCR są zazwyczaj krótsze (<200 zasad) niż konwencjonalne produkty PCR, dlatego wydłużanie jest często łączone z wyżarzaniem w jednym kroku w temperaturze 60 °C podczas pracy z podwójnie znakowanymi amplikonami (Procedura cykliczna 3.2).
Cycling Procedure 3.1
Przykład standardowego profilu qPCR z użyciem barwnika wiążącego dsDNA, Molecular Beacon lub Scorpions® Wykrywanie sondy.
- Denaturacja początkowa: Temperatura reakcji jest zwiększana do 95 °C, a próbka jest inkubowana przez 2-10 min (czas zależy od mechanizmu gorącego startu enzymu polimerazy), aby zapewnić, że wszystkie złożone cele (dsDNA) są oddzielone i są jednoniciowe i dostępne do amplifikacji
- Cykling:
- Denaturacja: Temperatura reakcji jest zwiększana do 95 °C na 10 sekund w celu stopienia całego dsDNA.
- Annealing: Temperatura jest obniżana do 60 °C przez 30 s w celu promowania wiązania starterów i sondy (jeśli jest dołączona) z matrycą.
- Wydłużanie: Kolejne wydłużanie zachodzi w podwyższonej temperaturze 72 °C, która jest optymalna dla procesywności polimerazy TaqDNA. Czas trwania wydłużania zależy od wielkości amplikonu (30 sekund na 1 kb). Okres wydłużania zależy od pożądanej długości amplikonu i użytego enzymu. Ponieważ amplikony qPCR są krótkie, jest to zwykle 5-30 s.
- Denaturacja: Temperatura reakcji jest zwiększana do 95 °C na 10 sekund w celu stopienia całego dsDNA.
- Powtórzenie: Kroki 1-3 są powtarzane, zwykle przez 40 cykli.
Cycling Procedure 3.2
Przykładowy dwuetapowy profil qPCR do wykrywania DLP.
- Denaturacja początkowa: Temperatura jest zwiększana do 95 °C i reakcja jest inkubowana przez 2-10 min (w zależności od właściwości gorącego startu enzymu polimerazy)
- Cykling:
- Denaturacja: Temperatura reakcji jest zwiększana do 95 °C na 10 sekund w celu stopienia całego dsDNA.
- Annealing i wydłużanie: Temperatura jest obniżana do 60 °C przez 30 s w celu promowania wiązania starterów z matrycą, a następnie następuje wydłużanie ze względu na wystarczającą aktywność polimerazy DNA w tej temperaturze.
- Powtórzenie: Kroki 1-2 są powtarzane, zwykle przez 40 cykli.
Zmiana fluorescencji w trakcie reakcji jest mierzona przez termocykler PCR w czasie rzeczywistym. Dedykowane instrumenty do PCR w czasie rzeczywistym łączą cykle temperaturowe z jednostką optyczną. Jednostka optyczna dostarcza światło o odpowiedniej długości fali w celu wzbudzenia fluoroforu i wykrywa wynikową emisję. Wiele nowoczesnych przyrządów ma przymocowany ekran lub pobliski monitor komputerowy, który umożliwia śledzenie przebiegu reakcji (Rysunek 3.1).
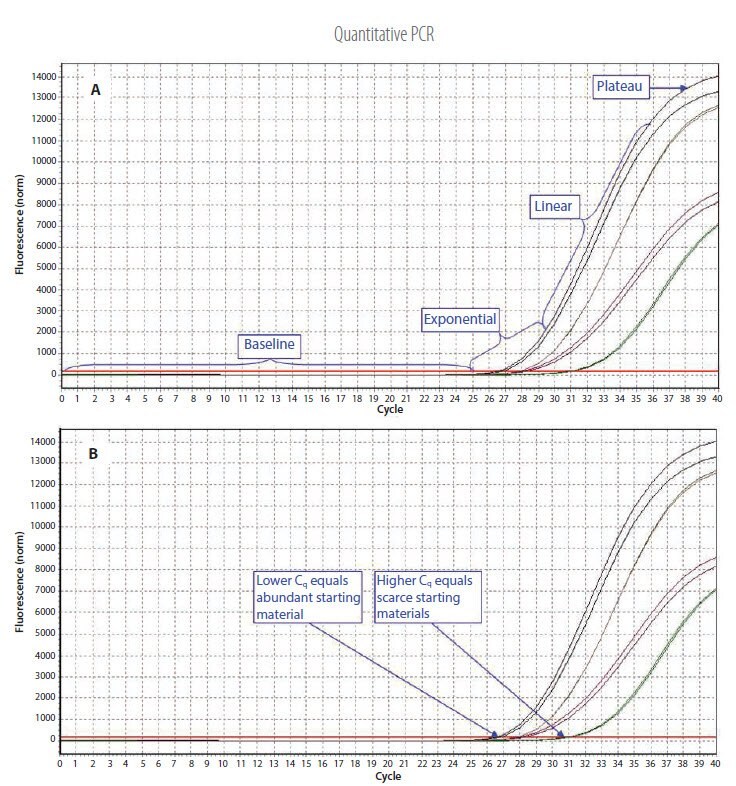
Rysunek 3.1.Przykładowe dane testu qPCR. A) Różne fazy reakcji. Linia bazowa: Początkowe stężenie matrycy jest niskie; dlatego intensywność fluorescencji jest zbyt niska, aby można ją było wykryć i widoczny jest tylko sygnał tła. Wykładniczy: Po osiągnięciu przez docelową wydajność progu detekcji, pokazanego jako czerwona linia progowa, przebieg reakcji można śledzić w fazie wykładniczej. Liniowa: Wraz ze wzrostem stężenia matrycy zmniejsza się dostępne stężenie polimerazy DNA i spada szybkość reakcji. Plateau: Reakcja osiąga maksymalną wydajność. B) Poszczególne reakcje charakteryzują się cyklem, w którym fluorescencja po raz pierwszy wzrasta powyżej progu, co jest określane jako cykl kwantyfikacji (Cq). Jeśli materiał wyjściowy jest obfity, amplifikację obserwuje się we wcześniejszych cyklach, a Cq jest niższe. Jeśli materiału wyjściowego jest mało, amplifikację obserwuje się w późniejszych cyklach, a Cq jest wyższe. Ta korelacja między fluorescencją, Cq i ilością amplifikowanego produktu umożliwia kwantyfikację matrycy w szerokim zakresie dynamicznym.
Kwantyfikacja i analiza celów gDNA
Quantitative real-time PCR może być łatwo zastosowany do analizy celów gDNA. Badania takie mogą obejmować określanie genotypów/SNP
, analizę metylacji, badanie sekwencji transgenicznych lub monitorowanie insercji i delecji.
.Kwantyfikacja i analiza transkryptów mRNA
Powszechnym zastosowaniem qPCR jest analiza ekspresji genów, np.g., porównanie stężeń mRNA genu będącego przedmiotem zainteresowania między próbkami kontrolnymi i poddanymi działaniu. MRNA można określić ilościowo za pomocą dwuetapowego lub jednoetapowego procesu RT-qPCR (patrz Reverse Transcription Quantitative Real-time PCR, aby uzyskać więcej szczegółów na temat reakcji RT).
Two-step RT-qPCR
Reakcje odwrotnej transkrypcji i qPCR są przeprowadzane sekwencyjnie w oddzielnych probówkach. Całkowity RNA lub, rzadziej, poli(A)+ RNA jest używany jako materiał wyjściowy, a cDNA jest wytwarzany przez wydłużanie ze starterów oligo-dT, starterów losowych, mieszanki starterów oligo-dT / starterów losowych lub starterów specyficznych dla genu przy użyciu enzymu odwrotnej transkryptazy. Podwielokrotność tej reakcji jest następnie dodawana do qPCR.
Jednoetapowy RT-qPCR
.Gdy reakcje odwrotnej transkrypcji i qPCR są połączone w jednej probówce, konfiguracja eksperymentalna jest uproszczona
i istnieje mniejsze prawdopodobieństwo zanieczyszczenia, ponieważ nie ma wymogu dalszego obchodzenia się z próbką.
Kwantyfikacja i analiza transkryptów ncRNA.
Większość niekodujących RNA jest dłuższa niż 100 nukleotydów i dlatego może być wykrywana i oznaczana ilościowo za pomocą RT-qPCR
przy użyciu tych samych technik, które są stosowane do analizy mRNA. Natomiast krótkie niekodujące RNA, takie jak mikro i piwi RNA, mają zasadniczo długość pojedynczego startera PCR. W związku z tym przed wykonaniem qPCR potrzebna jest technika, która wydłuża te krótkie RNA. W dostępnych na rynku systemach stosowane są dwa główne podejścia: 1) użycie startera RT z pętlą macierzystą i 2) ogonowanie poli-A, a następnie RT ze starterem adapterowym oligo-dT (Rysunek 3.2). Alternatywne podejście opracowane przez Casoldi i in, które nie jest dostępne w handlu, wykorzystuje ligację cząsteczki adaptera do całego RNA w próbce i specyficzny projekt primera w celu rozróżnienia pożądanych celów1,2.
Podejście primera RT z pętlą macierzystą zostało opracowane i skomercjalizowane przez Applied Biosystems (obecnie Life Technologies/Thermo) do późniejszego qPCR opartego na sondzie TaqMan3. Jak pokazano na Rysunku 3.2A, 5'-koniec startera RT może być sparowany zasadą z regionem kilku nukleotydów od jego własnego 3'-końca, tworząc sparowany zasadą trzon oddzielony niesparowaną pętlą. Wszystkie, z wyjątkiem kilku ostatnich nukleotydów na 3'-końcu startera RT są uniwersalne, to znaczy zawierają tę samą sekwencję we wszystkich starterach miRNA. Ostatnie kilka nukleotydów, które rozciągają się 3' od trzonu, są komplementarne do 3'-końca docelowego miRNA. Przedłużenie startera wzdłuż matrycy miRNA tworzy
cDNA, które można amplifikować za pomocą startera do przodu specyficznego dla miRNA i uniwersalnego startera odwrotnego, z których ten ostatni jest
komplementarny do 5'-końca startera RT z pętlą macierzystą.
Wszystkie inne komercyjne metody qPCR miRNA, w tym nasza marka MystiCq® wykorzystują polimerazę poly-A do wydłużenia miRNA, jak opisali Shi i Chiang4. Polimeraza poli(A) (PAP), enzym niezależny od szablonu, katalizuje przeniesienie reszt adenozyny z ATP na 3'-koniec dowolnego RNA. Jak pokazano na Rysunku 3.2B, RT można następnie przeprowadzić przy użyciu startera oligo-dT. Starter oligo-dT zawiera sekwencję adaptera na swoim 5'-końcu, co umożliwia późniejsze qPCR ze starterem do przodu, który jest komplementarny do określonego miRNA i starterem odwrotnym, który jest komplementarny do sekwencji adaptera.
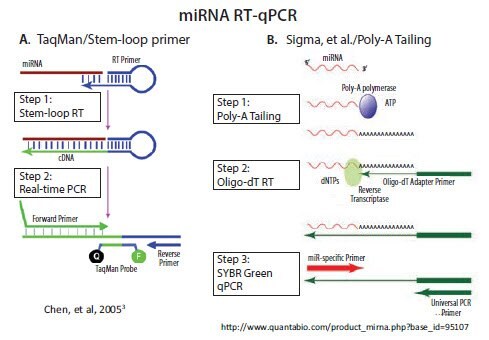
Rysunek 3.2.Istnieją dwie komercyjnie dostępne metody RT i qPCR miRNA. A) Zapętlony primer jest używany do rozpoczęcia etapu RT po hybrydyzacji z 3' końcem miRNA. Przedłużenie PCR przez pętlę skutkuje połączeniem miRNA i uniwersalnej sekwencji, która jest wystarczająco długa do amplifikacji B) Dodanie ogona poli A do miRNA zapewnia miejsce primingu dla primera, który składa się z oligo-dT i uniwersalnej sekwencji primingu. Badania nad kwasami nukleinowymi prowadzone przez Oxford University Press. Powielono za zgodą Oxford University Press w formacie do ponownego wykorzystania w książce/e-booku za pośrednictwem Copyright Clearance Center.
Oba komercyjne podejścia mają zalety i wady. Priming z pętlą macierzystą umożliwia 2-etapową RT-qPCR, podczas gdy większość metod poli-A wymaga dodatkowego etapu RT przed qPCR. Istnieją produkty, które łączą poly-A tailing i RT w jednej reakcji dla 2-etapowego PAP tailing/RT-qPCR. Jednak wykrywanie może być mniej czułe przy takim podejściu (niepublikowane wyniki wewnętrzne). I odwrotnie, poli-A tailing, po którym następuje RT, wytwarza stabilną pulę cDNA, która może być wykorzystana do wykrywania dowolnego mikroRNA, mRNA lub innego RNA, natychmiast lub w dowolnym momencie w przyszłości. W podejściu stem-loop/TaqMan do wykrywania każdego miRNA potrzebny jest inny primer. Do tej samej reakcji RT można dodać wiele starterów z pętlą macierzystą5 a pule starterów RT dla wielu miRNA można zakupić od Life Technologies w celu przeprowadzenia multipleksowej RT. Żadna z tych opcji nie pozwala jednak na wykrywanie za pomocą qPCR miRNA odkrytych w późniejszym czasie, ani nie pozwala na wykrywanie za pomocą qPCR mRNA z tego samego cDNA. System opisany przez Castoldi i wsp1,2 jest wyjątkowy, ponieważ zapewnia środki do wykrywania mRNA, prekursora i w pełni przetworzonych cząsteczek miRNA z tej samej próbki całkowitego RNA.
Wymagania qPCR
Instrumenty
Wiele instrumentów qPCR zostało zaprojektowanych do obsługi określonego zakresu aplikacji, np.g., Porównaj możliwości wysokowydajnego aparatu ABi 7900 wykorzystującego automatyczne ładowanie płytek 384-dołkowych z produkowanym i sprzedawanym przez firmę Illumina aparatem Eco, który obsługuje pojedynczą płytkę 48-dołkową. Dlatego najbardziej odpowiedni instrument spełnia potrzeby badań. Pożądane jest wybranie urządzenia z przyjaznym dla użytkownika oprogramowaniem, które wykonuje najbardziej pożądane funkcje i jest elastyczne pod względem danych wyjściowych, dzięki czemu można nimi łatwo manipulować w dalszych pakietach oprogramowania do analizy statystycznej. Skraca to czas wymagany do przeszkolenia personelu, a tym samym do rozpoczęcia generowania wyników. Dodatkowe wymagane funkcje obejmują blok PCR, który jest absolutnie jednolity (bezwzględne maksymalne odchylenie 1 Cq = 2-krotne w 96 studzienkach replikacji) oraz system optyczny, który wzbudza i wykrywa emisję tak czule i równomiernie, jak to możliwe w szerokim zakresie długości fal. Pozwala to na szeroki wybór fluoroforów i umożliwia multipleksowanie. Inne cechy, które należy wziąć pod uwagę, to koszty operacyjne związane z określonymi materiałami eksploatacyjnymi, np, jeśli standardowa płytka mikrotitracyjna nie jest używana do reakcji, a także wygoda ładowania płytek/probówek o niestandardowym formacie.
Template
Do zainicjowania qPCR wymagana jest bardzo mała liczba kopii docelowego kwasu nukleinowego (równoważna około 100 pg gDNA lub cDNA). Aby zminimalizować zanieczyszczenie inhibitorami reakcji, początkowa ilość matrycy powinna być ograniczona do minimum wymaganego do uzyskania dokładnej kwantyfikacji. Gdy materiałem wyjściowym jest RNA, projektowanie starterów i traktowanie DNazą I zmniejszy sygnały, które mogą być generowane przez zanieczyszczenie gDNA.
Prymery
Niezależnie od tego, czy używany jest barwnik wiążący dsDNA, czy chemia detekcji oparta na sondach, projektowanie wysokiej jakości primerów jest jednym z najbardziej krytycznych etapów przedeksperymentalnych w qPCR. Szczegółowe omówienie projektowania testów można znaleźć w PCR, qPCR, dPCR Assay Design. Aby zmaksymalizować czułość i specyficzność, należy uwzględnić wszelkie wymagane modyfikacje, takie jak zablokowany kwas nukleinowy (qPCR Detection Chemistry).
Sonda(y)
Przy użyciu sond jako mechanizmu wykrywania amplikonów, dimery starterów i niespecyficzne produkty nie są wykrywane, ale należy ich unikać, ponieważ mogą obniżyć wydajność reakcji. Aby zmaksymalizować czułość i specyficzność, należy wybrać odpowiedni typ sondy dla danego zastosowania i uwzględnić wszelkie wymagane modyfikacje, takie jak zablokowany kwas nukleinowy (qPCR Detection Chemistry).
dNTPs
Standardowe mieszanki wzorcowe PCR i qPCR zawierają dATP, dCTP, dGTP i dTTP. Dostępne są jednak mieszanki, które zastępują dTTP dUTP. Produkty z poprzednich reakcji przeprowadzonych z dUTP będą zawierać uracyl zamiast tyminy. Są one następnie podatne na rozszczepienie przez glikozylazę Uracil-DNA (UNG). Dlatego wcześniejsza inkubacja kolejnych reakcji z UNG zapobiega przenoszeniu zanieczyszczeń między reakcjami. Aby być skutecznym, wszystkie reakcje PCR w laboratorium muszą wykorzystywać dUTP.
Magnez
Chlorek magnezu (MgCl2) jest niezbędny dla aktywności odwrotnej transkryptazy, polimerazy Taq DNA i egzonukleazy Taq DNA 5' do 3'. Optymalne stężenie Mg2+ dla reakcji zawierających DLP wynosi zwykle 3-6 mM. Większość mieszanin wzorcowych zawiera MgCl2, jednak czasami konieczne jest zoptymalizowanie stężenia, więc dodatkowa probówka czystego MgCl2 jest zwykle dołączana do produktu master mix (patrz Assay Optimization and Validation). W niektórych przypadkach może być wymagana mieszanina reakcyjna, która nie zawiera MgCl2 aby można było zastosować niskie stężenie, np, podczas korzystania z wykrywania sondy Scorpions® Probe.
Odwrotna transkryptaza
Enzym odwrotnej transkryptazy, który zapewnia wysoką wydajność cDNA, zachowując aktywność w wysokiej temperaturze, ma kluczowe znaczenie dla powodzenia RT-qPCR. Wydajność w wysokich temperaturach pomaga zapewnić, że regiony RNA o znaczącej strukturze drugorzędowej są destabilizowane i dostępne do hybrydyzacji i późniejszej amplifikacji. Podczas wykonywania jednoetapowego RT-qPCR, wysoka temperatura pozwala na stosowanie starterów specyficznych dla genów o wysokich temperaturach topnienia (Tm), co zwiększa specyficzność reakcji. Podczas wykonywania protokołów dwuetapowych ważne jest, aby zapewnić, że enzym daje liniową i proporcjonalną wydajność cDNA z RNA (patrz Reverse Transcription dla szczegółów oceny RT).
Polimeraza Taq DNA
Podobnie jak w przypadku wyboru najbardziej odpowiedniej odwrotnej transkryptazy do RT, wybór odpowiedniego enzymu polimerazy jest kluczowy. Podstawowym problemem związanym z naturalną polimerazą Taq DNA jest to, że enzym ten wykazuje szczątkową aktywność w niskiej temperaturze. Niespecyficzne wiązanie starterów prowadzi do niespecyficznego tworzenia produktu w wyniku tej resztkowej aktywności polimerazy. Zablokowane przeciwciałami lub chemicznie polimerazy Taq DNA ("gorący start") pomagają naprawić tę sytuację, zapobiegając aktywności enzymu do momentu rozpoczęcia etapu denaturacji w wysokiej temperaturze.
Bufor
Bufory lub wzorcowe mieszaniny reakcyjne zazwyczaj zawierają dNTP, polimerazę Taq DNA, MgCl2 i stabilizatory. Barwnik SYBR Green I, ROX™, fluoresceina i obojętne barwniki ładujące mogą być również zawarte (barwniki kontrolne ładowania), w zależności od chemii wykrywania, instrumentu i wymagań reakcji. Składniki buforu PCR i stabilizatory są zazwyczaj zastrzeżone przez producenta. W przypadku zakupu oddzielnie, możliwa jest maksymalna elastyczność, ponieważ każdy składnik może być optymalizowany indywidualnie w reakcji. Jednak w przeciwieństwie do tego, podczas gdy zakup składników razem jako mieszaniny wzorcowej zmniejsza elastyczność, zwiększa spójność partii i wygodę, jednocześnie zmniejszając liczbę etapów pipetowania, a tym samym ryzyko błędu i zanieczyszczenia.
Ładowanie barwników kontrolnych
Niektóre termocyklery PCR w czasie rzeczywistym wymagają dołączenia do każdej reakcji barwnika ładującego, takiego jak ROX, w celu kontroli zmienności systemu optycznego i normalizacji różnic w intensywności sygnału. Podobnie, niektóre termocyklery wymagają początkowego sygnału fluoresceiny, aby stworzyć wirtualne tło podczas pracy z testami barwnika SYBR Green I (które mają bardzo niskie tło). Mogą one być dostarczane w mieszaninie wzorcowej lub jako oddzielne składniki, aby można było użyć odpowiedniego stężenia.
Wytyczne MIQE.
Jednym z wyraźnych problemów związanych z wykorzystaniem qPCR jest to, że bardzo łatwo jest uzyskać liczby, które następnie można łatwo zinterpretować jako wyniki ilościowe. Może to być jednak równie mylące, ponieważ kontrola jakości i dalsza analiza mogą być wymagane w celu rozróżnienia potencjalnych artefaktów spowodowanych niewłaściwym projektem testu, wykonaniem lub analizą danych. Publikacja pozornie istotnych danych bez odpowiedniej kontroli jakości doprowadziła do powstania raportów, które w najlepszym przypadku nie mają znaczenia biologicznego lub klinicznego, a w najgorszym są niedokładne.
Każdy etap procesu eksperymentalnego qPCR, od pozyskania próbki do analizy danych qPCR, jest podatny na zmienność6. Dlatego zmienność może wystąpić podczas wyboru i przetwarzania próbek, ekstrakcji kwasów nukleinowych (może powodować różnice w jakości, które powodują brak powtarzalności wykrywania celu qPCR), projektowania i optymalizacji testu (przyczynia się do różnic w wydajności i czułości testu) oraz analizy danych (wymaga pewnej subiektywnej interpretacji i zastosowania odpowiednich metod statystycznych). Niezwykle ważne jest, aby informacje dostarczane odbiorcom naukowym zawierały odpowiednie informacje umożliwiające krytyczną ocenę eksperymentu7, jednak oczywiste jest, że nie zawsze tak jest8. Wytyczne "Minimum Information for Publication of Quantitative Real-time PCR Experiments" (MIQE)9 wychodzą naprzeciw tym obawom. Celem wytycznych MIQE jest zwiększenie przejrzystości poprzez określenie minimalnych informacji o teście, które są wymagane, aby czytelnik mógł ocenić techniczną poprawność publikacji oraz dostarczenie wskazówek dotyczących projektowania, optymalizacji i walidacji nowych testów qPCR.
MIQE składa się z dziewięciu sekcji (projekt eksperymentu, próbka, kwasy nukleinowe, odwrotna transkrypcja, cel, startery i sondy, szczegóły testu, cykl PCR i analiza danych; odwiedź naszą stronę MIQE aby pobrać listę kontrolną, Tabela 1). Wszystkie parametry odnoszą się do informacji, które należy uzyskać podczas procesu projektowania eksperymentalnego, optymalizacji i walidacji. W przypadku korzystania z testów komercyjnych lub tych z opublikowanej literatury, optymalizacja i walidacja powinny być nadal wykonywane. Dokument MIQE zawiera listę kontrolną z czynnikami oznaczonymi jako Niezbędne, które są uważane za niezbędne do odpowiedniego opisu testu qPCR, podczas gdy inne elementy są oznaczone jako Pożądane, wskazując, że te informacje są przydatne, ale nawet bez nich test może zostać powtórzony, a dane w publikacji ocenione. Ważne jest, aby zdawać sobie sprawę, że raportowanie wydajności PCR, czułości analitycznej i specyficzności każdego indywidualnego testu jest niezbędne i powinno być określone przez badacza w warunkach laboratoryjnych. Istnieją szczególne względy podczas wykonywania cyfrowej PCR (patrz Digital PCR), które zostały omówione w niedawnej publikacji cyfrowej PCR MIQE10.
Czy jesteś zgodny z MIQE?
Minimum Information for Publication of Quantitative Real-time PCR Experiments
aClinical chemical Copyright 2009 by American Association For Clinical Chemistry, Inc. Reproduced with permission of American Association For Clinical Chemistry, Inc. in the format Internet posting via Copyright Clearance center.
bAll essential information must be submitted with the manuscript, Desirable information should be submitted if available. Jeśli startery pochodzą z RTPrimerDB, informacje na temat celu qPCR, oligonukleotydów, protokołów i walidacji są dostępne z tego źródła.
cOcena braku DNA przy użyciu testu bez odwrotnej transkrypcji jest niezbędna przy pierwszej ekstrakcji RNA. Po potwierdzeniu, że próbka jest wolna od DNA, włączenie kontroli bez odwrotnej transkrypcji jest pożądane, ale nie jest już niezbędne.
dUjawnienie sekwencji sondy jest wysoce pożądane i zdecydowanie zalecane. Jednakże, ponieważ nie wszyscy komercyjni dostawcy wstępnie zaprojektowanych testów dostarczają tych informacji, nie może to być niezbędny wymóg. Stosowanie takich testów jest odradzane.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?