Zablokowany kwas nukleinowy i mniejsze spoiwo / Eclipse Dark Quencher
Locked Nucleic Acid i Minor Groove Binder / Elipse Dark Quencher (MGB:EDQ) to modyfikacje, które zwiększają wydajność oligonukleotydów. Zwiększają stabilność i poprawiają specyficzność oraz powinowactwo do parowania zasad z sekwencjami docelowymi. Zablokowany kwas nukleinowy wzmacnia startery i sondy (w połączeniu z BHQ™) w qPCR, a także skuteczność metod terapeutycznych, takich jak oligonukleotydy antysensowne (ASO). Tymczasem MGB:EDQ, z dołączonym wygaszaczem, szczególnie wzmacnia sondy w qPCR.
Zablokowany kwas nukleinowy
Locked Nucleic Acid to nowy rodzaj analogu kwasu nukleinowego, który zawiera mostek metylenowy 2'-O, 4'-C (Rysunek 1). Mostek ten zablokowany w konformacji 3'-endo ogranicza elastyczność pierścienia rybofuranozy i blokuje strukturę w sztywnej formacji bicyklicznej. Zapewnia to lepszą wydajność testu i zwiększony zakres zastosowań.

Rysunek 1.Struktura zablokowanego kwasu nukleinowego i monomerów DNA w stanie natywnym.
Zablokowany kwas nukleinowy w starterach PCR, sondach qPCR i innych typach oligonukleotydów jest rozpuszczalny w wodzie i standardowych buforach, a także jest zgodny z zasadami parowania zasad Watson-Crick.1
Zalety zablokowanego kwasu nukleinowego
Po włączeniu do oligonukleotydów, zablokowany kwas nukleinowy oferuje kilka korzyści w porównaniu do samych zasad DNA w stanie natywnym, w tym następujące:
- Zwiększona stabilność termiczna i specyficzność hybrydyzacji
- Dokładniejsza kwantyfikacja genów i rozróżnianie alleli
- Łatwiejsze i bardziej elastyczne projekty dla problematycznych sekwencji docelowych
Zwiększona stabilność termiczna i specyficzność hybrydyzacji
Włączenie zablokowanego kwasu nukleinowego do oligonukleotydów zwiększa stabilność termiczną dupleksu2 i poprawia specyficzność hybrydyzacji oligonukleotydów do sekwencji docelowych.3 W przypadku qPCR, zmniejsza to fluorescencję tła z fałszywego wiązania, co zwiększa stosunek sygnału do szumu. Ponadto, lepsza hybrydyzacja startera PCR lub sondy qPCR z celem może zwiększyć temperaturę topnienia (Tm) nawet o 8°C na zablokowaną sekwencję nukleinową.C na podstawienie monomeru zablokowanego kwasu nukleinowego w warunkach średniego zasolenia w porównaniu do oligonukleotydów DNA w stanie natywnym4 (Tabela 1). Ten wzrost hybrydyzacji powoduje znaczne poszerzenie zakresu warunków testu i pozwala na bardziej skuteczne multipleksowanie.5
Dokładniejsza kwantyfikacja genów i rozróżnianie alleli
Zdolność oligonukleotydów do rozróżniania alleli za pośrednictwem SNP jest znacznie zwiększona przez włączenie zablokowanych zasad kwasu nukleinowego6-8 (Rysunek 2). Obecność niedopasowania pojedynczej zasady ma większy destabilizujący wpływ na tworzenie dupleksu między oligonukleotydem zablokowanego kwasu nukleinowego a jego celem niż w przypadku oligonukleotydu DNA w stanie natywnym. Krótsze oligonukleotydy zawierające zablokowane zasady kwasu nukleinowego mogą być stosowane w tych samych temperaturach, co dłuższe oligonukleotydy DNA w stanie natywnym.

Rysunek 2.Podwójnie znakowane sondy z zablokowanym kwasem nukleinowym dyskryminują lepiej niż podwójnie znakowane sondy DNA w analizie genotypowania SNP9. Różowy) analiza zmutowanego DNA z sondą mutanta z zablokowanym kwasem nukleinowym (16mer z 3 zablokowanymi zasadami kwasu nukleinowego). Zielony) analiza zmutowanego DNA z sondą mutanta DNA w stanie natywnym (25mer). Czerwony) DNA typu dzikiego ze zmutowaną sondą DNA w stanie natywnym (25-merowa). Fioletowy) DNA typu dzikiego z zablokowaną sondą mutanta kwasu nukleinowego (16mer z 3 zablokowanymi zasadami kwasu nukleinowego).
Łatwiejsze i bardziej elastyczne projekty dla problematycznych sekwencji docelowych
Dzięki ulepszonej charakterystyce hybrydyzacji Locked Nucleic Acid wraz ze wzrostem Tm, oligonukleotydy Locked Nucleic Acid mogą być syntetyzowane tak, aby były krótsze, co przezwycięża pewne ograniczenia projektowe, które występują w przypadku oligonukleotydów DNA w stanie natywnym. W szczególności, oligonukleotydy z zablokowanym kwasem nukleinowym mogą być projektowane w celu rozwiązania tradycyjnie problematycznych sekwencji docelowych, takich jak regiony bogate w AT lub GC. Na przykład oligonukleotydy DNA w stanie natywnym bogate w AT często muszą mieć długość ponad 30 zasad (czasami ponad 40 zasad), aby spełnić wytyczne dotyczące projektowania amplikonów, ale nadal mogą działać słabo. W przypadku oligonukleotydów z zablokowanym kwasem nukleinowym, selektywne umieszczenie zablokowanych zasad kwasu nukleinowego ułatwia optymalne projektowanie wysoce specyficznych, krótszych oligonukleotydów, które działają dobrze, nawet przy długości zaledwie 13 do 20 zasad. Ponadto, projektowanie oligonukleotydów do namierzania trudnych SNP, takich jak względnie stabilne niedopasowanie G:T, jest znacznie ułatwione dzięki Locked Nucleic Acid.
Dodatkowe korzyści
Prymery PCR z Locked Nucleic Acid i sondy qPCR są kompatybilne ze wszystkimi termocyklerami czasu rzeczywistego i urządzeniami do wykrywania analitycznego punktu końcowego. Nie są potrzebne żadne specjalistyczne instrumenty.
Zastosowania zablokowanego kwasu nukleinowego
Zablokowany kwas nukleinowy można włączyć do wszystkich dostępnych chemii detekcji qPCR, w tym:
- SYBR® Green Primers
- Dual-Labeled Probes
- Molecular Beacons
- LightCycler® Probes
- Scorpions® Probes
Jest przydatny do następujących celów:
- Wykrywanie SNP
- Rozróżnianie alleli
- Wykrywanie patogenów
- Multipleksowanie
- Kwantyfikacja obciążenia wirusem
- Analiza ekspresji genów
- Określanie kopii genów
Może być również stosowany w tych sekwencjach:
- Oligonukleotydy antysensowne
- Oligonukleotydy decoy
- Sondy wychwytujące
- Aptamery
- Ribozymy
MGB:EDQ
Sonda MGB:EDQ to jednoniciowy oligonukleotyd znakowany dwoma różnymi barwnikami. Barwnik reporterowy znajduje się na końcu 5', a Minor Groove Binder / Eclipse Dark Quencher na końcu 3' (rysunek 1). Cząsteczka wygaszacza hamuje naturalną emisję fluorescencji reportera poprzez transfer energii rezonansu fluorescencji (FRET, rysunek 2).
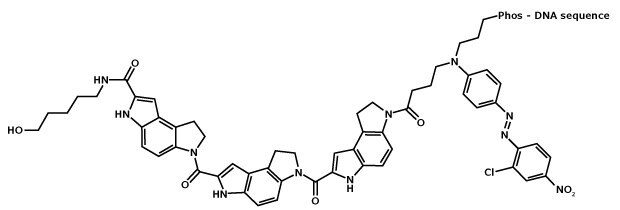
Rysunek 1.Struktura MGB:EDQ. MGB, lub CDPI3, jest tripeptydem dihydropiroloindolokarboksylanu, a jego bumerangowa krzywizna pozwala mu dopasować się do kształtu, a tym samym silnie wiązać się z mniejszym rowkiem DNA w kształcie litery B.
1. Sonda w roztworze emituje niską fluorescencję
2. Emisja fluorescencji przez hydrolizę
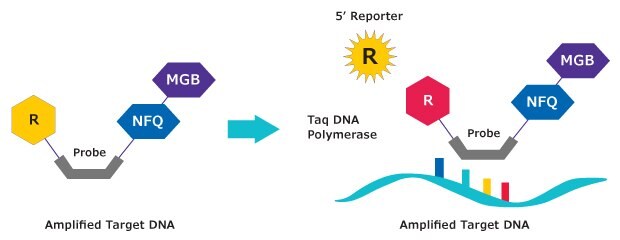
Rysunek 2.Mechanizm działania. Sondy MGB:EDQ są sondami hydrolizującymi. Starter jest wydłużany przez polimerazę, a sonda wiąże się z określoną matrycą DNA. Hydroliza uwalnia reporter z hybrydy sonda-cel, powodując wzrost fluorescencji. Zmierzony sygnał fluorescencji jest wprost proporcjonalny do ilości docelowego DNA.
Korzyści z MGB
MGB zwiększa wydajność qPCR na kilka sposobów:
- Zwiększona specyficzność: MGB zwiększa specyficzność hybrydyzacji sonda-cel, zmniejszając prawdopodobieństwo niespecyficznej amplifikacji
- Wyższa czułość: MGB umożliwia stosowanie krótszych sond, które zwiększają czułość testów qPCR
- Regulacja temperatury topnienia: MGB umożliwia regulację Tm sondy, co może być korzystne przy projektowaniu sond qPCR o optymalnych wartościach Tm
- Zmniejszony sygnał tła: MGB przyczynia się do zmniejszenia fluorescencji tła, co prowadzi do poprawy stosunku S:N lub stosunku sygnału do szumu
- Zwiększona stabilność: MGB może poprawić stabilność hybrydy sonda-cel, zmniejszając prawdopodobieństwo degradacji sondy
- Elastyczność projektu: Zastosowanie MGB rozszerza możliwości projektowania sond, ułatwiając tworzenie wydajnych testów qPCR
Sondy qPCR wzmocnione MGB są cennym narzędziem do uzyskiwania wiarygodnych i solidnych wyników qPCR.
APLIKACJE MGB:EDQ
MGB:EDQ można włączyć do tej chemii detekcji qPCR:
- Sondy podwójnie znakowane (z 6-FAM™, HEX™ i TET™; zapytaj o inne)
Jest to przydatne w następujących przypadkach:
- wykrywanie SNP poprzez dyskryminację niedopasowania
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?