PCR kolonii
Kolonijny PCR jest prostą i szybką metodą weryfikacji poprawności montażu sklonowanego konstruktu DNA. Tradycyjnie weryfikacja insercji DNA podczas klonowania była czasochłonna, po transformacji bakteryjnej z pracochłonnym oczyszczaniem DNA i późniejszym trawieniem enzymami restrykcyjnymi. Colony PCR to Bezpośrednia metoda PCR, która omija zarówno izolację DNA, jak i trawienie enzymami restrykcyjnymi, zapewniając szybkie, łatwe i niedrogie rozwiązanie do sprawdzania sklonowanych konstruktów. Aplikacje PCR kolonii są również kompatybilne z badaniami przesiewowymi klonów utworzonych metodami niezależnymi od sekwencji i restrykcji.
REDExtract-N-Amp™ PCR Ready Mix™ i Jump Start™ REDTaq® PCR Ready Mix™ dla Ckolonii PCR Aaplikacji
Nadaje się do stosowania w koloniach PCR, REDExtract-N-Amp™ PCR ReadyMix™ (Cat. R4775) i JumpStart™ REDTaq® PCR ReadyMix™ (Cat. No. P0982) dostarczają bufor, polimerazę, dNTP i stabilizatory jako pojedynczy 2x roztwór. Zostały one opracowane w celu umożliwienia bezpośredniego PCR, a następnie załadowania próbek na żel agarozowy po PCR bez potrzeby stosowania dodatkowych buforów lub barwników ładujących. Obojętny barwnik migruje z taką samą szybkością jak fragment o długości 125 par zasad, zapewniając wizualne potwierdzenie migracji DNA.
Features and Benefits
- Ultimate Flexibility: Oczyszczanie DNA przed amplifikacją nie jest konieczne. Nadaje się do stosowania z popularnymi szczepami E. coli, takimi jak SIG10 5a, NovaBlue (K-12), BL21(DE3) i DH5α, a także z drożdżami S. pombe. Próbka może być również ponownie amplifikowana, na przykład w zagnieżdżonym PCR.
- Wysoka specyficzność i wydajność: Polimeraza DNA JumpStart™ Taq, inaktywowany przeciwciałem enzym gorącego startu, został zaprojektowany w celu zminimalizowania niespecyficznej amplifikacji przy jednoczesnym zwiększeniu wydajności i specyficzności. JumpStart™ Taq amplifikuje cele o długości do 7 kb.
- Wygoda: Funkcje gorącego startu w produktach JumpStart™ umożliwiają przygotowanie reakcji w temperaturze pokojowej, co jest korzystne podczas pracy z wieloma próbkami. REDExtract-N-Amp™ PCR ReadyMix™ i JumpStart™ REDTaq® PCR ReadyMix™ są stabilne w temperaturze 4 °C lub -20 °C.
- Tolerancja na inhibitory: Wykazano, że JumpStart™ REDTaq® PCR ReadyMix™ toleruje stężenia LB do 20% i LB Agar lub Terrific Broth do 10%.
- Produkowalność: Zmniejsza ryzyko zanieczyszczenia w wyniku wielokrotnego pipetowania i zapewnia stałą wydajność w wielu reakcjach.
- Gel-ready: Nie trzeba dodawać buforu ładującego ani barwnika śledzącego. Produkt PCR jest ładowany bezpośrednio do żelu agarozowego po amplifikacji. Czerwony znacznik migruje w przybliżeniu z taką samą szybkością jak fragment 125bp (nieco szybciej niż błękit bromofenolowy).
Ckolonia PCR Pprotokół
Protokół PCR kolonii jest prosty: wystarczy dodać niewielką ilość kolonii z płytki lub gęstej płynnej kultury bakteryjnej do mieszanki wzorcowej PCR i przejść do termocyklingu. Liza komórek następuje podczas początkowej inkubacji w wysokiej temperaturze. Uzyskane produkty PCR można analizować za pomocą elektroforezy żelowej lub innych metod wykrywania DNA. Ta oszczędzająca czas metoda jest idealna do badań przesiewowych tylko kilku kolonii lub do wysokowydajnych badań przesiewowych wielu klonów.
1. Przygotuj mieszankę wzorcową PCR (skalowaną w oparciu o liczbę próbek do analizy):
2.
2. Dozować mieszaninę wzorcową PCR (20 µL) do każdej probówki lub płytki PCR.
3. Za pomocą sterylnej końcówki mikropipety lub sterylnej wykałaczki przenieś komórki z każdej kolonii do probówki PCR i krótko zamieszaj, aby ponownie zawiesić je w mieszaninie wzorcowej PCR. Mieszanina może wyglądać na lekko mętną. Uwaga: Nie należy pobierać zbyt wielu komórek. Przeładowanie komórek zakłóci PCR.
4. Amplifikuj docelowe DNA w następujących warunkach termocyklingu:
Uwaga: Etap lizy komórek jest dłuższy niż typowa początkowa denaturacja, aby skutecznie rozbić ścianę bakterii
Ckolonia PCR Azastosowania z Bbakteriami i Ydrożdżami
Kolonię PCR przeprowadzono przy użyciu reprezentatywnych szczepów bakterii i drożdży. E. coli zostały przebadane pod kątem obecności wstawek przy użyciu REDExtract-N-Amp™ PCR ReadyMix™ i starterów flankujących, które amplifikowały 1 kbp wstawki. Wszystkie szczepy E. coli były kompatybilne z koloniami PCR, wytwarzając docelowe amplikony bezpośrednio z surowych danych wejściowych komórek bez etapu ekstrakcji DNA (Rysunek 1).

Rysunek 1Żel agarozowy produktów PCR kolonii E. coli. PCR kolonii wstawki 1 kbp przeprowadzono przy użyciu REDExtract-N-Amp™ PCR ReadyMix™ i analizowano na 1% żelu agarozowym. Produkty zostały wykryte ze wszystkich transformowanych komórek zawierających pET28a(+) z insertami (próbki 3-10), podczas gdy żaden produkt nie został amplifikowany z komórek transformowanych pustym wektorem pET28a(+) (próbki 1-2).
Drożdże Schizosaccharomyces pombe transformowano plazmidem pESP-1. PCR przeprowadzono na uzyskanych koloniach i kontroli plazmidu. Kolonie drożdży zostały przebadane pod kątem obecności insertów przy użyciu JumpStart™ REDTaq® PCR ReadyMix™ i starterów flankujących, które amplifikowały 226 bp insertu. Podobnie jak w przypadku E. coli, sekwencje docelowe zostały amplifikowane z próbek drożdży bez oczyszczania DNA (Rysunek 2).

Rysunek 2Żel agarozowy produktów PCR kolonii S. pombe. PCR kolonii wstawki 226 bp przeprowadzono przy użyciu JumpStart™ REDTaq® PCR ReadyMix™ i analizowano na 1% żelu agarozowym. Produkty wykryto ze wszystkich transformowanych komórek (kolonia 1-3) i matrycy plazmidu (kontrola).
Eefekt z Growth Media na PCR Ypolu Using Jump Start™ REDTaq® PCR Ready Mix Rodczynniki
Aby określić ilościowo wpływ zanieczyszczenia pożywki wzrostowej na PCR, powszechne E. coli media LB, LB Agar i Terrific Broth (TB) zostały dodane do odczynników JumpStart™ REDTaq® PCR ReadyMix™. Amplifikację przeprowadzono z pBR322 jako matrycą, odpowiednimi starterami, α [32P] dCTP i różnymi poziomami (0 - 50%) LB, LB Agar i TB. Wydajność PCR określono po wytrąceniu kwasu trichlorooctowego na szklanych filtrach, a następnie zliczeniu scyntylacyjnym. Wydajność znormalizowano do 0% dodanej pożywki. Reakcje przeprowadzono w trzech powtórzeniach. Wszystkie dodane pożywki miały wpływ na wydajność produktu, ale PCR z użyciem JumpStart™ REDTaq® PCR ReadyMix™ był nadal stabilny przy do 20% mieszaniny reakcyjnej składającej się z LB i do 10% mieszaniny reakcyjnej składającej się z agaru TB lub LB (Rysunek 3). Poziomy te przekraczają to, co zanieczyściłoby typową kolonię PCR i wykazują wysoką tolerancję na inhibitory odczynników JumpStart™ REDTaq® PCR ReadyMix™.
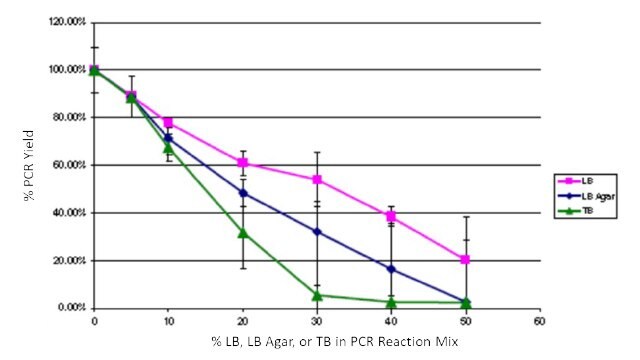
Rysunek 3Efekty dodawania LB, agaru LB i TB do odczynników PCR ReadyMix™. LB, różowe kwadraty; agar LB, niebieskie romby; TB, zielone trójkąty.
Colony PCR Performance z Jump StartTM oraz REDTaq® Ready Mix™ Reagents
REDExtract-N-Amp™ PCR ReadyMix™, JumpStart™ REDTaq® PCR ReadyMix™ i inne odczynniki JumpStart™ i REDTaq® ReadyMix™, takie jak R2523 i P1107, są solidnymi mieszaninami wzorcowymi, które umożliwiają bezpośrednią reakcję PCR kolonii i kultur z popularnych szczepów do klonowania i ekspresji, w tym E. coli i S. pombe, ułatwiając szybkie przesiewanie sklonowanych konstruktów. Mają wysoką tolerancję na powszechnie stosowane inhibitory PCR, w tym stężenia LB do 20% i LB Agar lub Terrific Broth do 10%, co skutkuje wysoką wydajnością w kolonii PCR. Mieszaniny wzorcowe mogą przezwyciężyć wpływ resztek komórkowych i mogą być stosowane bezpośrednio z surowymi koloniami lub zawiesinami do amplifikacji celów o długości do 7 kb. Dodatkowe ultraszybkie mieszanki wzorcowe PCR, w tym nasza KOD One™ PCR Master Mix są również dostępne i odpowiednie do zastosowań PCR w koloniach. Dodatkowe odczynniki i zasoby do PCR można znaleźć w naszym centrum zasobów odczynników i zestawów do PCR.
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?