Rozwiązywanie problemów z RT-PCR
Na tej stronie
- Opracowanie protokołu rozwiązywania problemów
- Optymalizacja projektowania oligo
- Optymalizacja testów PCR
- Projektowanie testów RT-PCR i qPCR
- Jakość szablonu RNA lub DNA
- Program testu PCR
- Nieprawidłowe działanie termocyklera
- Przykłady rozwiązywania problemów: Narzędzia diagnostyczne
- Krzywe dysocjacji / topnienia
- Studia przypadków rozwiązywania problemów RT-PCR
- Podsumowanie - Lista kontrolna rozwiązywania problemów PCR
Opracowanie protokołu rozwiązywania problemów PCR lub RT-PCR
Potencjalne źródła błędów i/lub problemów Błąd operatora
Istnieje wiele możliwości popełnienia błędu przez operatora. Źródła tych błędów często pozostają niezidentyfikowane. Pierwszym krokiem w każdej procedurze rozwiązywania problemów jest sprawdzenie protokołu i powtórzenie eksperymentu. Sprawdzenie protokołu (patrz Appendix A, Protokoły, niniejszego przewodnika) i poproszenie doświadczonego biologa molekularnego o sprawdzenie planu eksperymentu jest ważne. Opowieść o pracowniku naukowym, który przeprowadził kilka nieudanych reakcji PCR, zanim zdał sobie sprawę, że w PCR master mix przypomina, że nawet najlepsi przepracowani naukowcy są podatni na proste błędy.
Master Mix
Błędy lub problemy z mieszanką składników reakcji mogą być źródłem katastrofalnego niepowodzenia amplifikacji we wszystkich próbkach i kontrolach pozytywnych. Przed powtórzeniem eksperymentu należy sprawdzić wszystkie składniki i ich stężenia. Jeśli używana jest nowa partia odczynnika, przydatnym środkiem ostrożności jest porównanie nowego odczynnika ze starym przed rozpoczęciem głównej serii eksperymentów.
Przy zmianie produktów typu master mix należy pamiętać, że niektóre testy są szczególnie wrażliwe na skład buforu / temperaturę wyżarzania (Ta) / kombinacje stężeń starterów. Zmiana któregokolwiek z nich może skutkować inną wydajnością. Dlatego przed wprowadzeniem radykalnych zmian należy zweryfikować wszystkie testy w wybranych mieszaninach wzorcowych i na wszystkich pożądanych urządzeniach. Istotne jest również zapoznanie się z instrukcjami dostarczonymi z każdą mieszaniną wzorcową, ponieważ określają one zalecane warunki, które są zoptymalizowane dla danego enzymu, mechanizmu Hot Start i składników buforu.
| Problem | JumpStart™ Taq ReadyMix™ nie działa tak dobrze jak podobny produkt od innego dostawcy |
|---|---|
| Możliwe przyczyny |
|
| Test diagnostyczny |
|
| Roztwór |
|
| Problem | PCR ReadyMix działa dla PCR, ale nie dla qPCR, ale równoważne produkty od innego dostawcy działają dobrze |
|---|---|
| Możliwe przyczyny |
|
| Test diagnostyczny |
|
| Rozwiązania |
|
Dobrą praktyką laboratoryjną jest przygotowanie wystarczającej ilości wzorcowej mieszaniny reakcyjnej dla wszystkich próbek, które mają być przeprowadzone razem. Należy upewnić się, że wszystkie składniki są starannie rozmrożone i dobrze wymieszane, a mieszanina wzorcowa eksperymentu jest bardzo dobrze wymieszana przed podzieleniem na próbki. Jest to szczególnie istotne w przypadku niektórych buforów 2×, takich jak KiCqStart® które są bardziej lepkie niż zwykłe bufory PCR.
Oligo Optimization
Oligo mogą powodować problemy, jeśli mają nieprawidłową sekwencję lub są źle zaprojektowane, działają w nieoptymalnym stężeniu, nieoptymalnym Ta lub są nieodpowiednio oznakowane lub wygaszone (w przypadku sond). Test przeprowadzony w nieoptymalnych warunkach dla oligo lub przy użyciu złego projektu może dostarczyć pewnych danych, jednak może to nie odzwierciedlać prawdziwej biologii. Po otrzymaniu liofilizowanego oligo bardzo ważne jest, aby:
- zweryfikować sekwencję
- upewnić się, że całe DNA zostało ponownie zawieszone przed użyciem
- upewnić się, że roztwór ma oczekiwane stężenie
- upewnić się, że roztwór ma oczekiwane stężenie.Potwierdź, że roztwór ma oczekiwane stężenie
Resuspensuj oligo poprzez podgrzanie oligo do 90 °C przez 5 minut, a następnie dobrze wymieszaj. Powtarzające się cykle zamrażania i rozmrażania mogą również wpływać na wydajność oligo, dlatego wszystkie oligo w stężeniu podstawowym (zwykle 100 μM) powinny być podzielone na porcje i przechowywane w temperaturze -20 °C lub -80 °C przez dłuższy czas.
Podczas fazy rozwiązywania problemów kluczowe znaczenie ma sprawdzenie, czy zamówiono prawidłową sekwencję, powracając do sekwencji docelowej i potwierdzając, że sekwencje oligo są rzeczywiście obecne. Upewnij się, że jakość oligo była prawidłowa, kontaktując się z dostawcą oligo. Zmierz stężenie robocze oligo i wizualnie sprawdź cząsteczki fluorescencyjne, aby potwierdzić, że są one oznakowane. Przetestuj startery sond w mieszaninie SYBR® Green I qPCR, aby zweryfikować amplifikację. Rozważ optymalizację stężeń starterów lub Ta (patrz Optymalizacja i walidacja testu). Używając sondy po raz pierwszy, należy zebrać dane fluorescencyjne dla jak największej liczby potencjalnych długości fal, aby można było zaobserwować potencjalny wyciek sygnału między kanałami i wykryć błędy w znakowaniu.
Nieodpowiednia optymalizacja PCR
.Efekt optymalizacji testu został opisany i zademonstrowany w Optymalizacja i walidacja testu. Gdy test zawodzi lub działa nieoptymalnie, ale nie ma błędów w projekcie lub procedurach operacyjnych, może skorzystać z optymalizacji warunków eksperymentalnych. Podczas rozwiązywania problemów należy przetestować startery przy końcowym stężeniu 100 nM, 500 nM i 900 nM i/lub Ta od 55 °C do 70 °C (przy użyciu gradientu temperatury), aby określić, czy test poprawi się wraz z dalszą optymalizacją.
RT-PCR and qPCR Assay Design
.Projekt testu został opisany w PCR/qPCR/dPCR Assay Design. Podczas rozwiązywania problemów z testem należy upewnić się, że projekt został zweryfikowany. Upewnij się, że starter PCR/qPCR i pozycja amplikonu są zgodne z protokołem starterowania RT. Na przykład upewnij się, że testy zastosowane do cDNA, które zostało przygotowane po primingu oligo-dT, znajdują się w kierunku 3' transkryptu. Upewnij się, że informacje o sekwencji są wiarygodne i że uwzględniono odpowiednie warianty splicingu i SNP.
| Problem | Test jest nieczuły, a wykresy amplifikacji wyglądają nieprawidłowo (Rysunek 11.1) |
|---|---|
| Możliwe przyczyny |
|
| Test diagnostyczny |
|
| Rozwiązania |
|

Rysunek 11.1A.Test ma nietypowy profil wykresu amplifikacji z wyraźnym dryftem linii bazowej.

Rysunek 11.1B.Sekwencja sondy, która została włączona do testu, została wprowadzona do oprogramowania mfold do przewidywania fałdowania. Oczywiste jest, że sonda może przyjąć stabilną strukturę fałdową w roztworze, co prawdopodobnie spowodowało zaobserwowany problem.
Jakość szablonu RNA lub DNA
Wpływ jakości szablonu na wydajność testu został opisany w Oczyszczanie próbek i ocena jakości. Jakość szablonu obejmuje ilość, integralność i obecność inhibitorów. Kluczowe znaczenie ma zapewnienie, że jakość RNA jest dopasowana do najbardziej odpowiedniego protokołu primingu RT (patrz Reverse Transcription) i użycie szablonu najlepszej możliwej jakości. Podobnie, ilość RNA dodawanego do reakcji RT musi mieścić się w zakresie protokołu i w wielu przypadkach powinna być taka sama dla wszystkich reakcji. ReadyScript® jest godnym uwagi wyjątkiem od tej wytycznej, ponieważ przyjęcie tego odczynnika i protokołu daje liniowe stężenie cDNA, które jest proporcjonalne do wejściowej ilości RNA. Podczas rozwiązywania problemów z próbką, która daje wyższe niż oczekiwane Cq, przeprowadź test SPUD lub rozcieńcz próbkę w serii rozcieńczeń 1:5 lub 1:10 i powtórz test (rysunek 11.2), aby zidentyfikować próbki, które zawierają inhibitory.
| Problem | Dane Cq dla rozcieńczeń krzywej standardowej są nieregularnie rozmieszczone (Rysunek 11.2) |
|---|---|
| Możliwe przyczyny |
|
| Test diagnostyczny |
|
| Rozwiązania |
|


Rysunek 11.2.Amplifikacja 10-krotnego seryjnego rozcieńczenia matrycy DNA. Replikacje są dokładne, ale ΔCq jest niespójne, zmniejszając się wraz ze wzrostem rozcieńczeń. Dane pokazują również pozytywny sygnał w kontroli bez matrycy (NTC) wskazujący na zanieczyszczenie lub tworzenie dimerów starterów, a rozcieńczenia do mniej niż 105 kopii mają identyczne dane jak NTC.
| Rozcieńczenie | ΔCq |
|---|---|
| 107 → 106 | 5.4 |
| 106 → 105 | 5.0 |
| 105 → 104 | 3.6 |
Ilość matrycy jest również ważnym czynnikiem. Włączenie zbyt dużej lub zbyt małej ilości matrycy do reakcji PCR spowoduje nieudane reakcje i nieprawidłowe wykresy amplifikacji qPCR. Rysunek 11.3A przedstawia reakcję zawierającą 10-krotne seryjne rozcieńczenie sztucznej matrycy oligo. Niższe rozcieńczenia są zbyt skoncentrowane, aby reakcja była wydajna lub aby urządzenie mogło skutecznie przetwarzać dane wyjściowe (Rysunek 11.3B), co skutkuje nieprawidłowymi wykresami amplifikacji i niewiarygodnymi danymi.

Rysunek 11.3. A)Amplifikacja 10-krotnego seryjnego rozcieńczenia sztucznej matrycy ze specyficznymi starterami i sondą znakowaną FAM. Cq jest bardzo niskie dla stężonych próbek, wykresy amplifikacji nie są regularnie rozmieszczone i są nieprawidłowe. B) Pokazuje surowe dane dla tych wykresów amplifikacji. Reakcje zawierające najwyższe stężenie celu mają również znacznie wyższą fluorescencję tła i minimalną wydajność fluorescencji w reakcji.
Program PCR do testu
Warunki cyklu PCR muszą być odpowiednie zarówno dla eksperymentu, jak i odczynników (np,patrz Master Mix). Akceptacja domyślnych ustawień urządzenia bez weryfikacji jest niewskazana.
.Usterka termocyklera
Usterki przyrządów mogą mieć podstępny początek i dlatego mogą być trudne do zdiagnozowania. Aby zapobiec kosztownym naprawom, należy upewnić się, że wszyscy operatorzy przyrządów są w pełni przeszkoleni i początkowo nadzorowani. Niektóre usterki instrumentów powodują katastrofalne awarie, skutkujące brakiem amplifikacji lub danych fluorescencyjnych, podczas gdy inne zniekształcają dane lub traktują próbki w niejednolity sposób, tworząc sztuczne różnice między identycznymi próbkami biologicznymi. Wykorzystanie próbek kontrolnych z testami kontrolnymi jest nieocenione przy rozwiązywaniu problemów. W przypadku podejrzenia usterki urządzenia, we wszystkich studzienkach należy przeprowadzić wiarygodny, zoptymalizowany test. Ta kontrola jednorodności ujawni problemy, które są specyficzne dla regionów urządzenia, a także oddzielne problemy związane z testem i urządzeniem.
.Przykłady rozwiązywania problemów PCR demonstrujące użycie narzędzi diagnostycznych
Po przeprowadzeniu dobrze zaplanowanej reakcji PCR dostępnych jest kilka narzędzi diagnostycznych do rozwiązywania problemów:
- Próbki kontrolne i testy
- End-point gel/SYBR Green I odczynnik barwiący
- Wykresy amplifikacji (sprawdź replikaty i profil wykresu amplifikacji)
- Krzywe standardowe (gradient i R2)/serie rozcieńczeń
- Wykresy topnienia/dysocjacji (barwnik SYBR Green I, Molecular Beacons, Scorpions® Probes)
- Raw data/multicomponent views
Control Samples/Reactions
Użycie kontroli jest zdecydowanie zalecane. Rozwiązywanie problemów z nieudanym testem jest prawie niemożliwe bez informacji z odpowiedniego zestawu kontroli.
| Kontrola | Przykładowy materiał | Oczekiwany wynik | Możliwe przyczyny pozytywnego wyniku Pozytywny wynik | Możliwe przyczyny Negatywnego wyniku |
|---|---|---|---|---|
| Próbka pozytywna | Próbka, o której wiadomo, że zawiera sekwencje testu, np.RNA/gDNA wyrażające/ zawierające cel | Pozytywna | Poprawna | Niepowodzenie testu. Wszelkie pozytywne dane z innych próbek są niewiarygodne. |
| Kontrola pozytywna | Dowolny kwas nukleinowy zgodny z projektem testu PCR np.sztuczny oligo-nukleotyd lub plazmid zawierający amplikon PCR. | Dodatni | Prawidłowy | Niepowodzenie testu. Wszelkie pozytywne dane z innych próbek są niewiarygodne. |
| Kontrola negatywna | Próbka, o której wiadomo, że nie zawiera sekwencji testu, np. RNA/gDNA nie ulega ekspresji/nie zawiera celu. | Negatywny | Test jest niespecyficzny lub nastąpiło zanieczyszczenie kontroli podczas przygotowania PCR./td> | Prawidłowy |
| .Zanieczyszczenie Negatywna kontrola testu (No Template Control NTC) | Woda | Negatywny | Prymery ulegają samodimeryzacji, w wyniku czego powstaje produkt dimeryzacji primerów lub nastąpiło zanieczyszczenie kontroli podczas przygotowywania PCR. | Prawidłowy |
| Kontrole specyficzne dla RT | Przykładowy materiał | Oczekiwany wynik | Możliwe powody uzyskania wyniku pozytywnego Wynik pozytywny | Możliwe przyczyny wyniku negatywnego |
|---|---|---|---|---|
| Kontrola negatywna enzymu minus RT | Próbka RNA i wszystkie składniki reakcji RT z wyjątkiem enzymu RT. Należy to wykonać na wszystkich próbkach, aby sprawdzić, czy nie zawierają one sekwencji, które ulegają amplifikacji w warunkach PCR bez potrzeby RT, np. zanieczyszczenie gDNA | Negatywny | Próbka zawiera gDNA. Reakcja została zanieczyszczona podczas konfiguracji. Startery utworzyły produkty dimerów starterów. Analizuj w połączeniu z NTC | Prawidłowe |
| Specyficzne dla genotypowania | Przykładowy materiał | Oczekiwany wynik | Możliwe przyczyny wyniku pozytywnego | Możliwe przyczyny wyniku negatywnego |
|---|---|---|---|---|
| Pozytywny cel specyficzny dla testu. | Szablon typu dzikiego (WT) wykryty w teście WT i zmutowany cel wykryty w teście mutacji. | Dodatni | Poprawny |
|
| Szablon WT wykryty w teście mutanta i zmutowany cel wykryty w teście WT. | Negatywny (lub stosunkowo wysokie Cg w porównaniu do kontroli pozytywnej). |
| Poprawne | |
| Kontrola heterozygoty w teście | Szablon heterozygoty lub mieszanka 1:Mieszanka 1: 1 każdej homozygoty wykrytej w teście WT i teście mutanta. | Dodatnie w obu testach. | Poprawne |
|
| Problem | Kontrola pozytywna amplifikuje, ale nie ma wyników amplifikacji z próbki, o której wiadomo, że zawiera cel (Rysunek 11.4A) |
|---|---|
| Możliwe przyczyny |
|
| Test diagnostyczny |
|
| Rozwiązania |
|

Rysunek 11.4. A)Nierozcieńczona matryca nie ulega amplifikacji, podczas gdy rozcieńczenia wykazują lepszą wydajność amplifikacji. B) Dodanie 0,3% BSA do mieszaniny qPCR wspomaga amplifikację z nierozcieńczonej matrycy.
Dochodzenie do całkowicie nieudanego testu może być trudne, ponieważ istnieje niewiele informacji do pracy z rozwiązywaniem problemów. Ponieważ wiele niepowodzeń testów jest wynikiem jakiegoś katastrofalnego błędu, pierwszym sprawdzeniem powinno być zweryfikowanie konfiguracji eksperymentu, a następnie powtórzenie PCR. Jeśli to się nie powiedzie, proces rozwiązywania problemów zależy od informacji dotyczących każdego składnika eksperymentu (Rysunek 11-5).
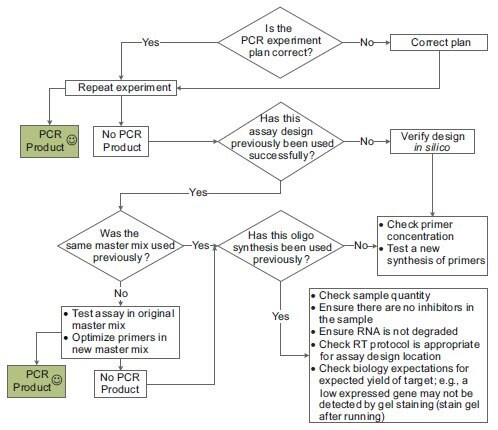
Rysunek 11.5.Podstawowy proces rozwiązywania problemów dla PCR.
Kiedy eksperyment qPCR całkowicie się nie powiedzie, pierwszym krokiem jest sprawdzenie projektu testu, sekwencji oligo i danych QC od producenta oligo. Chociaż test mógł zakończyć się niepowodzeniem, wieloskładnikowe/przetworzone dane qPCR mogą być wykorzystane do dostarczenia dalszych informacji. Rysunek 11.6A przedstawia wykres nieprzetworzonych danych dla dwóch testów zawierających sondę znakowaną 6-FAM™ lub HEX™ (VIC®). Chociaż oba testy wykazują wzmocnienie, sygnał HEX jest w przybliżeniu o połowę mniejszy niż sygnał FAM. Ponieważ jest to z natury słabszy barwnik, jest to normalna obserwacja. Analiza żelu agarozowego (Rysunek 11.6B) pokazuje, że obie reakcje dają podobne stężenie produktów, potwierdzając obserwację, że wartości qPCR Cq są podobne.
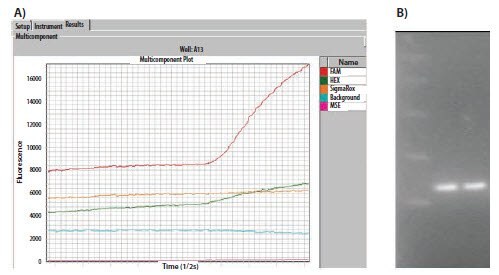
Rysunek 11.6. A)Wykresy surowych danych z testu dupleksowego zawierającego sondę znakowaną FAM i HEX. Sonda FAM naturalnie daje wyższą fluorescencję. B) Żel agarozowy pokazujący, że w każdej reakcji wytworzono równe ilości produktu i potwierdzający obserwację qPCR Cq.
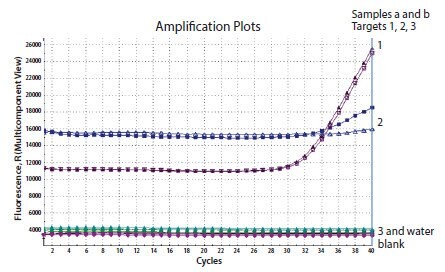
Badanie surowych danych jest przydatne do sprawdzenia, czy sonda jest prawidłowo oznakowana i została dodana do reakcji. Rysunek 11.7 przedstawia surowe dane dla amplifikacji trzech celów w eksperymencie triplex. Sondy specyficzne dla każdego celu są oznaczone FAM, HEX i TAMRA. Sondy HEX i TAMRA wykazują niskie tło i wydajną amplifikację, jednak sygnał FAM jest stale wysoki przez cały czas trwania eksperymentu i nie ma dowodów na amplifikację. Jest to zgodne ze zbyt wysokim stężeniem sondy w reakcji lub wadą sondy, która nie powoduje początkowego wygaszenia sygnału. W takich przypadkach należy zweryfikować stężenie sondy i projekt testu, upewniając się, że sonda ma kompatybilną etykietę i wygaszacz oraz, jeśli to konieczne, przetestować nową sondę.

Rysunek 11.7.Reakcja triplex została użyta do wykrycia trzech celów przy użyciu sond znakowanych FAM, HEX i TAMRA. Sondy HEX i TAMRA dały amplifikację z celów, ale sonda FAM nie wykazała amplifikacji. Analiza surowych danych wykazała, że fluorescencja tła była wyjątkowo wysoka i nie zaobserwowano żadnej różnicy podczas całej reakcji. Jest to zgodne ze zbyt wysokim stężeniem sondy w reakcji lub wadliwą sondą z nieodpowiednim wygaszaniem.
Jeśli pierwotny eksperyment opierał się na wykrywaniu sondy, test należy powtórzyć przy użyciu odczynników SYBR Green I, w tym kontroli pozytywnej i negatywnej (ale nie cennych próbek). Alternatywnie, produkty nieudanej reakcji można sprawdzić na żelu agarozowym barwionym bromkiem etydyny. Przyjęcie podejścia SYBR Green I do powtarzania eksperymentu jest preferowane, ponieważ pozwala uniknąć ryzyka zanieczyszczenia i zapewnia powtórzenie eksperymentu w celu zweryfikowania początkowego niepowodzenia. Jeśli eksperyment SYBR Green I dostarczy danych, możliwe jest, że pierwotna awaria sondy była spowodowana błędem technicznym lub usterką sondy. Aby odróżnić błąd eksperymentalny od usterki sondy, należy powtórzyć eksperyment z sondą; jeśli reakcja ponownie się nie powiedzie, należy wymienić sondę. Podejście to można zastosować do badania reakcji, które generują słabe dane. W przykładzie pokazanym na Rysunku 11.8 reakcja sondy była nieoptymalna, a po porównaniu z reakcją przeprowadzoną przy użyciu SYBR Green I można zauważyć, że sygnał sondy nie odzwierciedla eksperymentu. W takich przypadkach należy zweryfikować projekt testu i przetestować nową sondę.

Rysunek 11.8.Przeprowadzono identyczne reakcje zawierające sondę qPCR lub barwnik SYBR Green I (jak wskazano). Reakcja SYBR Green I była około jedenastu cykli bardziej czuła i dała znacznie wyższą fluorescencję punktu końcowego. Wskazuje to na błąd w sondzie lub problem z jej konstrukcją (dane uprzejmie dostarczone przez Prof. Stephen Bustin, UK).
Weryfikacja znakowania sondy
Nieprzetworzone dane lub wykres wieloskładnikowy są użytecznym narzędziem diagnostycznym do sprawdzenia, czy do reakcji włączono odpowiednie stężenie sondy oraz czy sonda jest odpowiednio wyznakowana i wygaszona.Rysunek 11.9 przedstawia wykres wieloskładnikowy dla reakcji zawierającej trzy sondy. Pierwsze dwie generują wykresy amplifikacji i widoczna jest fluorescencja tła. Nie ma danych z trzeciej sondy, a analiza surowych danych ujawnia, że fluorescencja tła jest równoważna kontroli ślepej próby wodnej, która nie zawiera żadnej sondy. Dlatego dane te są wynikiem braku fluorescencji w reakcji. Może to być spowodowane błędem podczas konfiguracji, w której sonda nie została uwzględniona lub sonda nie została oznakowana.
Rysunek 11.9.Trzy geny zostały wykryte w tej samej próbce. Dwie reakcje zakończyły się amplifikacją (1 i 2), jednak trzecia reakcja była negatywna. Badanie widoku wieloskładnikowego ujawnia, że fluorescencja tła dla trzeciej reakcji jest równoważna kontroli wody, co wskazuje na brak sygnału.
Dalszą kontrolę znakowania sondy można przeprowadzić za pomocą trawienia DNazą I. Należy to wykonać z najwyższą ostrożnością, aby upewnić się, że zapasy sondy i starterów nie są zanieczyszczone enzymem, co doprowadziłoby do katastrofalnych wyników. Podwielokrotność uszkodzonej sondy (Rysunek 11.10A) równoważna tej zawartej w reakcji, np. 300 nM jest inkubowana z i bez DNazy I. Można to przeprowadzić w czasie rzeczywistym (Rysunek 11.10B), tak aby wydajność fluorescencji była mierzona w odniesieniu do czasu lub alternatywnie, odczyt punktu początkowego i końcowego (po 10 minutach) dostarcza wystarczających informacji. Podczas wykonywania tego testu ważne jest, aby porównać dane z sondą, która działa dobrze i ma tę samą etykietę fluorescencyjną i wygaszacz (Rysunek 11.10B).

Rysunek 11.10. A)Dwa szablony zostały wykryte przy użyciu różnych sond, obie znakowane FAM. Podczas gdy detekcja przy użyciu jednej sondy skutkowała wysokim sygnałem fluorescencyjnym, druga była znacznie słabsza. B) Kontrolę i sondę testową (300 nM) inkubowano w temperaturze 37 °C w aparacie czasu rzeczywistego w buforze DNase I w obecności lub nieobecności enzymu DNase I. Uwalnianie fluorescencji z sondy 1 było około dwukrotnie większe niż z sondy 2, co wskazuje, że znakowanie sondy 2 było niewystarczające.
| Problem | Niska fluorescencja lub jej brak zarówno w próbce testowej, jak i w kontroli pozytywnej. Prawidłowy produkt PCR jest widoczny na żelu, a projekt jest weryfikowany | ||
|---|---|---|---|
| Detekcja oparta na zielonym barwniku SYBR | Wykrywanie sondy | ||
| Możliwa przyczyna | Zły barwnik wiążący SYBR Green I. |
|
|
| Test diagnostyczny |
|
|
|
| Rozwiązania | Zakup nowego barwnika wiążącego SYBR Green I lub nowej mieszaniny qPCR z barwnikiem wiążącym SYBR Green. |
| Zakup nowej sondy. |
Wykresy amplifikacji
Struktura wykresów amplifikacji i odtwarzalność powtórzeń technicznych dostarczają wielu informacji dotyczących jakości testu qPCR, a także mogą stanowić pierwsze sygnały ostrzegawcze, że wszystko jest nie tak, jak powinno. Wykresy amplifikacji na Rys. 11.11A są nietypowe, bardzo zaszumione i trudne do dokładnej interpretacji. Dalsze badanie wartości fluorescencji dR ujawnia, że wydajność fluorescencji w punkcie końcowym wynosi tylko 400 jednostek, co wskazuje, że reakcja jest nieodpowiednia, ale wykresy amplifikacji zostały wygenerowane przez oprogramowanie urządzenia i skalowane automatycznie. Podobnie, dane na Rysunku 11.11B mają wyraźny ogon (malejąca krzywa) na początku profilu, przed ponownym wzrostem po sekcji linii bazowej. Wygląd lisiego ogona jest spójny w dwóch reakcjach, ale jedna reakcja ma znacznie niższy punkt końcowy (Rysunek 11.11C), co skutkuje wzmocnionym, względnym lisim ogonem.
Rysunek 11.11. A) Wykresy amplifikacji, które są zaszumione z powodu automatycznego skalowania przez oprogramowanie przyrządu słabych danych o niskiej fluorescencji. B) Reakcje dające niski punkt końcowy dR mają wyraźny początkowy lisi ogon. C) Lisi ogon jest postrzegany jako normalny efekt, gdy jest proporcjonalny do wysokiej jakości testu.
Podobnie, wykresy amplifikacji na Rysunku 11.12A są wyraźnie nieprawidłowe i nie mogą być używane w takiej postaci, w jakiej zostały przedstawione. Wykres amplifikacji, który spada poniżej zera dR (Rysunek 11.12A) jest klasycznym wskaźnikiem zastosowania niewłaściwych ustawień linii bazowej. Badanie surowych danych dla tej reakcji (Rysunek 11.12B) pokazuje, że rzeczywiste wykresy amplifikacji mają normalny profil, potwierdzając, że analizowane dane są wynikiem błędu oprogramowania urządzenia. Odpowiednią linię bazową można wydedukować z surowych danych i zastosować w oprogramowaniu. W tym przypadku cykle od 6 do 16 reprezentują początkową liniową, podstawową fazę reakcji i po zastosowaniu skutkują normalnymi wykresami amplifikacji (Rysunek 11.12C).

Rysunek 11.12. A)Wykresy amplifikacji były wyraźnie nieprawidłowe, a część profilu spadła poniżej linii podstawowej. B) Analiza wykresu surowych danych pokazuje, że dane reakcji są zgodne z oczekiwaniami. C) Ustawienie linii bazowej urządzenia zgodnie z odpowiednimi cyklami przywraca normalny profil danych analizowanych wykresów amplifikacji.
Profil wykresu amplifikacji może być również interpretowany w celu dostarczenia informacji o jakości testu i optymalizacji. Rysunek 11.13 pokazuje próbę amplifikacji 10-krotnego seryjnego rozcieńczenia matrycy z każdym stężeniem przeprowadzonym w duplikacie qPCR. Odtwarzalność między powtórzeniami jest słaba, różnica cykli (ΔCq) między danymi nie jest stała i nie wynosi 3,323 cykli, jak oczekiwano dla 10-krotnego seryjnego rozcieńczenia. Analiza wykresów amplifikacji, z uwzględnieniem tego, że jest to krzywa standardowa, ujawnia, że test jest poniżej standardu i nie może być użyty do analizy. Przyczyny wymagają dalszego zbadania, ale mogą być wynikiem; złego projektu testu (patrz PCR/qPCR/dPCR Assay Design), nieoptymalne warunki testu (patrz Assay Optimization and Validation) lub słabe pipetowanie (powtórzenie testu).

Rysunek 11.13.Próbka cDNA została rozcieńczona w 10-krotnym seryjnym rozcieńczeniu, a specyficzna matryca została wykryta przy użyciu duplikatów qPCR dla każdego rozcieńczenia. Replikacje są słabe, co wskazuje na problem z pipetowaniem lub optymalizacją testu.
| Problem | Powierzchnie fluorescencji nagle rosną w górę (Rysunek 11.14). |
|---|---|
| Możliwe przyczyny | Zły barwnik referencyjny lub błąd urządzenia (np, drzwiczki zostały otwarte podczas pracy). |
| Test diagnostyczny |
|
| Rozwiązania |
|

Rysunek 11.14.Podczas standardowego qPCR dane nagle rosną w górę z nietypowym profilem.
| Problem | Spadające lub zahaczone wykresy fluorescencji (Rysunek 11.15) |
|---|---|
| Możliwe przyczyny | W miarę gromadzenia się produktu PCR, komplementarna nić konkuruje ze starterem i/lub sondą o annealing do matrycy. |
| Test diagnostyczny | Patrz Rysunek 11.15. |
| Rozwiązania | Odrzuć, jeśli nie ma to wpływu na Cq |

Rysunek 11.15.W przypadku malejących lub zahaczonych wykresów fluorescencji możliwą przyczyną może być konkurowanie nici komplementarnej ze starterem i/lub sondą o annealing do matrycy. Zignoruj, o ile nie ma to wpływu na Ct
Krzywe dysocjacji / topnienia
.Analiza dysocjacji lub krzywej topnienia jest przeprowadzana po qPCR i jest narzędziem analitycznym, które jest używane w połączeniu z barwnikami wiążącymi DNA (takimi jak SYBR Green I) lub nierozkładającymi się sondami, takimi jak Molecular Beacons lub Scorpions® Probes, w celu sprawdzenia, czy pojedynczy produkt został amplifikowany. Po amplifikacji PCR powstały amplikon jest inkubowany w rosnących temperaturach, a zmiany sygnału fluorescencyjnego są wykrywane, gdy DNA przechodzi ze stanu dwuniciowego do jednoniciowego. Gdy reakcja zawiera pojedynczy amplikon, topi się on równomiernie, a wykres dF/dT (szybkość zmiany fluorescencji w odniesieniu do temperatury) pokazuje pojedynczy pik. Badanie krzywej topnienia jest szczególnie skuteczne w połączeniu z danymi z kontroli. Rysunek 11.16A przedstawia profil topnienia po qPCR dla serii eksperymentalnych próbek testowych, kontroli pozytywnej i kontroli bez szablonu. Profil topnienia dla próbek testowych jest identyczny z kontrolą pozytywną i każda z nich wykazuje pojedynczy pik dla dF/dT. Profil topnienia dla kontroli bez szablonu ma szerszy profil i niższą Tm. Obserwacje te są zgodne z obecnością dimerów starterów widocznych w kontroli negatywnej. Zostało to potwierdzone przy użyciu żelu agarozowego barwionego bromkiem etydyny (rysunek 11.16B), który pokazuje również, że dimery starterów stają się widoczne, gdy matryca jest obecna w niskim stężeniu. Powoduje to zawyżone oszacowanie celu, gdy jest wykrywany w próbkach o niskim stężeniu docelowym. Dlatego test powinien zostać zoptymalizowany lub przeprojektowany. W przeciwieństwie do tego, Rysunek 11.16C pokazuje, że profil topnienia dla produktu w kontroli bez szablonu jest identyczny z profilem topnienia dla kontroli pozytywnej i próbki testowej. Jest to wyraźne wskazanie na zanieczyszczenie kontroli bez szablonu szablonem podczas konfiguracji eksperymentu. Ostatni przykład demonstruje rozpoznawanie amplifikacji celu z gDNA, który jest obecny w próbce cDNA (Rysunek 11.16D). Amplikon pochodzący z gDNA jest dłuższy, a zatem ma
wyższy Tm niż ten z cDNA.

Rysunek 11.16A.Kontrola pozytywna, reakcja testowa i NTC zostały poddane amplifikacji, a następnie analizie topnienia po PCR. W NTC widoczny jest produkt, który topi się w niższej temperaturze i z szerszym pikiem topnienia, co jest zgodne z tworzeniem dimeru startera.

Rysunek 11.16B.Dimery starterów są widoczne w rozdzielczości żelowej tych próbek (wraz z innymi), przy czym tworzenie dimerów starterów jest odwrotnie proporcjonalne do stężenia matrycy wejściowej.

Rysunek 11.16C.Przykład zastosowania analizy krzywej topnienia do identyfikacji zanieczyszczenia reakcji w NTC.
Rysunek 11.16D.Identyfikacja większego amplikonu powstałego w wyniku PCR gDNA.
| Problem | Wiele pików Tm peaks in melt profile data | |
|---|---|---|
| Single Product on a Gel (Figure 11.17) | Wiele produktów na żelu | |
| Możliwe przyczyny | Zlokalizowane regiony bogate w AT lub GC lub krótkie powtórzenia w PCR.br> lub krótkie powtórzenia w produkcie PCR powodujące niedoskonałą reanalizę amplikonu z pozornie różnymi profilami topnienia. | Startery są niespecyficzne i wytwarzają wiele produktów.
|
| Test diagnostyczny | Sprawdź sekwencję amplikonu pod kątem regionów lub powtórzeń bogatych w AT lub GC. | BLAST sekwencji starterów względem sekwencji organizmu źródłowego, aby zweryfikować pojedynczy cel. |
| Rozwiązania | Nie stanowi to problemu, jeśli analiza żelu pokazuje, że cały produkt jest specyficzny. Kontynuuj używanie starterów. |
|

Rysunek 11.17. A)Profil topnienia i B) analiza żelu agarozowego reakcji SYBR green I. Chociaż profil topnienia sugeruje produkty o różnym Tm, obraz żelu wskazuje, że obecny jest pojedynczy amplikon. Wskazuje to na sekwencję amplikonu, która zawiera regiony bogate w AT lub GC lub powtarzalny element powodujący nieregularne topnienie
Seryjne rozcieńczenie wzorca/krzywe standardowe
Niezależnie od tego, czy projekt eksperymentu obejmuje wymóg krzywej standardowej do ostatecznego oznaczenia ilościowego, wykrywanie seryjnego rozcieńczenia odpowiedniego wzorca jest skutecznym podejściem do walidacji testu i rozwiązywania problemów. Wykrycie seryjnego rozcieńczenia pozwala na zdefiniowanie eksperymentalnego liniowego zakresu dynamicznego testu. Rysunek 11.18A przedstawia krzywą standardową z punktami danych o niskim stężeniu, które nie pasują do profilu liniowego. Najczęstszą przyczyną tego wzorca danych jest to, że dimery starterów zostały utworzone w próbkach o niskim stężeniu (jak pokazano na Rys. 11.18B). Jest to krzywa standardowa wygenerowana na podstawie danych przedstawionych na Rysunku 11.2. Rysunek 11.18C przedstawia krzywą standardową z próbkami o wysokim stężeniu wypadającymi z zakresu liniowego. Najczęstszymi przyczynami takiego stanu rzeczy jest hamowanie reakcji przez szablon lub niewłaściwe ustawienia linii bazowej.
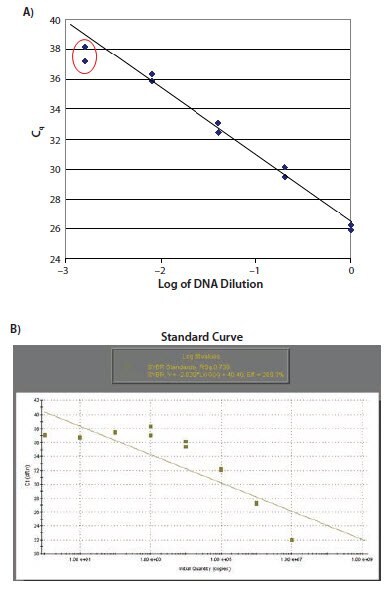
Rysunek 11.18. A) Punkty danych odnoszące się do niższych stężeń celu nie leżą na krzywej standardowej. B) Jest to typowe dla reakcji, w wyniku której powstają dimery starterów, jak pokazano na rysunku. W tym przypadku nie obserwuje się wzrostu Cq dla próbek o niskim stężeniu.

Rysunek 11.18C.Próbki o wysokim stężeniu matrycy nie leżą na krzywej standardowej. Jest to typowe dla reakcji, które są hamowane przez stężenie matrycy lub z powodu nieprawidłowego ustawienia linii bazowej.
Krzywa standardowa jest również używana do pomiaru wydajności reakcji w dynamicznym zakresie rozcieńczeń. Należy zachować ostrożność, aby upewnić się, że wszystkie punkty używane do obliczeń wydajności leżą na linii. Reakcje powinny być jak najbardziej zbliżone do 100% wydajności, a te z pozornie wysoką (110%) lub niską (85%) wydajnością powinny być dalej badane.

Rysunek 11.19A.Szablonowy kwas nukleinowy został rozcieńczony w 10-krotnej serii. Wykresy amplifikacji mają nienormalnie płytki gradient, a ΔCq wynosi 4 cykle zamiast oczekiwanych 3,3.

Rysunek 11.19B.Gradient krzywej standardowej wykresu Cq względem ilości jest wykorzystywany do obliczenia wydajności reakcji.
| Problem | Wydajność PCR jest większa niż 120% (Rysunek 11.20) |
|---|---|
| Możliwe przyczyny |
|
| Test diagnostyczny |
|
| Rozwiązania |
|
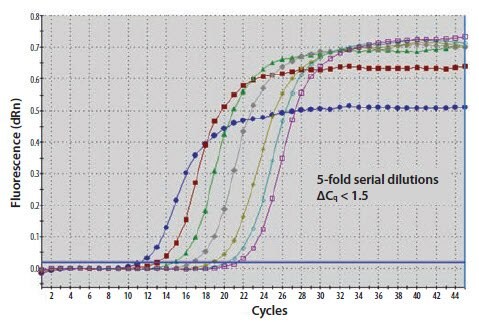
Rysunek 11.20A.Szablonowy kwas nukleinowy został rozcieńczony w 10-krotnej serii. ΔCq między działkami amplifikacji wynosi 1,5 cyklu zamiast 3,3

Rysunek 11.20B.Gradient krzywej standardowej wykresu Cq względem ilości jest używany do obliczenia wydajności reakcji, która jest bliska 140%.

Rysunek 11.20C.Badanie profilu krzywej topnienia ujawnia, że próbki o niższym stężeniu (żółte i niebieskie ślady) również zawierają sygnał z amplifikowanych dimerów starterów (pik przy niższej Tm).
Troubleshooting RT-PCR Case Studies
Nieudany test z sondą
Test oparty na sondzie został zaprojektowany do wykrywania EIFB1 w ludzkich próbkach cDNA, ale nie wykazuje amplifikacji. Początkowe reakcje przeprowadzono na urządzeniu ABi StepOne przy użyciu kompatybilnych odczynników. Podjęto próbę optymalizacji starterów przy użyciu zakresu stężeń od 200 nM do 900 nM (Rysunek 11.21), ale bez poprawy. Projekt testu został zweryfikowany i uznany za odpowiedni dla celu, a in silico przewidywano, że jest to test wysokiej jakości. Nowe startery zostały zsyntetyzowane i uruchomione wraz z podwielokrotnością oryginalnej syntezy przy użyciu innego operatora, odczynników SYBR Green I (a więc innych odczynników) i instrumentu (Eppendorf Realplex) (Rysunek 11.22). Przyjmując takie podejście, pamiętaliśmy, że głównym celem było rozwiązanie problemu, podczas gdy drugorzędnym celem było wyjaśnienie niepowodzenia. Reakcja ta dała równoważną amplifikację z obu partii starterów. Na tym etapie okazało się, że problem z reakcją leżał po stronie sondy, dlatego zsyntetyzowano nową sondę i obie partie zostały porównane przez drugiego operatora na urządzeniu Realplex przy użyciu odczynników LuminoCt® (innych odczynników niż te, które pierwotnie wypróbowano) (Rysunek 11.23). Obie sondy dały dane amplifikacji, przy czym nowa sonda okazała się nieco lepsza niż oryginalna, chociaż warto zauważyć, że oryginalna sonda została wysłana między laboratoriami testowymi i dlatego znajdowała się w temperaturze pokojowej, w roztworze przez kilka dni. Na tym etapie było jasne, że zarówno oryginalne, jak i zastępcze testy działały po uruchomieniu przez drugiego operatora w odczynnikach LuminoCt® na urządzeniu Realplex.
W związku z tym za pozostałe przyczyny pierwotnego niepowodzenia uznano:
- Operator: eksperyment został powtórzony kilka razy przez doświadczonego naukowca i dlatego uznano to za mało prawdopodobne wyjaśnienie.
- Instrument: może mieć pewne problemy, ponieważ niektóre inne testy zawiodły.
- Odczynniki: najłatwiejsze wyjaśnienie do przetestowania. Odczynniki LuminoCt® zostały porównane z istniejącymi odczynnikami przy użyciu obu partii oligo na instrumencie ABi StepOne przez pierwszego operatora. Reakcja nie powiodła się z oryginalnymi odczynnikami, ale dała dobrą amplifikację z odczynnikami LuminoCt® (Rysunek 11.24).

Rysunek 11.21.Primery do EIFB1 testowano w stężeniach od 200 nM do 900 nM. Nie zaobserwowano amplifikacji w żadnych warunkach (oligo i odczynniki ABi na ABi StepOne Plus).

Rysunek 11.22.Porównano dwie partie starterów do EIFB1 w odczynniku SYBR Green I; oryginalna wadliwa partia i nowa partia. (oligo i odczynniki na urządzeniu Eppendorf Realplex). Oba zestawy starterów wspierały amplifikację.

Rysunek 11.23.Porównano dwie partie starterów i sond do EIFB1 w odczynniku LuminoCt®; oryginalną wadliwą partię i nową partię. (oligosy i odczynniki na urządzeniu Eppendorf Realplex). Oba zestawy oligos wspierały amplifikację.
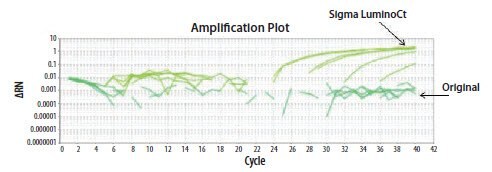
Rysunek 11.24.Test primera i sondy EIFB1 został przeprowadzony w dwóch różnych odczynnikach (Original ABi lub odczynnik LuminoCt®). Dane z tego testu uzyskano tylko wtedy, gdy przeprowadzono go przy użyciu odczynników LuminoCt®.
Wydajność reakcji była nieprawidłowa i zmienna
Test przeprowadzono na seryjnym rozcieńczeniu sztucznego oligo, przy użyciu standardowego testu primera i sondy. Test został pierwotnie opracowany i zoptymalizowany na innym urządzeniu, ale dziwny efekt rozcieńczenia nie był oczekiwany po przeniesieniu do innego laboratorium testowego i urządzenia (Rysunek 11.25A). Wszystkie warunki testu zostały ponownie zoptymalizowane, aby były specyficzne dla nowego laboratorium, ale bez zmiany danych. Operator zaobserwował, że efekt był bardziej wyraźny, gdy test został powtórzony w ciągu kilku godzin, przy użyciu tej samej serii rozcieńczeń. W ramach procesu rozwiązywania problemów test został przeprowadzony na innym urządzeniu przez innego operatora, który ponownie wygenerował oczekiwaną krzywą standardową. Doprowadziło to do sugestii, że początkowy problem był spowodowany: błędem operatora, awarią urządzenia lub subtelnymi zmianami w procedurze eksperymentalnej. Ponieważ obaj operatorzy są bardzo doświadczeni, a urządzenie działało dobrze w innych eksperymentach, zbadano opcję subtelnych różnic. Ważną wskazówką była obserwacja zmienności danych z tej samej serii rozcieńczeń po okresie przechowywania próbek w temperaturze 4 °C (Rysunki 11.25A i 11.25B). Doprowadziło to do zbadania probówek używanych do serii rozcieńczeń i przetestowania alternatyw. Po przejściu na probówki reakcyjne Eppendorf 1,5 ml do serii rozcieńczeń, wygenerowano przewidywaną krzywą standardową (Rysunek 11.25C), wykazując, że istotne jest, aby biologia molekularna, naczynia plastikowe o niskiej retencji były wybierane do PCR i że testy te są wrażliwe na subtelne różnice w protokole.

Rysunek 11.25A.Sztuczna matryca oligo została rozcieńczona 10-krotnie i wykryta przy użyciu testu opartego na specyficznej sondzie. Istnieją niespójne różnice między wykresami amplifikacji.

Rysunek 11.25B.Sztuczną matrycę oligo rozcieńczono 10-krotnie (są to rozcieńczenia wykryte na rysunku 11.25A) i pozostawiono w temperaturze 4 °C na kilka godzin przed wykryciem za pomocą testu opartego na specyficznej sondzie. Istnieją niespójne różnice między wykresami amplifikacji, które są pogarszane przez czas między rozcieńczeniem a testowaniem.

Rysunek 11.25C.Sztuczną matrycę oligo rozcieńczono 10-krotnie w probówkach klasy biologii molekularnej i wykryto za pomocą testu opartego na specyficznej sondzie. Zgodnie z oczekiwaniami istnieją spójne różnice między wykresami amplifikacji.
Podsumowanie - Lista kontrolna rozwiązywania problemów PCR
- Sprawdź jakość próbki (zdegradowany materiał spowoduje pogorszenie wyników). Lista kontrolna rozwiązywania problemów PCR
- Sprawdź jakość próbki (zdegradowany materiał spowoduje błędne wyniki).
- Sprawdź, czy protokół RT jest zgodny z projektem (np, Oligo-dT primed RT musi mieć test qPCR w 3' 1 kb sekwencji).
- Sprawdź projekt testu.
- Sprawdź wszystkie kontrole.
- Sprawdzenie starterów przy użyciu barwnika SYBR green I/wykonanie żelu.
- Sprawdzenie poprawności ustawień oprogramowania (linia bazowa, wykrywanie barwnika, stężenia wzorców).
- Upewnij się, że stężenie ROX jest odpowiednie dla urządzenia (i nie zakłóca multipleksu).
- Sprawdź poziomy fluorescencji tła.
- Sprawdź znakowanie sondy za pomocą testu DNase I lub powtórz syntezę sondy.
- Sprawdź jakość próbki (zdegradowany materiał spowoduje błędne wyniki).
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?