Analiza danych PCR/qPCR
A Technical Guide to PCR Technologies
- Analiza jakościowa PCR/qPCR
- Analiza danych qPCR
- Deriving Accurate Cq Values
- Setting the Threshold
- Strategie kwantyfikacji qPCR
- Standardowa kwantyfikacja krzywej
- Relative/Comparative Quantification
- Normalization
- Wybór genu referencyjnego
- Analiza stabilności genu referencyjnego
- Alternatywne metody normalizacji
- Analiza statystyczna i wizualizacja danych
- Techniki wizualizacji dla analizy jednoczynnikowej
- Testy statystyczne
- Hierarchical Clustering
- Principal Component Analysis
Analiza danych jakościowych PCR/qPCR
Po zakończeniu tradycyjnego PCR, dane są analizowane poprzez rozdzielczość w żelu agarozowym lub, ostatnio, w systemie elektroforezy kapilarnej. W przypadku niektórych zastosowań, qPCR zostanie uruchomiony z danymi punktu końcowego wykorzystanymi do analizy, takimi jak genotypowanie SNP. W każdym przypadku dane punktu końcowego zapewniają analizę jakościową po osiągnięciu przez PCR fazy plateau. W niektórych przypadkach może być możliwa analiza danych punktu końcowego w celu przeprowadzenia półilościowej analizy wydajności PCR, ale pomiary ilościowe są częściej wykonywane przy użyciu qPCR i analizy wartości cyklu kwantyfikacji (Cq)1 wartości.
Analiza danych qPCR
W niniejszym przewodniku podkreślono czynniki, które przyczyniają się do różnic w pomiarach kwasów nukleinowych przy użyciu PCR lub qPCR. Każdy z tych czynników powinien być zoptymalizowany w celu uzyskania testu, który zapewnia możliwie najbliższą wartość rzeczywistej ilości genu (celu) w reakcji. Wynikiem tych procesów jest wygenerowanie zestawu wartości Cq dla każdego celu w każdej próbce. Proces uzyskiwania i analizowania tych wartości Cq w celu zapewnienia wiarygodnych danych, które reprezentują historię biologiczną, został przedstawiony w tym rozdziale.
Określanie dokładnych wartości Cq Values
Podstawowa korekta
Wartość Cq jest określana dla każdego celu w każdej próbce. Różne pakiety analityczne, które są powiązane z różnymi instrumentami, mają alternatywne podejścia do określania Cq (a także używają alternatywnych nazw, np. Ct, Cp, punkt startu). Zagłębianie się w szczegóły wszystkich tych algorytmów wykracza poza zakres tego przewodnika. Jednak pomiary qPCR oparte na krzywych amplifikacji są wrażliwe na fluorescencję tła. Fluorescencja tła może być spowodowana szeregiem czynników, które obejmują wybór plastikowych naczyń, pozostałą fluorescencję sondy, która nie jest wygaszona, światło przeciekające do studzienki z próbką oraz różnice w detekcji optycznej dla danej studzienki płytki mikrotitracyjnej. W dobrze zaprojektowanych testach tło jest niskie w porównaniu do wzmocnionego sygnału. Jednak różnice w sygnale tła mogą utrudniać ilościowe porównanie różnych próbek. Dlatego ważne jest, aby skorygować zmiany fluorescencji tła, które powodują różnice w linii bazowej (Rysunek 10.1).

Rysunek 10.1Składniki wykresów amplifikacji. Ten wykres pokazuje wzrost fluorescencji wraz z liczbą cykli dla różnych próbek. Próg jest ustawiony powyżej granicy wykrywalności, ale znacznie poniżej fazy plateau, podczas której tempo amplifikacji spada.
Powszechnym podejściem jest wykorzystanie intensywności fluorescencji podczas wczesnych cykli, takich jak cykle od 5 do 15, w celu zidentyfikowania stałego i liniowego składnika fluorescencji tła. Jest ona następnie definiowana jako tło lub linia bazowa dla wykresu amplifikacji. Ze względu na efekty przejściowe zaleca się unikanie kilku pierwszych cykli (np. cykli od 1 do 5) w celu zdefiniowania linii bazowej, ponieważ często wykazują one artefakty stabilizujące reakcję. Im więcej cykli zostanie wykorzystanych do korekcji linii bazowej, tym lepsza będzie potencjalna dokładność składowej liniowej zmian linii bazowej. Wiele pakietów oprogramowania przyrządów umożliwia ręczne ustawienie cykli, które mają być brane pod uwagę przy definiowaniu linii bazowej. Funkcje te powinny być zbadane przez użytkownika, a pokusa zaakceptowania ustawień domyślnych powinna być zdecydowanie odrzucona.
Przykład efektu ustawienia linii bazowej pokazano na Rysunku 10.1. Jak widać, dokładne ustawienie linii bazowej ma wpływ na wartości Cq i pozorny kształt wykresu amplifikacji. W przykładzie linia bazowa dla krzywej oznaczonej jako C3 została nieprawidłowo wyregulowana ręcznie, tak aby cykle linii bazowej były obliczane na podstawie danych w cyklach od 5 do 31. Powoduje to, że krzywa spada do zerowego poziomu linii bazowej (Rysunek 10.2A) z wartością Cq równą 28,80. Aby to skorygować, surowe dane, R, są przeglądane i identyfikowany jest ostatni cykl liniowego tła (ostatni cykl przed amplifikacją). Na Rysunku 10.2B widać, że jest to cykl 22. Linia bazowa jest prawidłowo ustawiona na zero między cyklem 5 a cyklem 22 (Rysunek 10.2C), a wykres amplifikacji jest następnie korygowany (Rysunek 10.2D). Skorygowany współczynnik Cq wynosi 26,12. Należy zatem zauważyć, że wystąpiła znaczna różnica między wartościami Cq przy nieprawidłowych i prawidłowych ustawieniach linii bazowych, co pokazuje, że ustawienie prawidłowej linii bazowej jest ważnym elementem analizy danych.

Rysunek 10.2A-B. A)Typowy przykład spadku danych poniżej zera znormalizowanego odczytu fluorescencji, gdy ustawienie linii bazowej jest nieprawidłowe (niebieski wykres amplifikacji). B) Surowe dane z tych samych wykresów amplifikacji pokazujące granicę liniowej linii bazowej i że dane nie są błędne.

Rysunek 10.2C-D. C)Granice początku i końca linii bazowej są definiowane przy użyciu odpowiednich ustawień oprogramowania. D) Zastosowanie skorygowanych ustawień linii bazowej zapewnia dobrą jakość danych
Ustawianie progu
Chociaż niektórzy badacze zalecają mapowanie indywidualnego wykresu amplifikacji w celu oszacowania wydajności amplifikacji i ilości docelowych w mierzonych próbkach2,3,4.sup>2,3,4, oryginalnym i najbardziej powszechnym podejściem do wyznaczania Cq jest użycie progu. Szeroka akceptacja tego podejścia jest prawdopodobnie spowodowana tym, że metoda progowa jest prostą i skuteczną metodą kwantyfikacji.
Zasadą metody progowej jest to, że w celu wizualizacji powiązanego sygnału fluorescencyjnego z amplifikacji qPCR, sygnał musi wzrosnąć tak, aby był powyżej granicy wykrywalności instrumentu (a zatem linii bazowej; Rysunek 10.1). Liczba cykli wymaganych do osiągnięcia tego celu jest proporcjonalna do początkowej początkowej liczby kopii celu w próbce. W związku z tym potrzeba więcej cykli, aby sygnał wzrósł powyżej linii podstawowej, jeśli pierwotna liczba kopii jest niska, a mniej cykli, jeśli liczba kopii jest wysoka. Ponieważ linia bazowa jest ustawiona na granicy wykrywalności systemu, pomiary na linii bazowej byłyby bardzo niedokładne. Dlatego zamiast mierzyć intensywność minimalnej fluorescencji, którą system może wykryć, wybiera się wyższą fluorescencję i wprowadza sztuczny próg.
Wybór intensywności progu wymaga przestrzegania pewnych podstawowych zasad. Ważne jest, aby próg był ustawiony na stałą intensywność dla danego celu i dla wszystkich próbek, które mają być porównywane. Jeśli jest zbyt wiele próbek, aby zmieścić się na jednej płytce, należy przyjąć schemat kalibracji między płytkami, np. włączenie replikowanej kontroli, która służy jako kontrola między płytkami lub seryjne rozcieńczenie krzywej standardowej. Teoretycznie próg można ustawić w dowolnym miejscu na logarytmiczno-liniowej fazie krzywej amplifikacji. Jednak w praktyce logarytmiczno-liniowa faza amplifikacji może zostać zakłócona przez dryfowanie linii bazowej fluorescencji tła, fazę plateau lub różnice w wydajności testu, a tym samym gradient wykresu amplifikacji przy wyższych cyklach. Zaleca się ustawienie progu w następujący sposób:
- Wystarczająco powyżej linii bazowej fluorescencji tła, aby mieć pewność, że wykres amplifikacji nie przekroczy progu przedwcześnie z powodu fluorescencji tła.
- W fazie logarytmicznej wykresu amplifikacji, na który nie ma wpływu faza plateau (najłatwiej to zauważyć, przeglądając wykresy amplifikacji w widoku logarytmicznym, Rysunek 10.3A).
- W położeniu, w którym fazy logarytmiczne wszystkich wykresów amplifikacji są równoległe.
Proces ustawiania progu został przedstawiony na Rysunku 10.3. Na Rysunku 10.3A wykresy amplifikacji są wyświetlane w skali logarytmicznej osi Y, zapewniając w ten sposób wizualną ekspansję logarytmicznej fazy amplifikacji i przedstawiając ją jako liniową część wykresu amplifikacji. Próg jest ustawiany na najwyższą intensywność fluorescencji (patrz oś Y), która znajduje się w tej fazie logarytmicznej i gdzie wszystkie wykresy amplifikacji są równoległe. Skala jest następnie przywracana do widoku liniowego (Rysunek 10.3B), pokazując najwyższe ustawienie, które spełnia wymagania dotyczące ustawienia progu. Alternatywnie próg można ustawić na dolnym końcu tej fazy logarytmicznej (Rysunki 10.3C i 10.3D). Dopóki fazy logarytmiczne wykresów amplifikacji są równoległe, ustawienie progu nie ma wpływu na ΔCq między próbkami.

Rysunek 10.3Ustawienie progu wpływa na bezwzględną wartość Cq i może wpływać na ΔCq między próbkami. A). Używając wykresu logarytmiczno-liniowego danych, próg jest ustawiony na najwyższą intensywność fluorescencji, ale gdzie wykresy amplifikacji pokazują równoległe fazy logarytmiczne. B). Ustawienie progu jest utrzymywane od A) i jest wyświetlane na wykresie liniowym vs liniowym. C). Używając wykresu logarytmiczno-liniowego danych, próg jest ustawiany przy najniższej intensywności fluorescencji, ale tam, gdzie wykresy amplifikacji pokazują równoległe fazy logarytmiczne. D). Ustawienie progu jest utrzymywane od C) i jest wyświetlane na wykresie liniowym vs liniowym. W każdym przypadku wartości ΔCq między próbkami są takie same.
Wymóg ustawienia progu w pozycji, w której logarytmiczno-liniowe fazy wykresów amplifikacji są równoległe, staje się bardziej istotny, gdy dane przy wyższych cyklach są uwzględniane w analizie. Procedura ustawiania progu, która została opisana dla danych na Rysunku 10.3 została powtórzona na zestawie danych o wyższym Cq a wyniki przedstawiono na Rysunku 10.4. Wynikowe dane Cq w Tabeli 10.1 służą do zilustrowania zmienności wartości Cq, a co ważniejsze, wartości ΔCq dla trzech wykresów amplifikacji z trzema ustawieniami progu (Rysunek 10.4). Wartości ΔCq a zatem oszacowanie względnej ilości celu w każdej próbce są w dużym stopniu zależne od ustawienia progu (Rysunek 10.4), ponieważ wykresy amplifikacji nie są równoległe.

Rysunek 10.4.Analiza, która została przeprowadzona i zademonstrowana na rysunku 10.3, została powtórzona przy użyciu innego zestawu danych. W tym przypadku wykresy amplifikacji nie są równoległe ze względu na różnicę w wydajności reakcji przy wysokim Cq. Najniższe ustawienia dla A) i B) skutkują innymi wartościami ΔCq niż najwyższe ustawienia dla C) i D) (podsumowane w tabeli 10.1).
Strategie kwantyfikacji qPCR
Dokładne ustawienie linii bazowej i progu jest niezbędne dla wiarygodnej kwantyfikacji. Po ustawieniu każdego z nich generowana jest wartość Cq i jest ona wykorzystywana jako podstawa do kwantyfikacji. Ilość celu w danej próbce jest następnie określana przy użyciu krzywej standardowej lub kwantyfikacji względnej/porównawczej.
Kwantyfikacja krzywej standardowej
Jak sama nazwa wskazuje, kwantyfikacja krzywej standardowej wymaga użycia krzywej standardowej do określenia ilości celów w próbkach testowych. Wszystkie ilości określone dla próbek są zatem względne w stosunku do ilości przypisanej do krzywej standardowej. Wymaga to zastosowania dodatkowych, zewnętrznych wzorców wraz z każdym zestawem reakcji próbki. Wybór materiału dla krzywej standardowej jest ważny dla wyeliminowania potencjalnych różnic w kwantyfikacji wynikających z różnic między wydajnością testu w próbkach i standardach. Miejsca wiązania starterów zewnętrznych wzorców muszą być takie same jak te w celu, zawierać sekwencje, które są takie same jak cel, mieć podobną złożoność i być obsługiwane w możliwie podobny sposób. Dlatego podczas pomiaru stężenia celu w cDNA preferowany jest pomiar tego samego cDNA w seryjnym rozcieńczeniu próbki kontrolnej. Jednak w przypadku niektórych badań istnieją praktyczne powody, które to uniemożliwiają, dlatego ważne jest, aby jak najdokładniej odtworzyć warunki próbki, np. poprzez dodanie gDNA z gatunku niespokrewnionego z gatunkiem badanym, do sztucznego wzorca oligonukleotydowego lub linearyzowanego plazmidu niosącego standardową sekwencję. Po zidentyfikowaniu odpowiedniego konstruktu lub amplikonu generowana jest krzywa standardowa seryjnych rozcieńczeń. Współczynnik Cq dla celu jest określany dla każdego ze standardów i wykreślany względem stężenia lub względnego stężenia/współczynnika rozcieńczenia w skali logarytmicznej. Wynikiem jest krzywa standardowa, która jest następnie wykorzystywana do określenia stężeń próbek testowych poprzez porównanie wartości Cq uzyskanych z amplifikacji nieznanych próbek. W przypadku korzystania z krzywej standardowej do kwantyfikacji, ustawienie progu musi być utrzymywane na stałym poziomie w celu określenia Cq dla standardu i próbek na tej samej płytce. Próg może się różnić między płytkami.
Kwantyfikacja względna/porównawcza
Kwantyfikacja względna lub porównawcza wykorzystuje różnicę w Cq jako wyznacznik różnic w stężeniu sekwencji docelowej w różnych próbkach. Zamiast mierzyć ilości celu na próbkę, jak w przypadku metody krzywej standardowej, prowadzi to do zestawów danych pokazujących zmiany krotności między próbkami.
W oryginalnej formie tego podejścia5, wydajność wszystkich testów została przyjęta jako 100%, co prowadzi do założenia, że Cq różnica 1 (ΔCq = 1) była wynikiem 2-krotnej różnicy w celu. Aby określić krotną zmianę w celu lub genie zainteresowania (GOI), dane muszą być również odniesione do kontroli obciążenia (gen referencyjny, ref; patrz poniżej w celu omówienia normalizacji danych).

Rysunek 10.5.Konstrukcja krzywej standardowej. Cq zarejestrowane dla każdej próbki z serii rozcieńczeń jest wykreślane w logarytmicznej skali liniowej względem stężenia względnego.
W równaniu 1 stosunek GOI, po korekcie do genu ref, w 2 próbkach (A względem B) jest mierzony jako: 2 (przy założeniu 100% wydajności reakcji) podniesione do potęgi różnic w wartościach Cq dla GOI podzielone przez 2 podniesione do potęgi różnic w wartościach Cq dla genu ref

Równanie 1.Oryginalny (Livak) model kwantyfikacji względnej.
Jednak, jak zilustrowano w Assay Optimization and Validation, wydajności reakcji znacznie się różnią, co może mieć duży wpływ na dane. Dlatego założenia w Wzorze 1 zostały uwzględnione (Wzór 2)6, aby różnice w wydajności reakcji mogły zostać włączone do analiz. W tym przypadku współczynnik amplifikacji 2 jest zastępowany rzeczywistą wydajnością PCR (określoną przez analizę krzywej standardowej; patrz Assay Optimization and Validation).

Równanie 2.Względny model kwantyfikacji dostosowany do wydajności (Pfaffl)
Jako przykład zastosowania modelu kwantyfikacji względnej dostosowanego do wydajności (Wzór 2), zestaw wartości Cq przedstawiono w Tabeli 10.2. Wydajność dla GOI wynosi 1,8, a dla genu ref 1,94.
Jest to bardzo prosty przykład badania z wymogiem pomiaru krotnej różnicy między jednym genem w dwóch próbkach i po normalizacji do pojedynczego genu referencyjnego. Stosunek pokazuje krotną zmianę GOI w próbce 2 w stosunku do próbki 1, po korekcie do pojedynczego genu Ref. Stało się jednak oczywiste, że wybór pojedynczego, odpowiedniego genu referencyjnego jest często niemożliwy i dlatego zaproponowano bardziej wyrafinowane podejścia do normalizacji.
Normalizacja
Głównym celem większości eksperymentów opartych na PCR jest odpowiedź na podstawowe pytanie, czy cel jest obecny w próbce (nieznany, UNK). Na najprostszym poziomie odpowiedź na to pytanie uzyskuje się poprzez wykonanie żelu i zbadanie fragmentów pod kątem obecności lub braku pożądanego GOI. Gdy fragment jest obecny, potwierdzenie wielkości fragmentu daje pewność pozytywnego wyniku. Jednak w przypadku nieobecności istnieje możliwość uzyskania wyniku fałszywie ujemnego. Dlatego krytyczne jest powtórzenie testu testowego, a także wykonanie co najmniej jednego dodatkowego PCR, aby służyć jako ładunek i pozytywna kontrola PCR. Uniwersalny test kontroli inhibicji, SPUD (patrz Sample Purification and Quality Assessment), może być użyty w celu zwiększenia pewności co do wyniku negatywnego. Alternatywnym podejściem jest przeprowadzenie testu, który jest specyficzny dla genu lub genów referencyjnych. Tradycyjnie, testy PCR wykrywające geny referencyjne, GAPDH, 18S rybosomalny RNA lub β-aktynę były przeprowadzane wraz z tymi dla GOI, a powstałe fragmenty wizualizowane na żelu. GAPDH, 18S rybosomalny RNA i β aktyna ulegają konstytutywnej ekspresji i dlatego zostały użyte jako kontrole obciążenia w analizach półilościowych. Wkrótce jednak okazało się, że geny te nie ulegają wszechobecnej ekspresji w tym samym stężeniu we wszystkich komórkach, niezależnie od projektu eksperymentalnego. Dlatego też pojawiła się potrzeba stabilnego odniesienia, gdy celem był pomiar względnych stężeń kwasów nukleinowych, zwykle cDNA, ale także gDNA, gdy na przykład badano zmienność liczby kopii genu.
Normalizacja to proces korygowania pomiarów technicznych do stabilnego odniesienia w celu zbadania prawdziwej zmienności biologicznej. Istnieje wiele metod normalizacji różnic technicznych, co oznacza, że należy wybrać i zatwierdzić odpowiednie podejście do konkretnego eksperymentu7. Należy pamiętać, że zastosowanie niewłaściwych technik normalizacji może być bardziej szkodliwe dla całego procesu analitycznego niż brak normalizacji8.
Wpływ jakości próbki na normalizację testu
Wpływ integralności i czystości próbki na pomiary ilości docelowej za pomocą qPCR i RT-qPCR został szczegółowo omówiony (Oczyszczanie próbek i ocena jakości, Kontrola jakości próbek i Odwrotna transkrypcja, Odwrotna transkrypcja). Wykazano, że inhibitory w próbce i degradacja RNA mają zróżnicowany wpływ na pomiar danego celu9. Inhibitory wpływają na pomiar każdego celu, ale w różnym stopniu, w zależności od projektu testu. Degradacja całkowitego RNA wpływa na pomiar mRNA i miRNA10, ponownie w dużym stopniu zależąc od ogólnego projektu eksperymentu. Dlatego też kluczowe znaczenie ma uwzględnienie wpływu stężenia matrycy na reakcję RT oraz wpływu jakości próbki na dane po normalizacji. Normalizacja nie przeciwdziała efektowi niskiej jakości testów lub próbek (patrz Optymalizacja i walidacja testów).
Podejścia do normalizacji
Metody normalizacji przeciwdziałają zmienności, która może zostać wprowadzona podczas wieloetapowego procesu wymaganego do przeprowadzenia analizy qPCR (Rysunek 10.6). Jednak zastosowanie normalizacji na dowolnym etapie procesu może nie kontrolować błędu technicznego i/lub stronniczości, które zostały lub zostaną wprowadzone odpowiednio na wcześniejszym lub późniejszym etapie. Metody normalizacji nie wykluczają się wzajemnie, dlatego zaleca się stosowanie kombinacji kontroli11.

Rysunek 10.6.qPCR jest procesem wieloetapowym i każdy etap musi być kontrolowany. Normalizacja musi być uwzględniona w serii kontroli.
Celem normalizacji jest zapewnienie stabilnego punktu odniesienia, do którego można odnieść pomiary; dlatego wybór współczynnika normalizacji musi być pomiarem, który jest stabilny przez cały czas trwania eksperymentu. Może to być stabilny gen referencyjny lub jedna z alternatyw, takich jak liczba komórek, masa tkanki, stężenie RNA/DNA, zewnętrzny pik12 lub reprezentatywna miara globalnie wyrażonych genów.
Wybór genów referencyjnych
Geny referencyjne to cele, których ilość nie zmienia się w wyniku eksperymentu. Podczas ilościowego określania zmienności liczby kopii DNA, w której liczba kopii sekwencji będącej przedmiotem zainteresowania może ulec zmianie, pomiar jest po prostu normalizowany poprzez celowanie w alternatywny region genomu, o którym wiadomo, że się nie zmieni. Przykładem zastosowania tej metody jest pomiar amplifikacji genomu ludzkiego receptora naskórkowego czynnika wzrostu 2 (HER-2)13. Niestabilność genomowa HER-2 jest wskaźnikiem prognostycznym w raku piersi, a dokładny pomiar statusu amplifikacji HER-2 jest ważny w leczeniu pacjentów. Status HER-2 można zmierzyć za pomocą qPCR, porównując kopie HER-2 z innym celem genomowym, który działa jako kontrola.
Przy pomiarze ekspresji genów, geny referencyjne są celami o stężeniach mRNA, które nie zmieniają się w wyniku eksperymentu. Przykładem może być badanie, w którym mierzony jest wpływ na ekspresję genu X po dodaniu związku mitogennego do monowarstwy komórek. Aby zmierzyć zmianę w genie X, wymagany jest punkt odniesienia. Dlatego też mierzony jest również inny gen (lub geny), o których wiadomo, że dany mitogen nie ma na nie wpływu. Daje to badaczowi natychmiastowe wyzwanie znalezienia celu mRNA, na który nie ma wpływu procedura eksperymentalna, zanim będzie w stanie zbadać GOI. Ten proces walidacji genów referencyjnych ma fundamentalne znaczenie dla dokładnego pomiaru GOI. Najczęściej stosowanym podejściem do normalizacji jest zignorowanie tego procesu i normalizacja danych ekspresji genów do pojedynczego, niezwalidowanego genu referencyjnego. Praktyka ta nie jest zalecana i stoi w bezpośredniej sprzeczności z wytycznymi MIQE1. Ilościowe oznaczanie mRNA metodą RT-qPCR jest rutynowo zagrożone przez nieprawidłowy wybór genów referencyjnych. Niedopuszczalne jest stosowanie stosunkowo powszechnych praktyk stosowania genu referencyjnego, ponieważ startery są już w zamrażarce, był on używany w przeszłości na Northern blot, jest używany przez kolegę lub używany w innym laboratorium do innego eksperymentu. Geny referencyjne muszą być walidowane w określonych scenariuszach eksperymentalnych, aby mieć pewność, że eksperyment nie ma wpływu na dany gen referencyjny. Jeśli ta walidacja nie zostanie przeprowadzona, a eksperyment będzie miał wpływ na gen referencyjny, wyniki mogą być nieprawidłowe, a późniejsze interpretacje mogą skutkować bezsensownymi danymi8.
Istnieje szereg literatury naukowej opisującej różne metody normalizacji7-14 jak również mnóstwo publikacji opisujących protokoły wymagane do identyfikacji najbardziej odpowiednich genów normalizujących dla danego scenariusza eksperymentalnego. Podczas gdy w przeszłości kluczowym pytaniem było, czy wybrać pojedyncze czy wiele genów referencyjnych, niższe koszty bieżące oznaczają, że obecne najlepsze praktyki przesunęły się w kierunku pomiaru wielu genów referencyjnych.
Wybór stabilnych genów referencyjnych wymaga od analityka oceny stabilności qPCR dla pewnej liczby (zwykle 10 do 20 genów) kandydatów na docelowe mRNA7 na podzbiorze próbek, które reprezentują testowe i kontrolne mRNA. Pełny protokół znajduje się w Załącznik A, Protokoły, niniejszego przewodnika i może być stosowany w połączeniu z różnymi metodami analitycznymi przy użyciu programów takich jak REST15, GeNorm14, Bestkeeper16 lub NormFinder17. Procedura ta została opisana bardziej szczegółowo w poniższej sekcji, Analiza stabilności genu referencyjnego.
Analiza stabilności genu referencyjnego
Gen referencyjny jest dosłownie punktem zwrotnym dla testów kwantyfikacji względnej qPCR. Dlatego też dla niezawodności całego testu krytyczne znaczenie ma stabilność genu referencyjnego. Jeśli ekspresja genu referencyjnego zmienia się między próbkami, zmienność zostanie bezpośrednio przeniesiona na wyniki kwantyfikacji, a dodatkowa zmienność może zaciemnić pożądany obserwowalny efekt biologiczny lub, co gorsza, może stworzyć całkowicie sztuczny wygląd efektu biologicznego, który nie jest związany z rzeczywistym genem będącym przedmiotem zainteresowania. Z tych powodów zdecydowanie zaleca się przestrzeganie kilku środków bezpieczeństwa, aby uczynić zmienność genów referencyjnych nieistotną i uczynić pomiary efektów biologicznych tak istotnymi, jak to tylko możliwe.
Prawdopodobnie najważniejszym środkiem bezpieczeństwa jest użycie nie tylko jednego, ale dwóch lub więcej genów referencyjnych. Ekspresję kilku genów referencyjnych można uśrednić w celu zmniejszenia zmienności technicznej spowodowanej normalizacją. Może to być przydatne do poprawy istotności w pomiarach małych efektów biologicznych. Jednak, co ważniejsze, dwa lub więcej genów referencyjnych zapewnia wzajemną kontrolę utrzymywanej stabilności i kontrolę nieoczekiwanych zdarzeń, które mogą wpływać na poziomy ekspresji jednego z genów referencyjnych. W przypadku pojedynczego genu referencyjnego istnieje ryzyko, że nieoczekiwany wpływ ekspresji genu może zostać niewykryty w teście.
Innym środkiem bezpieczeństwa jest użycie więcej niż jednej metody identyfikacji stabilnych genów referencyjnych. Poniżej przedstawiono przykład ilustrujący kilka aspektów normalizacji genów referencyjnych, w tym możliwą korzyść z zastosowania metod geNorm i NormFinder na tym samym zestawie danych.
Tabela 10.3 zawiera listę kandydatów na geny referencyjne, które zostały ocenione podczas warsztatów, które wcześniej przeprowadziliśmy z EMBL. Próbki zostały pobrane z hodowli ludzkich komórek w dwóch różnych grupach leczenia. Ten zestaw danych zostanie wykorzystany do zademonstrowania aspektów walidacji genów referencyjnych.
Oba algorytmy NormFinder i geNorm zostały opracowane przy założeniu, że testowanie wielu kandydatów na geny referencyjne może być wykorzystane do uszeregowania stabilności poszczególnych kandydatów na geny referencyjne. Założenie to może być prawdziwe, jeśli na przykład wszyscy kandydaci na geny referencyjne zmieniają się stochastycznie wokół stabilnych poziomów ekspresji. Jednak niekoniecznie musi to być prawdą w rzeczywistości. Aby uniknąć mylących wyników, rozsądnie jest unikać regulowanych, a zwłaszcza współregulowanych kandydatów na geny referencyjne.
Lista kandydatów na geny referencyjne pokazana w Tabeli 10.3 została specjalnie wybrana, aby wybrać geny należące do różnych klas funkcjonalnych, zmniejszając prawdopodobieństwo, że geny mogą być współregulowane. Godnym uwagi wyjątkiem jest GAPDH, który występuje tutaj w dwóch wersjach. Chociaż nie ma to wpływu na tę analizę, najlepszą praktyką jest unikanie wielokrotnych wpisów genów, które mogą być podejrzewane o współregulację.
Pierwszym algorytmem, który zostanie zademonstrowany, jest geNorm. Zapewnia on ocenę stabilności genów poprzez obliczenie miary stabilności genów zwanej wartością M, która opiera się na porównaniach parami między analizowanym kandydatem na gen referencyjny a wszystkimi innymi kandydatami na gen referencyjny w zestawie danych. Jest to wykonywane w sposób iteracyjny, co oznacza, że w tym przykładzie procedura jest najpierw wykonywana na wszystkich 15 kandydatach na geny referencyjne, najmniej stabilny jest usuwany, proces jest powtarzany na pozostałych 14, drugi najmniej stabilny kandydat jest usuwany i tak dalej, aż pozostaną dwa geny referencyjne.
Mogą wystąpić sytuacje, w których identyfikacja najbardziej stabilnego genu referencyjnego może być szczególnie trudna. Jednym z przypadków może być sytuacja, w której wszyscy kandydaci na geny referencyjne osiągają słabe wyniki. Innym przypadkiem może być sytuacja, w której wszyscy kandydaci na geny referencyjne osiągają dobre wyniki. Aby rozróżnić te dwa przypadki, użyteczną wskazówką jest to, że geny referencyjne o wartości M poniżej 0,5 można uznać za stabilnie wyrażone.
Drugim algorytmem, który zostanie zademonstrowany, jest NormFinder, który jest bezpłatnie dostępnym pakietem do analizy genów referencyjnych (Dodatek B, Dodatkowe zasoby). Podstawowy algorytm przyjmuje podejście podobne do ANOVA do oceny stabilności genu referencyjnego, w którym całość i podgrupy są analizowane pod kątem zmian. Jedną z zalet jest to, że uzyskane miary są bezpośrednio związane z poziomami ekspresji genów. Odchylenie standardowe 0,20 w jednostkach Cq odpowiada zatem około 15% zmienności poziomów ekspresji liczby kopii danego kandydata na gen referencyjny.
Dla wygody, w tej demonstracji oba te pakiety analityczne są dostępne przy użyciu oprogramowania do analizy danych GenEx (MultiD), ale są one również dostępne jako niezależne pakiety (Dodatek B, Dodatkowe zasoby).
Wykresy słupkowe pokazane na Rysunku 10.7 zilustrują geny referencyjne uszeregowane zgodnie z ich odpowiednimi miarami stabilności przy użyciu obu algorytmów. Ponadto wykres przedstawiający skumulowane odchylenie standardowe z NormFinder wskazuje, że połączenie maksymalnie trzech najlepszych genów referencyjnych może przynieść poprawę stabilności.

Rysunek 10.7.Wykresy słupkowe przedstawiające miary stabilności: Wartości M dla geNorm i odchylenia standardowe dla NormFinder. Ponadto wykres przedstawiający skumulowane odchylenie standardowe z NormFinder wskazuje, że połączenie maksymalnie trzech najlepszych genów referencyjnych może przynieść poprawę stabilności. Zestaw danych został wygenerowany na podstawie testów zaprojektowanych dla kandydatów na geny referencyjne przedstawionych w tabeli 10.3 i zmierzonych na ludzkiej hodowli komórkowej w dwóch różnych grupach leczenia. Należy zauważyć, że w tym przypadku algorytmy stabilności genów referencyjnych geNorm i NormFinder nie zgadzają się co do najlepszych genów referencyjnych.

Rysunek 10.8.Średni wyśrodkowany profil ekspresji kandydatów na geny referencyjne dwóch próbek w każdej grupie leczenia. Próbki 1 i 2 należą do pierwszej grupy leczenia, a próbki 3 i 4 należą do drugiej grupy leczenia. Profile ekspresji SDHA i CANX zaznaczono na czerwono. Profil ekspresji UBC zaznaczono na żółto. Tabela zawiera zmierzone wartości Cq w zestawie danych.
Z powodu rozbieżnych profili ekspresji możliwe jest, że SDHA i CANX są regulowane przez różne alternatywy leczenia, a zatem nie są odpowiednie jako geny referencyjne. Usunięcie ich z zestawu danych i powtórzenie analizy skutkuje zgodnością obu algorytmów oraz tym, że najlepszym wyborem genów referencyjnych są EIF4A2 i ATP53 (Rysunek 10.9). W obliczeniach skumulowanych odchyleń standardowych NormFinder widać również, że dodanie większej liczby genów referencyjnych nie poprawia stabilności.

Rysunek 10.9.Inspekcja profili ekspresji i zmierzonych wartości Cq (rysunek 10.8) wzbudziła obawy, że SDHA i CANX mogą być współregulowane w zastosowanym teście. Współregulacja może zakłócać algorytmy stabilności genów referencyjnych. Wykresy słupkowe przedstawiające miary stabilności: A) wartości M dla geNorm i B) odchylenia standardowe dla NormFinder. Zestaw danych jest taki sam jak ten użyty na rysunku 10.8, z wyjątkiem tego, że dane dla SDHA i CANX zostały usunięte. Zauważ, że przy tym zredukowanym zestawie danych algorytmy stabilności genów referencyjnych geNorm i NormFinder zgadzają się co do najlepszych genów referencyjnych.
Analiza danych w tym przykładzie służy zilustrowaniu, że równoległe stosowanie geNorm i NormFinder pozwala na identyfikację kandydatów na współregulowane geny referencyjne, a usunięcie tych genów z dalszych badań zapewnia ostateczną identyfikację genów referencyjnych, które można przyjąć z większą pewnością niż po zastosowaniu pojedynczej analizy. Identyfikacja i wybór stabilnych genów referencyjnych prowadzi do większego bezpieczeństwa analizy danych.
Alternatywne metody normalizacji
Podczas gdy normalizacja do genów referencyjnych jest najbardziej powszechną metodą normalizacji testów, istnieją sytuacje, w których podejście to nie jest odpowiednie, na przykład gdy porównywana jest duża liczba genów w heterogenicznej grupie próbek lub podczas profilowania miRNA. W takich sytuacjach konieczne jest przyjęcie alternatywnej strategii.
Normalizacja do masy tkanki lub liczby komórek
Pomiar liczby komórek lub masy tkanki do wykorzystania jako współczynnik normalizacji nie jest tak prosty, jak mogłoby się wydawać. Eksperymenty z hodowlą komórkową są stosunkowo łatwe do normalizacji w oparciu o liczbę komórek. Jednak dodanie leczenia może wpłynąć na morfologię komórek, komplikując stosunek liczby komórek do całkowitego RNA/wyrażonych genów w porównaniu z kulturą kontrolną. Leczenie eksperymentalne może skutkować wytworzeniem dodatkowej macierzy komórkowej, powodując różnice w wydajności ekstrakcji kwasów nukleinowych.
Tkanki biologiczne mogą być wysoce heterogeniczne w obrębie i między podmiotami, przy czym większa zmienność jest widoczna, gdy zdrowa tkanka jest porównywana z chorą tkanką. Nawet pozornie mniej złożone tkanki, takie jak krew, mogą znacznie różnić się liczbą komórek i składem, tak że ekspresja genów różni się znacznie między pozornie zdrowymi dawcami18.
Wszelkie opóźnienia w procesach stosowanych do oczyszczania kwasu nukleinowego spowodują zmiany w mierzonym RNA. Na przykład opóźnienia w przetwarzaniu komórek jednojądrzastych krwi obwodowej i ekstrakcji RNA z komórek powodują znaczne zmiany w ekspresji genów19. Metody leżące u podstaw procedur ekstrakcji są również głównymi źródłami zmienności technicznej. Nawet proces izolacji wybrany do pobierania próbek komórek krwiopochodnych i oczyszczania RNA powoduje różnice w pozornych profilach ekspresji genów20. Dlatego pierwszą kwestią normalizacji jest zapewnienie, że pobieranie i przetwarzanie jest absolutnie identyczne dla wszystkich próbek. Następnie krytyczne jest przeprowadzenie wystarczającej kontroli jakości, aby mieć pewność co do stężenia, integralności i czystości próbki (Oczyszczanie próbek i ocena jakości i powiązane protokoły w Appendix A).
Normalizacja do stężenia RNA
Jako minimum, ważne jest oszacowanie stężenia matrycy (DNA dla qPCR lub RNA dla RT-qPCR) i, jak wspomniano w Oczyszczanie próbek i ocena jakości, kluczowe znaczenie ma zapewnienie, że ten sam instrument jest używany do wszystkich pomiarów, ponieważ określenie stężenia kwasu nukleinowego jest również zmienne i zależne od techniki.
Podczas pomiaru całkowitego stężenia RNA zdecydowana większość próbki składa się z rRNA, a tylko niewielka część składa się z mRNA będącego przedmiotem zainteresowania podczas badania ekspresji genów lub sncRNA podczas badania regulacji ekspresji genów. Oznacza to, że jeśli stężenie rRNA wzrośnie o niewielką ilość, ale mRNA pozostanie na stałym poziomie, całkowite stężenie RNA wzrośnie. Stężenie mRNA musi wzrosnąć znacznie, aby spowodować pozorny wzrost całkowitego stężenia RNA. Dlatego stężenie rRNA jest niewiarygodną miarą stężenia mRNA, ale w przypadku wielu protokołów wymagane jest równe stężenie RNA, aby zapewnić dokładną odwrotną transkrypcję (patrz odwrotna transkrypcja).
Normalizacja do globalnej ekspresji genów
Przy pomiarze dużej liczby celów, analityk może oszacować globalną średnią całkowitej ekspresji genów i zidentyfikować regulowane sekwencje RNA, które odbiegają od tej średniej. Podejście to jest konwencjonalnie stosowane do normalizacji macierzy ekspresji genów. Jest to cenna alternatywa dla stosowania genów referencyjnych i może być preferowana tam, gdzie mierzonych jest wiele celów.
Innym niedawno zbadanym podejściem jest pomiar endogennie wyrażanych elementów powtarzalnych (ERE), które są obecne w wielu mRNA. Wiele gatunków zawiera te powtarzające się elementy (ALU u naczelnych, elementy B u myszy), które mogą zapewnić oszacowanie frakcji mRNA. Wykazano, że pomiar tych sekwencji docelowych działa jak konwencjonalne systemy normalizujące9 (Le Bert, et al., w przygotowaniu) i może stanowić uniwersalne rozwiązanie lub alternatywę dla złożonych eksperymentów, w których stabilne kombinacje genów referencyjnych są niedostępne.
Normalizacja danych miRNA
Jak dotąd nie ma doniesień o uniwersalnym genie referencyjnym miRNA. Dlatego wybór systemu normalizacji jest nadal raczej empiryczny. Jeśli to możliwe, stabilne niezmienne miRNA można zidentyfikować na podstawie podejść obejmujących cały genom, tj. mikromacierzy. Małe nukleolarne RNA (snoRNA) zostały również wykorzystane jako geny referencyjne. Globalna ekspresja genów jest również użyteczną metodą normalizacji ekspresji miRNA, gdy nieznane jest stabilne odniesienie i przeanalizowano kilkaset celów21,22,23. Metoda ta jest bardziej odpowiednia dla tych, którzy stosują podejścia skutkujące wychwytywaniem wszystkich miRNA jako cDNA w postaci multipleksowanej, np, Systemy Exiqon i miQPCR (patrz Castoldi i wsp. w PCR Technologies, Current Innovations24).
Powtórzenia biologiczne i techniczne
Celem normalizacji jest uniknięcie błędów systematycznych i zmniejszenie zmienności danych do ewentualnej analizy statystycznej. Innym ważnym aspektem przygotowywania danych do analizy statystycznej jest wykorzystanie replikatów danych.
Replikaty biologiczne są absolutnie niezbędne do analizy statystycznej. Poziomy istotności statystycznej są często ustalane na poziomie 5%. W przypadku efektów biologicznych zbliżonych do takiego poziomu istotności może być konieczne posiadanie co najmniej 20 powtórzeń biologicznych w celu określenia poziomu istotności testów (1:20 odpowiadające 5%). W rzeczywistości sugeruje się, że do dokładnego oszacowania istotności wymagana jest co najmniej 50-krotnie większa liczba obserwacji25, tj. rzędu tysiąca próbek biologicznych. Oczywiście ograniczenia praktyczne rzadko pozwalają na replikacje biologiczne na tych poziomach. Co więcej, dokładne szacunki liczby niezbędnych powtórzeń biologicznych w celu osiągnięcia danego poziomu istotności zależą również od poziomu zmienności danych. Niemniej jednak ważne jest, aby zdać sobie sprawę, że częstym błędem jest niedoszacowanie niezbędnej liczby powtórzeń biologicznych, aby móc wyciągnąć wiarygodne wnioski. Zaleca się przeprowadzenie wstępnego badania pilotażowego w celu oceny nieodłącznej zmienności testu i potencjalnej wielkości obserwowalnego efektu biologicznego, aby mieć dobrą podstawę do oszacowania niezbędnej liczby powtórzeń biologicznych26.
Powtórzenia techniczne nie są wykorzystywane bezpośrednio do analizy statystycznej. Zamiast tego, repliki techniczne są wykorzystywane do tworzenia kopii zapasowych próbek (na wypadek, gdyby niektóre próbki zostały utracone w procesie obsługi technicznej) oraz do poprawy oceny dokładności danych. Repliki techniczne mogą poprawić dokładność danych, jeśli założenie jest prawdziwe, że różnią się one stochastycznie wokół dokładnego pomiaru na każdym etapie procesu obsługi technicznej. Średnia replik technicznych jest bliższa dokładnemu pomiarowi. Efekt uśredniania powtórzeń technicznych można zilustrować, zwracając uwagę na rozmiar przedziału ufności w symulowanym zestawie danych z wcześniej określoną zmiennością, tj. odchyleniem standardowym ustawionym na jeden. Jak widać w Tabeli 10.4, przedział ufności staje się mniejszy wraz ze wzrostem liczby powtórzeń technicznych (próbek), co wskazuje na bardziej precyzyjne oszacowanie dokładnego pomiaru. Co więcej, zwężenie przedziału ufności jest najbardziej dramatyczne przy niskiej liczbie powtórzeń technicznych. Zwiększenie liczby replikacji z 2-3 zmniejsza przedział ufności z 8,99 do 2,48, co oznacza ponad 3-krotną poprawę precyzji oszacowania dokładnego pomiaru. Podczas gdy dodatkowe repliki nadal poprawiają oszacowanie dokładności pomiaru, efekt jest coraz mniejszy. Dlatego oczywiste jest, że w przypadkach, w których zmienność obsługi technicznej jest kwestią, może być wielką zaletą stosowanie potrójnych powtórzeń zamiast duplikatów.
Powtórzenia techniczne mogą być zbierane na kilku etapach procesu obsługi próbki, w tym ekstrakcji RNA, odwrotnej transkrypcji i wykrywania qPCR. Jeśli repliki techniczne są wykrywane na kilku etapach, generowany jest zagnieżdżony projekt eksperymentalny. Badanie pilotażowe, które wykorzystuje zagnieżdżony projekt eksperymentalny, może pomóc w identyfikacji etapów obsługi próbek, które w największym stopniu przyczyniają się do technicznych błędów obsługi, a optymalny plan pobierania próbek można obliczyć na podstawie tych informacji27.
Analiza statystyczna i wizualizacja danych
Naukowa analiza danych biologicznych koncentruje się na formułowaniu i testowaniu hipotez. Sformułowanie hipotezy wymaga szczegółowego zrozumienia warunków i zmiennych testu. Pomyślne przetestowanie hipotezy wymaga starannego wykonania i odpowiedniego projektu eksperymentalnego, aby zmaksymalizować pożądany obserwowalny sygnał przy jednoczesnym zminimalizowaniu zmienności technicznej. W tym kontekście przydatne jest rozróżnienie między badaniami eksploracyjnymi i potwierdzającymi (Rysunek 10.10).
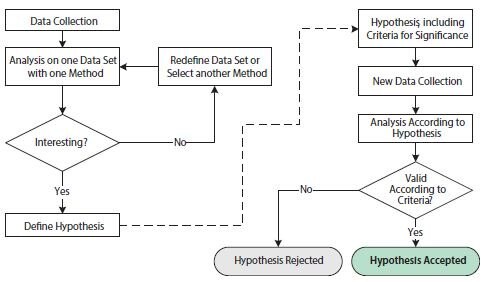
Rysunek 10.10.Schemat blokowy ilustrujący operacje związane z eksploracyjnymi i potwierdzającymi analizami statystycznymi. Lewa strona rysunku, przed przerywaną strzałką, przedstawia operacje w eksploracyjnym badaniu statystycznym. Prawa strona rysunku, po przerywanej strzałce, przedstawia operacje w potwierdzającym badaniu statystycznym.
Celem badania eksploracyjnego jest analiza danych za pomocą jednej lub kilku różnych technik w celu uzasadnienia hipotezy. Zbiór danych może zostać przedefiniowany i/lub różne techniki analizy mogą być stosowane wielokrotnie w celu wsparcia jednej lub kilku hipotez. Badanie eksploracyjne jest zatem bardzo elastyczne w stosunku do specyfiki każdego pytania naukowego. Jednak wielokrotne testowanie hipotez na jednym zestawie danych może prowadzić do kwestii, które podważają wnioski statystyczne. Wynika to z wielokrotnego testowania, które odnosi się do faktu, że test statystyczny z kilkoma niezależnymi hipotezami ma większe szanse na uzyskanie pozytywnej istotności i że szanse na to rosną wraz z testowaniem dodatkowych hipotez, nawet jeśli podstawowe rozkłady prawdopodobieństwa są identyczne. Aby uniknąć mylących wyników statystycznych, badanie eksploracyjne jest często łączone z badaniem potwierdzającym.
Wymogi dotyczące badania potwierdzającego opierają się na znacznie bardziej rygorystycznych kryteriach statystycznych. Po pierwsze, hipoteza badania, w tym kryteria istotności, musi zostać zdefiniowana przed zebraniem danych i przed analizą. Ponadto zestaw danych do analizy musi zostać zebrany wyłącznie w tym celu. Statystycznie niepoprawne jest ponowne wykorzystanie zestawu danych z badania eksploracyjnego w badaniu potwierdzającym, ponieważ ten zestaw danych z natury faworyzowałby proponowaną hipotezę. Końcowym wynikiem badania potwierdzającego jest odrzucenie lub przyjęcie hipotezy zgodnie z ustalonymi wcześniej kryteriami.
Testy statystyczne
W przypadku testów statystycznych analizowane jest prawdopodobieństwo, że obserwowane zjawisko wystąpiło przypadkowo. Nazywa się to hipotezą zerową28. Jeśli obserwowane zjawisko jest rzadkie zgodnie z hipotezą zerową, wniosek jest taki, że jest mało prawdopodobne, aby hipoteza zerowa była ważna. Hipoteza zerowa jest odrzucana, a prawdopodobieństwo hipotezy alternatywnej jako istotnej jest akceptowane.
Oszacowane prawdopodobieństwo, że obserwowane zjawisko wystąpiło przypadkowo, nazywane jest wartością p. Wartość p jest mierzona w zakresie od 0 do 1 lub równoważnie w jednostkach procentowych. Kryteria statystyczne dla badania potwierdzającego obejmują wartość graniczną alfa, poniżej której obliczone wartości p wskazywałyby na istotność obserwowanego zjawiska. Powszechnie stosuje się wartość graniczną alfa wynoszącą 5%, chociaż należy ją dostosować do pożądanych i niezbędnych kryteriów, które są specyficzne dla przedmiotu badania.
Opracowano wiele algorytmów obliczania wartości p przy różnych założeniach i do różnych celów. Powszechnie stosowanym algorytmem jest test t-Studenta. Test t-Studenta służy do obliczania wartości p na podstawie różnicy średnich wartości między dwiema grupami danych. Głównym założeniem testu t-Studenta jest to, że dwie grupy danych są niezależne i zgodne z rozkładem normalnym. Zaletą testu t-Studenta jest to, że jest on wydajny w porównaniu z nieparametrycznymi testami statystycznymi29. Testem nieparametrycznym, który jest równoważny testowi t-Studenta, może być jeden z najbardziej znanych nieparametrycznych testów statystycznych; test sumy rang Wilcoxona (czasami nazywany testem U Manna-Whitneya; nie mylić z testem rang Wilcoxona, który służy do porównywania dwóch sparowanych grup). Nieparametryczne testy statystyczne, takie jak test sumy rang Wilcoxona, mają przewagę nad parametrycznymi testami statystycznymi, takimi jak test t-Studenta, ponieważ nie zależą od wcześniejszych założeń dotyczących rozkładów zestawu danych. Test Kołmogorowa-Smirnowa dla rozkładu normalnego może być wykorzystany do podjęcia decyzji, czy zastosować test t-Studenta, czy jeden z testów nieparametrycznych
Oprócz wyboru algorytmu dla&
Oprócz wyboru algorytmu do obliczania wartości p, zbiory danych, które są wprowadzane do algorytmu obliczania wartości p mogą być manipulowane w celu ułatwienia obserwacji pożądanych właściwości w zbiorze danych. Połączenie etapów manipulacji surowymi danymi i wyboru algorytmu obliczania wartości p jest częścią budowania modelu hipotezy.
Istnieje wysoki poziom swobody w budowaniu modeli hipotez w fazie eksploracyjnej analizy statystycznej i jest to ważna część badania naukowego. Jednak hipoteza nigdy nie jest udowodniona przy użyciu naukowego, statystycznego podejścia. Prawidłowe podejście naukowe polega na sformułowaniu hipotezy zerowej, użyciu niezależnego (najlepiej nowo zebranego) zestawu danych i zaakceptowaniu lub odrzuceniu hipotezy zerowej zgodnie ze schematem badania potwierdzającego (Rysunek 10.10).
Techniki wizualizacji dla analizy jednoczynnikowej
Tak jak dostępnych jest wiele metod analizy, istnieje również wiele technik wizualizacji danych, z których można wybierać. W przypadku analizy danych jednozmiennowych odpowiednią techniką wizualizacji jest prosty wykres słupkowy z powiązanymi słupkami błędów. Mimo że jest to powszechna i prosta technika wizualizacji, istnieją kwestie, które warto podkreślić. Po pierwsze, słupki błędów mogą ilustrować różne źródła zmienności; nieodłączną zmienność danych (odchylenie standardowe, SD) lub precyzję, z jaką określono wartość średnią. Po drugie, precyzję, z jaką określono wartość średnią, można zilustrować na różne sposoby, ale ostatecznie zależy ona od kombinacji nieodłącznej zmienności danych wraz z liczbą próbek (N) i w swojej surowej postaci nazywana jest błędem standardowym średniej (SEM, Wzór 1):

Równanie 1. SEM
SEM nie jest jednak bardzo intuicyjną miarą i nie jest łatwo porównać SEM z różnych eksperymentów w znaczący sposób. Bardziej popularnym sposobem zilustrowania precyzji szacowanej średniej i wskazania istotności statystycznej w sposób graficzny jest przedział ufności (CI, Wzór 2):

Równanie 10-2.Cl
Obecność SEM można rozpoznać w równaniu dla przedziału ufności jako stosunek odchylenia standardowego (SD) do pierwiastka kwadratowego z liczby próbek (N), a zatem oczywiste jest, że przedział ufności opiera się na SEM. Dolna granica przedziału ufności jest konstruowana poprzez odjęcie SEM pomnożonej przez percentyl rozkładu t od średniej. Górna granica przedziału ufności jest konstruowana przez dodanie SEM pomnożonej przez percentyl rozkładu t od średniej. Poziom ufności przedziału ufności jest ustalany przez poziom ufności związany z wartością krytyczną t*; zazwyczaj jest to 95% poziom ufności.
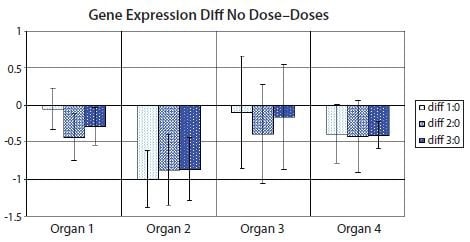
Rysunek 10.11 przedstawia wykres słupkowy ze słupkami błędów oznaczającymi 95% przedział ufności w każdej grupie eksperymentalnej, podkreślający niepewność związaną ze średnim oszacowaniem dla przykładowej ekspresji genu w próbkach z różnych narządów po leczeniu kilkoma dawkami leku. Ponadto, wartości istotności statystycznej testu t p są pokazane dla różnicy w ekspresji genów między próbkami kontrolnymi i każdą z trzech różnych próbek z różnych odpowiedzi na dawkę leku, wskazanych za pomocą notacji gwiazdkowej. Zwyczajowo jedna gwiazdka odpowiada wartości p poniżej 0,05, dwie gwiazdki odpowiadają wartości p poniżej 0,01, a trzy gwiazdki odpowiadają wartości p poniżej 0,001.

Rysunek 10.11.Krotna zmiana (log2) ekspresji genu zainteresowania względem pary genów referencyjnych, względem ekspresji w próbce o najniższej ekspresji w każdym typie narządu. Wysokości słupków wskazują średnią ekspresję genu w kilku próbkach w grupach próbek nieleczonych (dawka 0) lub próbek leczonych jedną z trzech różnych dawek leku (dawka 1, dawka 2 i dawka 3). Słupki błędu wskazują 95% przedział ufności dla średnich wartości ekspresji. Jedna gwiazdka wskazuje statystycznie istotną różnicę między średnimi zestawu próbek poddanych działaniu leku w porównaniu ze średnią zestawu próbek niepoddanych działaniu leku z dokładnością do 5%; dwie gwiazdki wskazują statystycznie istotną różnicę z dokładnością do 1%; trzy gwiazdki wskazują statystycznie istotną różnicę z dokładnością do 0,1%.
Zważywszy, że notacja gwiazdkowa ukrywa wartość bezwzględną p, często zaleca się dołączenie tabeli z wartościami bezwzględnymi p, jak pokazano w Tabeli 10.5. Jednym z powodów jest to, że wartość p wynosząca na przykład 0,032 jest tylko nieznacznie bardziej "znacząca" niż wartość p wynosząca 0,055. Przypadki graniczne, takie jak ten, mogą prowadzić do pewnych nieporozumień przy podejmowaniu decyzji, jaki dokładnie punkt odcięcia zastosować przy klasyfikowaniu danych jako znaczące. W realistycznych przypadkach wartość p wynosząca 0,051 może być tak samo istotna jak wartość p o wartości 0,049, ale ścisłe (choć zasadniczo arbitralne) odcięcie 0,05 sklasyfikowałoby jedną z nich jako znaczącą, a drugą nie.
Istnieje jednak wariant wizualizacji diagramu słupkowego, który wykorzystuje przedział ufności różnicy między średnimi, aby uniknąć wielu, jeśli nie wszystkich, wad tradycyjnych diagramów słupkowych24. Dzięki przedziałowi ufności różnicy między średnimi możliwe jest bezpośrednie oszacowanie istotności statystycznej z powiązanymi słupkami błędów, przy jednoczesnym podkreśleniu wielkości efektu biologicznego i zmienności danych. Rysunek 10.12 pokazuje wariant z przedziałem ufności różnicy między średnimi danych wykorzystanych na Rysunku 10.11. Zwróć uwagę, że przedziały ufności, które nie obejmują zerowej różnicy między średnimi, odpowiadają znaczącym wynikom na poziomie ufności odpowiadającym wartości granicznej p (5% na Rysunku 10.11 i Tabeli 10.5).
Rysunek 10.12.Wykres słupkowy przedstawiający różnicę między średnimi nietraktowanego zestawu próbek (dawka 0) i jednego z traktowanych zestawów próbek (dawka 1, dawka 2 lub dawka 3) w zestawie danych z rysunku 10.11. Słupki błędów pokazują przedział ufności różnicy między średnimi. Słupki błędu, które nie przecinają osi x wskazują, że porównanie odpowiednich średnich jest statystycznie istotne na poziomie 5% w teście t. PCR Technology, Current Innovations-3rd ed. by Taylor and Francis Group LLC Books. Reproduced with permission of Taylor and Francis Group LLC Books in the format reuse in a book/e-book via Copyright Clearance Center.
Dane wielowymiarowe to dane zebrane dla kilku zmiennych dla każdej jednostki próbkowania. Dane wykorzystane na rysunkach 10.11 i 10.12 są wielowymiarowe, ponieważ zależą od zmiennych takich jak dawka i typ narządu. Jednak analizy statystyczne na Rysunkach 10.11 i 10.12 są mimo wszystko jednoczynnikowe, ponieważ każda reprezentacja (słupek) ilustruje tylko jedną zmienną, ekspresję genu, w odniesieniu do stałych miar innych zmiennych. W przypadku technik analizy danych wielowymiarowych, hierarchiczne grupowanie i analiza składowych głównych są dobrymi opcjami reprezentacji danych.
Hierarchiczne grupowanie
Jedną z najprostszych i użytecznych metod charakteryzacji danych jest wykreślenie danych na wykresie rozrzutu (na przykład wykreślenie zmierzonego C< sub>q& C< sub>q).sub>q wartości jednego genu względem odpowiadających im wartości Cq innego genu dla zestawu próbek biologicznych na wykresie 2D). Wykresy w jednym lub dwóch wymiarach są wygodnie wizualizowane przez ludzkie oczy. Wykresy w trzech wymiarach mogą być również możliwe przy użyciu odpowiednich narzędzi, ale wykresy w wyższych wymiarach są znacznie trudniejsze do wizualizacji. Jednak w przypadku badań eksploracyjnych zestaw danych jest z natury wielowymiarowy, a wykresy rozrzutu całych zestawów danych mogą stać się niepraktyczne. Z zestawu danych qPCR może być na przykład reprezentowanych kilka genów i/lub kilka rodzajów próbek biologicznych.
Popularnym, alternatywnym sposobem charakteryzowania i wizualizacji danych z badań eksploracyjnych jest analiza miar odległości między punktami danych na wykresie rozrzutu. Istnieją różne miary odległości, w tym korelacje Euklidesa, Manhattanu i Pearsona. Dzięki mocy obliczeniowej można łatwo obliczyć odległości, nawet dla danych wielowymiarowych o znacznie większej wymiarowości niż trzy wymiary. W przypadku aglomeracyjnego grupowania hierarchicznego wykonywany jest następujący proces iteracyjny: 1) Znajdź dwa najbliższe obiekty i połącz je w klaster; 2) Zdefiniuj nowy klaster jako nowy obiekt za pomocą metody klastrowania; 3) Powtarzaj od 1), aż wszystkie obiekty zostaną połączone w klastry30. Alternatywne metody grupowania obejmują metodę Warda, pojedyncze powiązanie i średnie powiązanie31. Dendrogram jest często używany do wizualizacji wyników hierarchicznego grupowania.
Interpretacja dendrogramów hierarchicznego grupowania danych qPCR często prowadzi do wniosków na temat podobieństw profili ekspresji genów. W badaniu eksploracyjnym podobieństwa te mogą być następnie wykorzystane do sformułowania hipotez dotyczących koregulacji ekspresji genów, które mogą zostać zaakceptowane lub odrzucone w kolejnych badaniach potwierdzających. Zalety hierarchicznych dendrogramów grupowania obejmują przejrzystość, dzięki której wizualizowane są relacje podobieństwa. Z drugiej strony, silny nacisk na miary podobieństwa może być postrzegany jako ograniczający w odniesieniu do formułowania hipotez, ponieważ podobne profile ekspresji mogą być zbędnymi atrybutami w hipotezach. Większą wartość może mieć identyfikacja zestawów profili ekspresji, które uzupełniają się nawzajem w określonej kombinacji, aby odpowiedzieć na pożądaną hipotezę.
Principal Component Analysis
.Innym popularnym, alternatywnym sposobem charakteryzacji i wizualizacji danych z badań eksploracyjnych jest wykorzystanie informacji zawartych w całym, wielowymiarowym zbiorze danych, wybranie pożądanych właściwości i rzutowanie ich na wykres rozrzutu o niższym wymiarze, taki jak wykres 2D lub 3D. Można to osiągnąć za pomocą analizy głównych składowych (PCA)32,33,34, 35. W tym przypadku oryginalny układ współrzędnych zestawu danych (tj. profile ekspresji zmierzone za pomocą qPCR) jest przekształcany w nową wielowymiarową przestrzeń, w której konstruowane są nowe zmienne (główne składowe: PC lub czynniki). Każda PC jest liniową kombinacją podmiotów w oryginalnym zestawie danych. Zgodnie z definicją matematyczną, PC są wyodrębniane w kolejności ważności. Oznacza to, że pierwszy PC wyjaśnia większość informacji (wariancji) obecnych w danych, drugi mniej i tak dalej. W związku z tym pierwsze dwie lub trzy współrzędne PC (określane jako wyniki) można wykorzystać do uzyskania projekcji całego zestawu danych na dogodnie mały wymiar, odpowiedni do wizualizacji na wykresie 2D lub 3D. Używając pierwszych dwóch lub trzech PC do reprezentacji, uzyskuje się projekcję, która uwzględnia największą zmienność w zestawie danych. Oczekuje się, że wariancja z warunków projektu eksperymentalnego będzie systematyczna, podczas gdy wariancja zakłócająca będzie losowa, więc ta reprezentacja może być pożądana w odpowiednich warunkach.
Jak wcześniej zauważono w przypadku hierarchicznego grupowania, interpretacja qPCR PCA często prowadzi do wniosków na temat podobieństw profilu ekspresji genów. Chociaż PCA i hierarchiczne grupowanie mogą dawać komplementarny wgląd we wzorce współregulacji ekspresji genów, obie techniki koncentrują się na podobieństwach profili ekspresji genów. Nakłada to ograniczenia na rodzaje hipotez, które można znaleźć w badaniach eksploracyjnych wykorzystujących tylko te techniki. Aby rozszerzyć zasięg generowanych hipotez w badaniach eksploracyjnych, niedawno zaproponowano podejście oparte na hipotezach do analizy wielowymiarowej24. Oparte na hipotezach, niestandardowo zaprojektowane algorytmy mogą identyfikować istotne biologicznie hipotezy, które w przeciwnym razie mogłyby zostać pominięte przez powszechnie stosowane techniki analizy danych wielowymiarowych.
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?