Zaprojektowane siRNA
MISSION® Predesigned siRNA zostały stworzone przy użyciu zastrzeżonego algorytmu Rosetta Inpharmatics siRNA Design w ramach wyłącznej współpracy z Merck & Co. Algorytm Rosetta siRNA Design Algorithm wykorzystuje macierze punktacji specyficzne dla pozycji i wiedzę o regionie zalążkowym, aby przewidzieć najbardziej specyficzne i skuteczne sekwencje dla genów docelowych. Reguły algorytmu zostały opracowane z wykorzystaniem danych empirycznych zebranych z eksperymentów wyciszania genów przeprowadzonych w ciągu trzech lat.
Zalety produktu
- Najlepsze w swojej klasie, gwarantowane wyciszanie genów
- Wydajna eliminacja komunikatów o niskiej liczebności
- Uproszczona optymalizacja transfekcji dzięki 11 Positive Control siRNA
- Odróżnij wyciszanie specyficzne dla sekwencji od efektów niespecyficznych za pomocą 8 negatywna kontrola siRNA
- Setki funkcjonalnie zatwierdzonych, wstępnie zaprojektowanych siRNA
Cechy produktu
- Gatunki: Człowiek, mysz i szczur
- Ilości: 2 (10 nmol), 5 (25 nmol) i 10 (50 nmol) OD
- Oczyszczanie: Desalt lub HPLC
- Format sekwencji: 21-merowe dupleksy ze zwisami dTdT
- Kontrola jakości: 100% spektrometria masowa*
- Format: Dostarczany w postaci suchej w probówkach
*W zależności od miejsca produkcji, PAGE może być stosowany do oceny dupleksów siRNA.
Gwarancja wstępnie zaprojektowanego siRNA
Co najmniej dwa z trzech zakupionych wstępnie zaprojektowanych siRNA MISSION® ukierunkowanych na ten sam gen obniżą poziom docelowego mRNA o 75% w hodowanych komórkach, gdy zostaną transfekowane w stężeniu ≥30 nM. Jeśli dwa siRNA nie obniżą poziomu docelowego genu o 75%, dostarczymy bezpłatnie trzy dodatkowe siRNA dla tego genu. Jeśli nie będzie więcej siRNA dla tego genu lub wszystkie siRNA nie znokautują genu docelowego o 75%, zwrócimy cenę zakupu.
Otrzymanie odpowiednich danych potwierdzających skuteczność transfekcji jest wymagane do zastosowania gwarancji. Odpowiednie dane potwierdzające skuteczność transfekcji obejmowałyby dane qPCR porównujące poziomy docelowego mRNA siRNA MISSION Positive Control (GAPDH, MAPK1, TP53 itp.), transfekowanego przy ≥30 nM, z odpowiednią kontrolą negatywną (taką jak transfekcja mock, zakodowana sekwencja siRNA lub siRNA MISSION Universal Negative Control), wykazując knockdown docelowego mRNA o 75%.
Ze względu na zmienność przeciwciał i okresów półtrwania białek, nie jesteśmy w stanie zaakceptować danych z metod wykrywania opartych na białkach.
Pule produktów
Popularny format puli to 4 dupleksy po 5 nmol każdy połączone w jednej probówce (20 nmol w puli) plus dokładnie te same 4 dupleksy również po 5 nmol każdy w oddzielnych probówkach (kolejne 20 nmol indywidualnie). Jednak nasze zaawansowane urządzenia do obsługi cieczy pozwalają na szeroki zakres innych możliwości. Aby zapoznać się z wykonalnością konkretnych potrzeb, skontaktuj się z sirnarequest@sial.com.
Zwalidowane siRNA
Wiele popularnych celów genowych zostało zwalidowane dla ≥75% knockdownu mRNA (Rysunek 1 dla przykładowych danych i tabela z listą powszechnie stosowanych, zwalidowanych siRNA według symbolu genu). Zatwierdzone siRNA są odpowiednie do optymalizacji transfekcji i jako kontrole pozytywne.
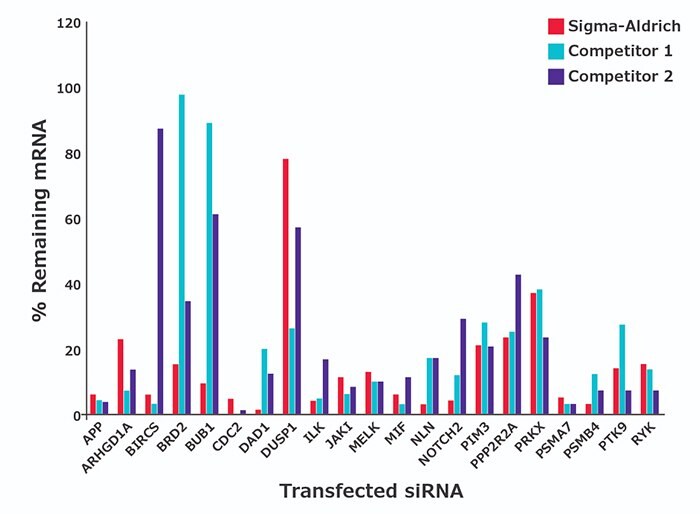
Rysunek 1.Komórki HeLa transfekowane wcześniej zaprojektowanym siRNA w stężeniu 30 nM. Procent pozostałych poziomów ekspresji genów mierzono za pomocą qPCR 48 godzin po transfekcji (w stosunku do mock). Dane reprezentują średnią z czterech powtórzeń biologicznych.
Wybierz cytaty
Jeśli potrzebna jest dodatkowa pomoc, prosimy o kontakt z naszą grupą usług technicznych pod adresem oligotechserv@sial.com.
MISSION jest znakiem towarowym firmy Merck KGaA, Darmstadt, Niemcy i/lub jej podmiotów stowarzyszonych. Label License.
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?