Test immunoprecypitacji chromatyny (ChIP)
Co to jest test ChIP?
Testy immunoprecypitacji chromatyny (ChIP) są wykorzystywane do oceny interakcji czynnik transkrypcyjny-DNA i mają kluczowe znaczenie dla postępów w badaniach nad regulacją ekspresji genów i modyfikacjami epigenetycznymi. ChIP może wykrywać i względnie określać ilościowo specyficzne interakcje białko-DNA i białko-białko in vivo w pojedynczym locus lub wielu loci. ChIP obejmuje chemiczne sieciowanie białek z sekwencjami DNA, po którym następuje immunoprecypitacja usieciowanych kompleksów (rysunek 1) i analiza powstałego DNA za pomocą końcowej lub ilościowej reakcji łańcuchowej polimerazy (qPCR) (rysunki 2-4), mikromacierzy (ChIP-chip) lub sekwencjonowania następnej generacji (ChIP-seq) (rysunki 5 i 6).
Przebieg testu immunoprecypitacji chromatyny (ChIP)
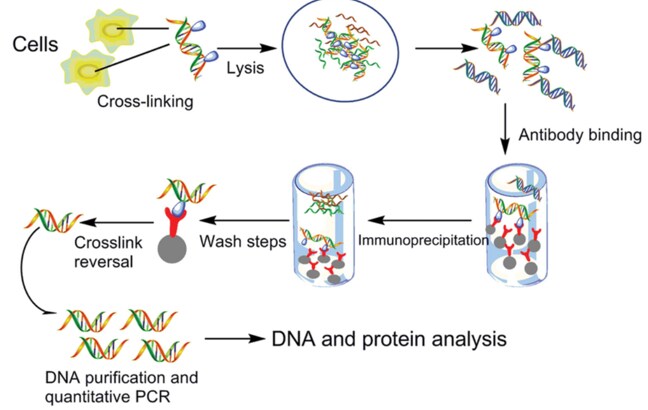
Rysunek 1.Przebieg procedury ChIP
Song et al., 2015
Białko i związana z nim chromatyna w żywych komórkach lub tkankach są sieciowane przy użyciu formaldehydu. Usieciowane kompleksy DNA-białko (chromatyna-białko) są następnie ścinane na fragmenty DNA o wielkości ∼500 bp przy użyciu trawienia enzymatycznego lub fizycznego ścinania za pomocą sonikacji. Kompleksy DNA-białko są następnie immunoprecypitowane przez odpowiednie przeciwciało specyficzne dla białka. Po odwróceniu wiązań krzyżowych powiązane fragmenty DNA są eluowane, po czym następuje immunoprecypitacja usieciowanych kompleksów i analiza powstałego DNA za pomocą końcowej lub ilościowej reakcji łańcuchowej polimerazy (qPCR), mikromacierzy (ChIP-chip) lub sekwencjonowania nowej generacji (ChIP-seq).
Testy ChIP mogą być wykorzystywane do następujących badań:
- Sekwencje DNA zajmowane przez wiele specyficznych białek docelowych
- Miejsca wiązania i dystrybucja określonego białka, takiego jak czynniki transkrypcyjne, kofaktory transkrypcji, czynniki replikacji DNA i białka naprawy DNA w całym genomie, w określonych warunkach komórkowych
- Transkrypcja genów i aktywność polimerazy
- Złożone interakcje DNA/białko leżące u podstaw fenotypów chorobowych
- Modyfikacje białek, takich jak histony, które mogą wpływać na strukturę chromatyny i ekspresję genów
- Architektura nukleosomów i regulacja utrzymania chromosomów
ChIP wzbogacanie sekwencji DNA z komórek
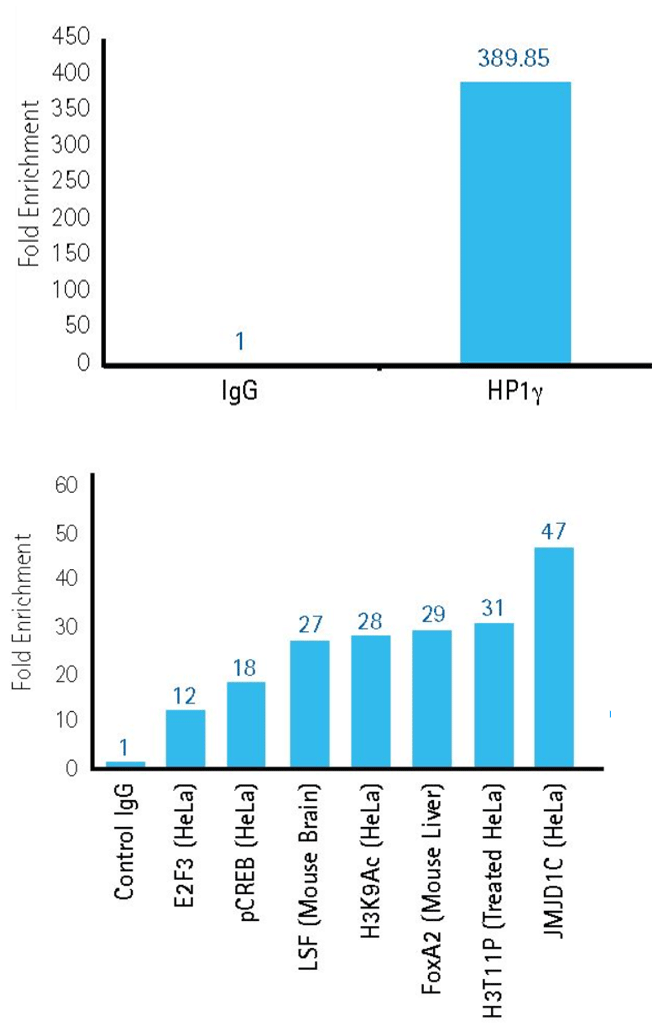
Rysunek 2.ChIP z próbkami linii komórkowych.
Górny panel: Udane wzbogacenie ChIP sekwencji DNA związanych z białkiem heterochromatyny 1d gamma (HP1gamma), ważnym markerem represji genów. Dane zebrane przy użyciu zestawu przeciwciał/primeru ChIPAb+™ Hp1gamma, mysiej IgG (kontrola niespecyficzna) i zestawu Magna ChIP™ G (nr produktu 17-10085).
Dolny panel: Wysokoprzepustowa (96-dołkowa płytka) analiza ChIP wielu docelowych białek w celu wyszukiwania loci genowych w różnych warunkach przy użyciu paneli przeciwciał i zestawu Magna ChIP™ HT96 (nr produktu 17-10077).
Efektywne testy ChIP z wykorzystaniem próbek tkanek
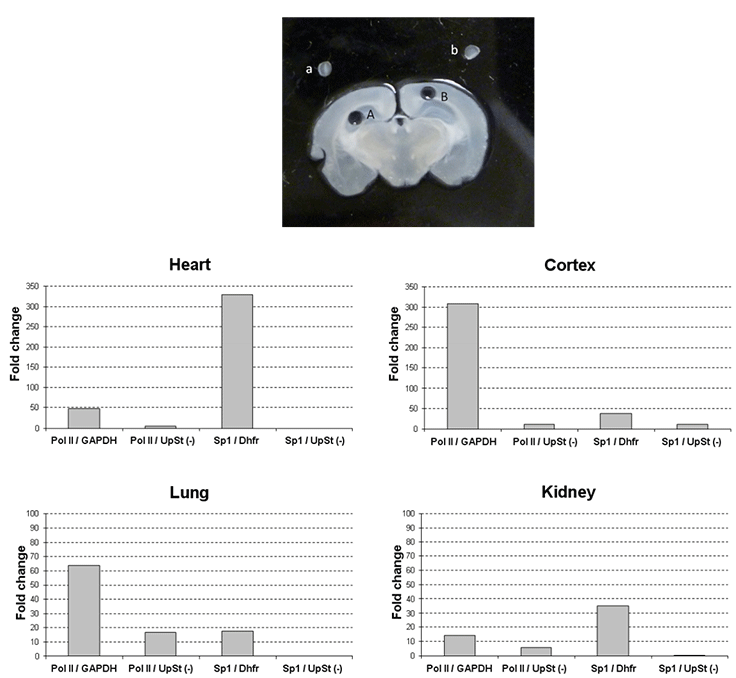
Rysunek 3.ChIP z próbkami tkanek.
Panel górny: Kriosekcja tkanki i wyizolowanie próbki tkanki specyficznej dla regionu za pomocą dostarczonego 1 mm stempla do mikrodysekcji. Przykład izolacji tkanki specyficznej dla regionu jest pokazany na obrazku. 300 µm przekrój koronalny mózgu myszy z mikrodysekcją specyficzną dla regionu hipokampa (A) i kory mózgowej (B). Wyizolowaną tkankę umieszczono nad wyciętym regionem (a: hipokamp, b: kora mózgowa). Oczyszczona tkanka jest następnie rozpraszana roztworem stabilizującym tkanki, a następnie poddawana działaniu formaldehydu. Formaldehyd sieciuje białka z DNA, aby zapewnić współstrącanie.
Dolny panel: Chromatyna z różnych linii komórkowych została poddana immunoprecypitacji ze wskazanymi przeciwciałami przy użyciu zestawu Magna ChIP™ G Tissue (nr produktu 17-20000). IgG stosowane do względnego porównania było albo króliczym oczyszczonym IgG (nr produktu PP64B) lub mysim oczyszczonym IgG (nr produktu 12-371B), w zależności od przeciwciała ChIP. Dane ilościowego PCR przedstawiono jako krotność względnego wzbogacenia w IgG z niezależnych eksperymentów lub jako % danych wejściowych. Dla biologicznej kontroli negatywnej, qPCR oceniano za pomocą starterów przed genem Dhfr (UpSt (-)). Zastosowano następujące przeciwciała i startery: Anti-RNA Polymerase II clone CTD4H8 (Product No. 05-623B): 1 µg mysiego monoklonalnego przeciwciała oczyszczonego metodą powinowactwa immunoprecypitowanego z chromatyną różnych tkanek myszy i poddanego qPCR ze starterami specyficznymi dla mysiego promotora GAPDH i Anti-SP1 (Product No. 07-645): 1 µg mysiego monoklonalnego przeciwciała oczyszczonego przez powinowactwo, immunoprecypitowanego z chromatyny różnych tkanek myszy i poddanego testowi qPCR ze starterami specyficznymi dla mysiego promotora Dhfr.
Większe wzbogacenie fałdu przy użyciu małej ilości chromatyny lub krótszy czas procedury testu ChIP
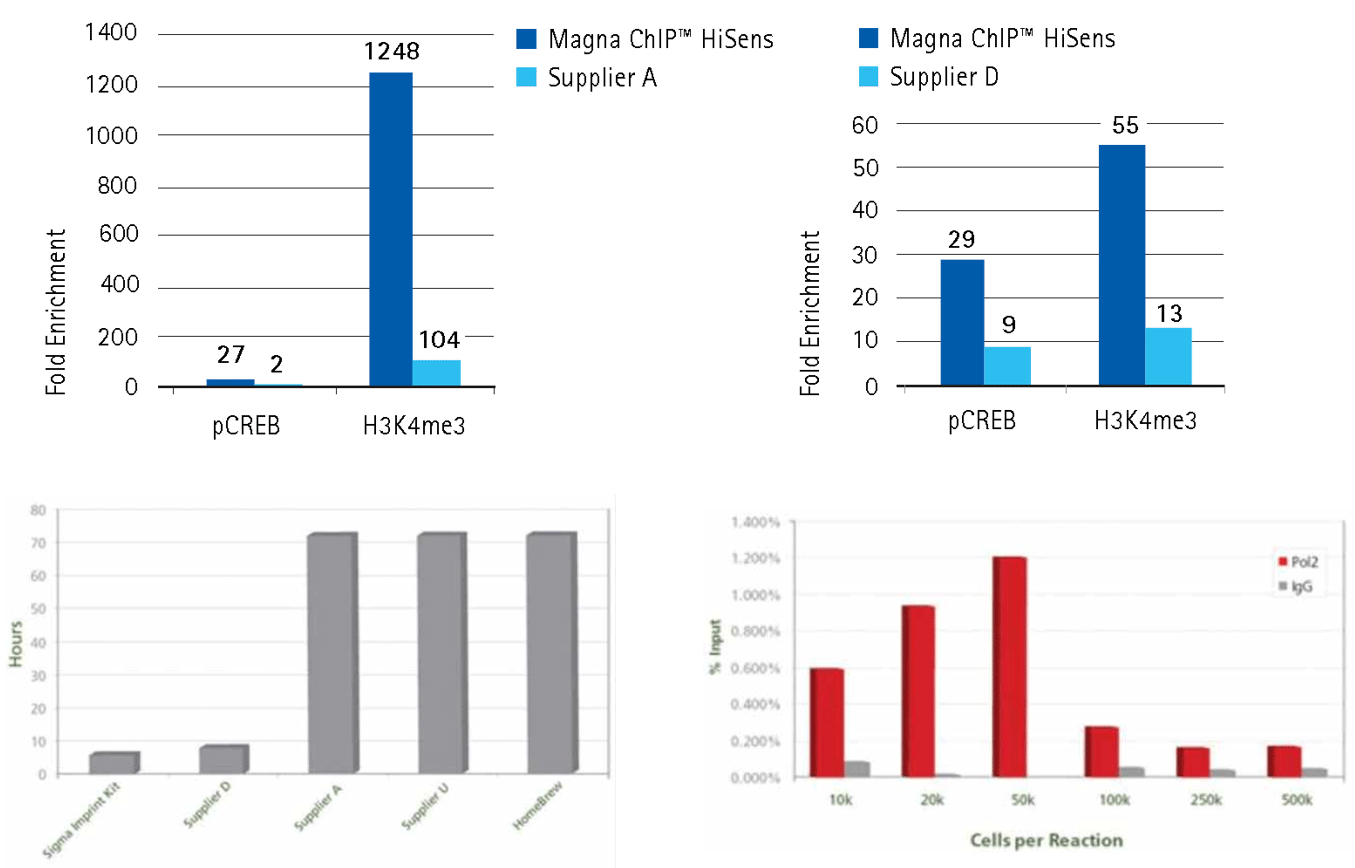
Rysunek 4.Nasze zestawy zapewniają większe wzbogacenie przy użyciu niewielkich ilości chromatyny lub krótszej procedury niż zestawy innych dostawców.
Górny panel: 10 000 równoważników komórkowych sonikowanej chromatyny przygotowanej z komórek HeLa poddano immunoprecypitacji chromatyny przy użyciu 1 µg oczyszczonej IgG (królicza IgG, nr produktu. 12-370) lub specyficznych przeciwciał (H3K4Me3, nr produktu 17-614; Phospho-CREB, nr produktu 17-10131). W każdym teście wykorzystano odczynniki i protokół dostarczony z zestawem Magna ChIP™ HiSens Kit (nr produktu 17-10460) lub te z niskonakładowego zestawu ChIP od dostawcy A lub dostawcy D. Immunoprecypitację fragmentów DNA związanych z przeciwciałem zweryfikowano za pomocą qPCR przy użyciu starterów kontroli pozytywnej (startery promotora GAPDH dla H3K4me3 i startery promotora c-Fos dla pCREB) i starterów kontroli negatywnej (startery promotora ludzkiej ß-globiny). Wyniki odzwierciedlają analizę 2 µL z 50 µL całkowitego DNA na reakcję qPCR.
Dolny panel: Kompletna reakcja ChIP w ciągu 6 godzin w elastycznym formacie dołka dla Imprint® Chromatin Immunoprecipitation Kit (nr produktu ChIP1) Porównanie czasu wymaganego do ukończenia protokołu od utrwalenia do oczyszczenia przy użyciu różnych zestawów do immunoprecypitacji chromatyny. Każdy eksperyment ChIP przeprowadzono przy użyciu protokołu zalecanego przez każdego producenta i porównano całkowity wymagany czas. Komórki HeLa zostały policzone, utrwalone i poddane immunoprecypitacji zgodnie z biuletynem Imprint CHP1 z opcjonalną metodą wysokiej czułości. Testy ChIP przeprowadzono przy użyciu anty-H3K9ac (H9286) i dostarczonych w zestawie przeciwciał przeciwko ludzkiej polimerazie RNA II i niespecyficznej mysiej IgG. Aby ocenić wzbogacenie ChIP, przeprowadzono SYBR qPCR ukierunkowany na region promotora GAPDH (gen podtrzymujący o wysokiej ekspresji). Procent wejścia opisuje ilość DNA z i bez selekcji przeciwciał. W przypadku tych przeciwciał pozorne wzbogacenie wzrosło wraz z mniejszą liczbą komórek na reakcję z powodu zmniejszonego niespecyficznego ściągania.
ChIP DNA analizowany przez sekwencjonowanie następnej generacji (ChIP-seq)
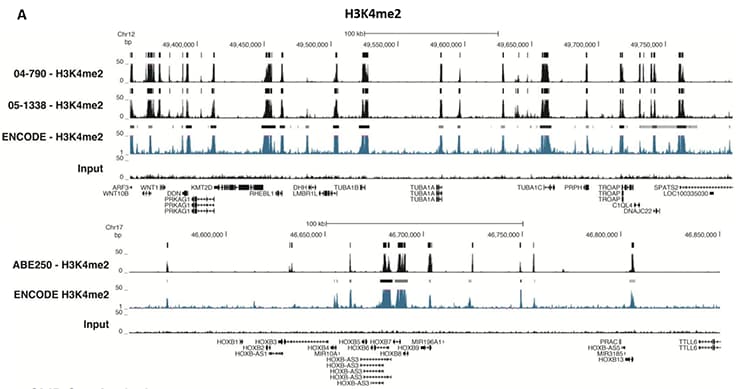
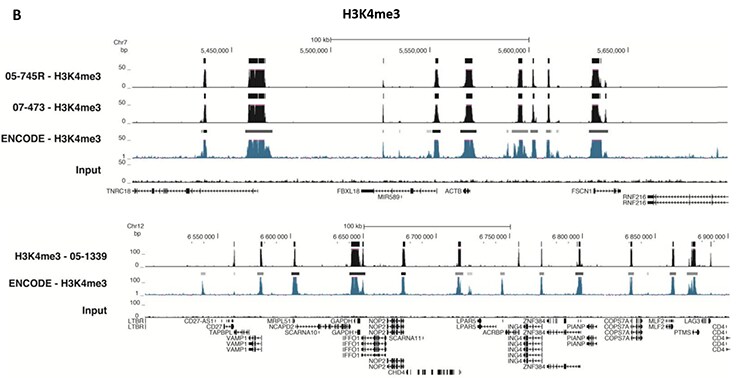
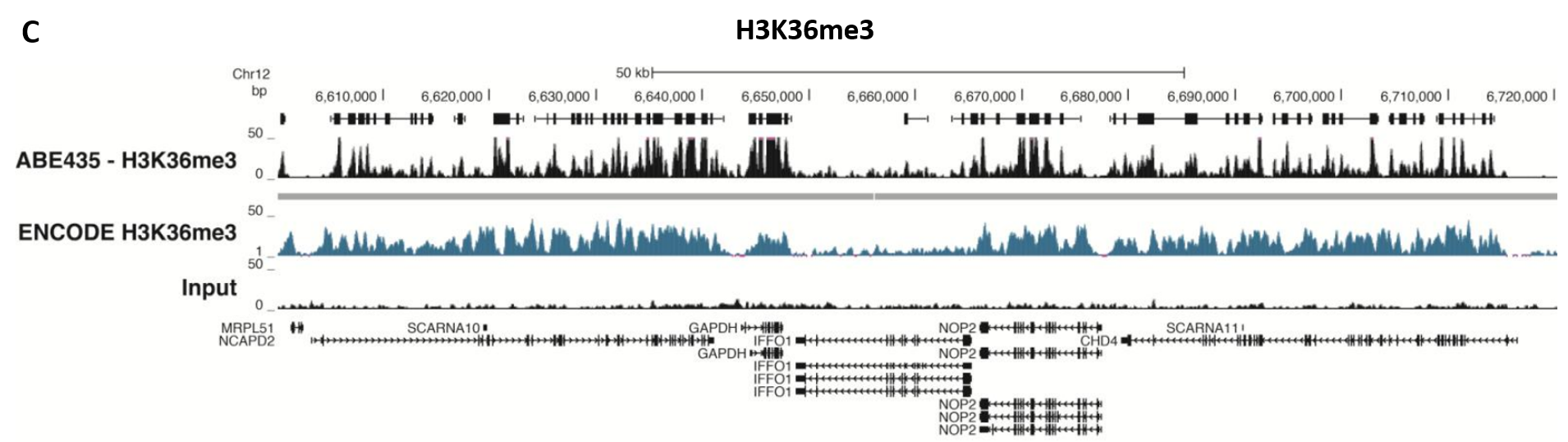
Rysunek 5.Analiza ChIP-Seq
Immunoprecypitację chromatyny przeprowadzono przy użyciu zestawu Magna ChIP™ HiSens (nr produktu 17-10460), 2 µl, 2 µg lub 5 µg przeciwciała anty-H3K4me2 (nr produktu. 04-790, 05-1338 lub ABE250) lub 1 lub 3 µL lub 4 µg przeciwciała anty-H3K4me3 (Nr produktu. 05-745R, 07-473 lub 05-1339), lub 2 µg przeciwciała anty-H3K36me3 (Nr produktu. ABE435), 20 µl kulek magnetycznych Protein A/G i 1e6 lub 4e6 lub 5e6 usieciowanej chromatyny komórek HeLa, a następnie oczyszczono DNA. Biblioteki zostały przygotowane z próbek Input i ChIP DNA przy użyciu standardowych protokołów z adapterami Illumina z kodem kreskowym i analizowane na instrumencie Illumina HiSeq™. Ponad dwanaście milionów odczytów z plików FastQ zmapowano za pomocą programu Bowtie (http://bowtiebio.sourceforge.net/manual.shtml) po usunięciu tagów TagDust (http://genome.gsc.riken.jp/osc/english/dataresource/). Piki zostały zidentyfikowane przy użyciu MACS (http://luelab.dfci.harvard.edu/MACS/), z pikami i odczytami wizualizowanymi jako niestandardowa ścieżka w UCSC Genome Browser (http://genome.ucsc.edu) z plików BigWig i BED. Najwyższe 25% pików zidentyfikowanych w danych wykazało 90-99% pokrycie z pikami zidentyfikowanymi w ścieżkach ENCODE H3K4me2 lub H3K4me3 lub H3K36me3 BROAD Histone dla HeLa S3. Pokazano dane w regionie transkrypcyjnie aktywnego genu podtrzymującego TUBA1A (tubulina, alfa 1A), HOXB, ACTB lub GAPDH.
Test ChIP i sekwencjonowanie następnej generacji przy użyciu niewielkich ilości DNA
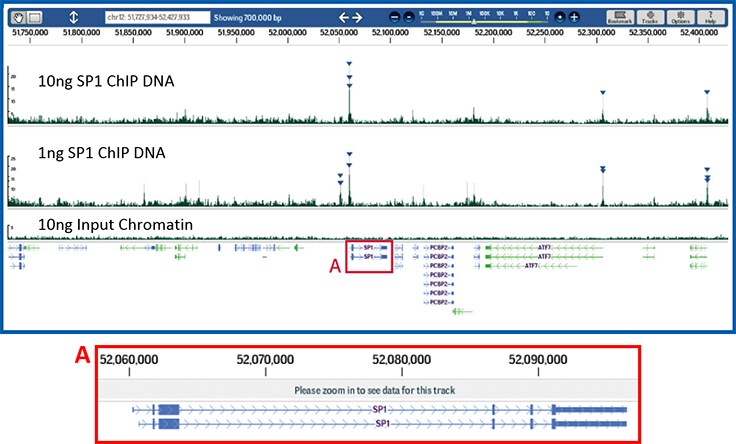
Rysunek 6.Skuteczne ChIP i niezawodne tworzenie bibliotek sekwencjonowania następnej generacji z ograniczonych ilości DNA.
Biblioteki sekwencjonowania następnej generacji zostały skonstruowane z DNA Sp1 ChIP przy użyciu 10 ng (górny panel) lub 1 ng kwantyfikowanego immunoprecypitowanego DNA (środkowy panel). Bibliotekę referencyjną utworzono przy użyciu 10 ng oczyszczonego DNA z chromatyny wejściowej. Próbki przygotowano przy użyciu zestawu Magna ChIP-Seq Kit (nr produktu 17-1010) i zestawu przeciwciał/primerów ChIPab+ Sp1 (nr produktu 17-601). Powstałe biblioteki zostały zsekwencjonowane na urządzeniu Illumina Genome Analyzer, a następnie wyrównane i zmapowane do ludzkiego genomu referencyjnego hg18. Powyżej przedstawiono wynikową analizę pików (uzyskaną przy użyciu QuEST) locus Sp1 z pewnie zmapowanych odczytów przeglądanych za pomocą oprogramowania DNAnexus™. Porównano powtórzone przebiegi sekwencjonowania z bibliotek 10 ng i 1 ng, wykazując nakładającą się identyfikację pików w promotorze genu Sp1. Trójkąty odzwierciedlają zdarzenia wiązania Sp1 o wysokim prawdopodobieństwie związane głównie w pobliżu miejsc startu transkrypcji w tym przedziale genomowym.
Przewodniki po eksperymentach ChIP, FAQ, porady dotyczące rozwiązywania problemów i protokoły uzupełniające
ChIP może stanowić wyzwanie nawet dla najbardziej doświadczonych badaczy. Jest to wieloetapowa technika (rysunek 1), która wymaga wysokiej jakości chromatyny, solidnych przeciwciał, zoptymalizowanych odczynników i protokołów w celu uzyskania wiarygodnych i powtarzalnych wyników. Zazwyczaj osiąga się to poprzez stosowanie komercyjnie przygotowanych produktów i protokołów lub przeprowadzanie wielu eksperymentów walidacyjnych. Niezależnie od preferowanej strategii, poniżej znajdują się kompleksowe przewodniki po eksperymentach ChIP, które pomogą Ci rozwiązać niektóre z problemów często napotykanych w ChIP, od przygotowania próbki do dalszej analizy DNA.
- Przewodniki po liczbie komórek dla ChIP i analizy punktu końcowego
- Kulki agarozowe vs. kulki magnetyczne
- Sieciowanie białek do DNA i liza komórek
- immunoprecypitacja, przemywanie i elucja
- ChIP-qPCR i analiza danych (% input i fold enrichment)
- Guide to peak calling for ChIP-Seq
- FAQs (Antibodies, fusion tag, cross-link and beads, chromatin fragmentation and data analysis)
- Wskazówki dotyczące rozwiązywania problemów (wysokie tło, niski odzysk DNA, brak amplifikacji DNA, pobieranie tylko dużych DNA, niespecyficzny osad DNA)
- Protokoły uzupełniające
Który zestaw ChIP jest odpowiedni dla Ciebie?
Standardowe zestawy ChIP
Specjalistyczne zestawy ChIP
Akcesoria ChIP
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?