Podwójnie znakowane sondy w cyfrowym PCR
Digital PCR (dPCR) to metoda PCR punktu końcowego, która jest stosowana do bezwzględnej kwantyfikacji i analizy sekwencji mniejszościowych na tle podobnych sekwencji większościowych, np. kwantyfikacji mutacji somatycznych. Podczas korzystania z tej techniki próbka jest pobierana do rozcieńczenia ograniczającego (dokładniej, dzielona), a liczba reakcji pozytywnych i negatywnych jest wykorzystywana do określenia dokładnego pomiaru stężenia docelowego.
Próbka jest dzielona w niektórych przypadkach nawet na milion lub więcej komór reakcyjnych, z których niektóre zawierają jedną kopię docelowej cząsteczki kwasu nukleinowego (pozytywna), a niektóre nie zawierają kopii (negatywna) - pozytywna i negatywna są również opisywane jako "1" i "0," odpowiednio, stąd "cyfrowy.
Niniejszy artykuł rozpoczyna się od podstawowego przeglądu cyfrowego PCR, a kończy studium przypadku, w którym nasze podwójnie znakowane sondy wypadły w porównaniu z sondami konkurencji testowanymi przez klienta.
Przegląd
Podczas wykonywania konwencjonalnego PCR, końcowe stężenie matrycy jest proporcjonalne do początkowej liczby kopii i liczby cykli amplifikacji. Jeden eksperyment o określonej liczbie reakcji jest wykonywany na pojedynczej próbce, a wynikiem jest analiza wielkości fragmentów lub, w przypadku ilościowego PCR w czasie rzeczywistym (qPCR), analiza jest oszacowaniem stężenia sekwencji docelowych w reakcji na podstawie liczby cykli wymaganych do osiągnięcia cyklu kwantyfikacji (Cq). W przypadku korzystania z dPCR próbka jest rozdzielana na dużą liczbę komór reakcyjnych, tak aby każda partycja zawierała jedną lub żadną kopię sekwencji docelowej (Rysunek 1, Tabela 1). Liczba komór reakcyjnych lub partycji różni się w zależności od systemu, od kilku tysięcy do milionów. PCR jest następnie wykonywany w każdej partycji, a amplikon wykrywany przy użyciu etykiety fluorescencyjnej, tak że zebrane dane są serią wyników pozytywnych i negatywnych. Teoretycznie skutkowałoby to pozytywnym sygnałem z partycji, która pierwotnie zawierała pojedynczą kopię celu i negatywnym (tj. brakiem sygnału) z partycji, która pierwotnie nie zawierała szablonu. Ponieważ jednak możliwe jest, że niektóre partycje będą zawierać więcej niż jedną kopię szablonu, rozproszenie próbki na partycje jest uważane za zgodne z rozkładem Poissona.
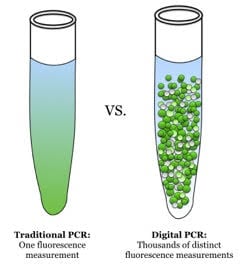
Rysunek 1. Tradycyjny PCR (qPCR) vs dPCR. W przypadku qPCR wykonywany jest jeden pomiar fluorescencji, podczas gdy w przypadku dPCR wykonywanych jest wiele takich pomiarów (źródło: Wikimedia Commons). Produkty końcowe PCR są często analizowane metodami niefluorescencyjnymi, np. wykrywaniem kolorymetrycznym.
W teorii, dPCR może być wykorzystywany do przezwyciężenia niektórych trudności napotykanych podczas stosowania konwencjonalnej metody PCR. Podczas korzystania z dPCR próbka jest dzielona tak, że poszczególne cząsteczki kwasu nukleinowego w próbce są zlokalizowane w wielu oddzielnych regionach, a zatem wykrywanie dowolnego celu nie zależy od liczby cykli amplifikacji. Podejście to skutkuje znacznie bardziej czułym różnicowaniem zmian krotności niż to zapewniane przez qPCR; dobrze zoptymalizowany qPCR może w najlepszym przypadku różnicować 1,5-krotne zmiany, podczas gdy dPCR został zgłoszony do różnicowania 1,2-krotnego.1 Rozcieńczenie próbki sprawia, że jest to przydatne narzędzie do badania sekwencji mniejszościowych w stosunku do większości podobnych, ale różniących się sekwencji. Na przykład dPCR może być stosowany do wykrywania somatycznego polimorfizmu pojedynczego nukleotydu (SNP) o niskiej częstości występowania w stosunku do wysokiego stężenia sekwencji WT. Dzieje się tak dlatego, że gdy cała próbka jest podzielona, rzadka sekwencja jest również podzielona na pojedynczą kopię, więc będzie amplifikowana przy braku konkurencji ze strony wybitnej sekwencji. Znacznie mniej partycji będzie zawierało wynik pozytywny dla rzadkiego SNP niż dla WT, więc możliwe jest dokonanie dokładnego pomiaru stosunku dwóch sekwencji.
Cyfrowa PCR ma wiele potencjalnych zastosowań, w tym wykrywanie i kwantyfikację patogenów niskiego poziomu, rzadkich sekwencji genetycznych, wariacji liczby kopii (CNV) i względnej ekspresji genów w pojedynczych komórkach. Amplifikacja klonalna możliwa dzięki jednoetapowemu dPCR jest kluczowym czynnikiem skracającym czas i obniżającym koszty wielu metod sekwencjonowania nowej generacji, umożliwiając tym samym osobistą genomikę.
Startery i sondy
Chociaż format reakcji i potrzebne oprzyrządowanie różnią się, dPCR wykorzystuje te same startery i sondy co qPCR.
Rysunek 2 przedstawia metodę działania sond podwójnie znakowanych (hydroliza), najczęściej stosowanej chemii do wykrywania.
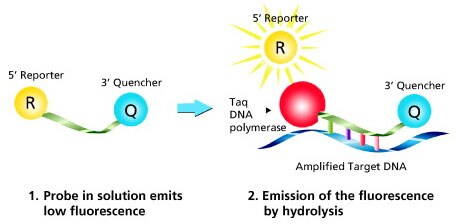
Rysunek 2. Mechanizm działania podwójnie znakowanych sond. Polimeraza DNA Taq wydłuża starter znajdujący się na tej samej nici co sonda, aż osiągnie pozycję sondy. Wrodzona aktywność egzonukleazy hydrolizuje sondę od 5' do 3', co uwalnia barwnik reporterowy do roztworu, a tym samym powoduje wzrost fluorescencji. Zmierzony sygnał fluorescencji jest wprost proporcjonalny do ilości docelowego DNA.
Analiza danych
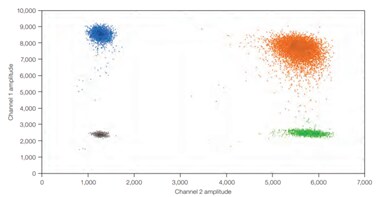
Zarówno PCR punktu końcowego, jak i qPCR wykrywają amplifikację docelowego zespołu cząsteczek kwasu nukleinowego obecnego w próbce, która jest jedną komorą reakcyjną (Rysunek 3). W dPCR próbka jest podzielona na wiele komór reakcyjnych (kropelek), z których każda ma lub nie ma jednej kopii docelowej cząsteczki kwasu nukleinowego (Rysunek 4).
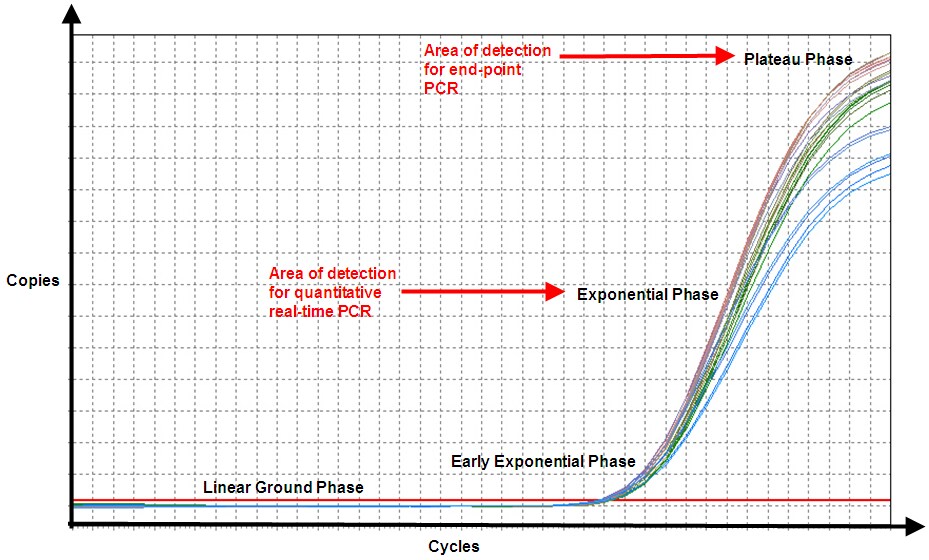
Rysunek 3. W reakcji PCR z punktem końcowym wykrywane są produkty reakcji z fazy plateau, a jak widać, repliki próbki (ta sama ilość początkowego kwasu nukleinowego) dają różne ilości produktu końcowego z powodu zmienności kinetyki reakcji. W qPCR, nawet jeśli wydajność nigdy nie wynosi 100%, produkt prawie podwaja się z każdym cyklem w fazie wykładniczej, a zatem reakcja jest bardziej specyficzna i precyzyjna (punkt wykrycia w qPCR jest określany przez linię progową, która nie jest pokazana).
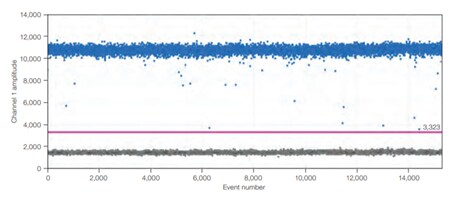
A)
B)
Rysunek 4.W dPCR stosunek reakcji pozytywnych do negatywnych jest wykorzystywany do obliczenia bezwzględnej ilości docelowej cząsteczki kwasu nukleinowego obecnej w oryginalnej próbce przed jej podziałem do analizy. Dlatego kinetyka reakcji nie ma znaczenia dla końcowej analizy. A) Przykładowa jednowymiarowa reakcja z jednym kompleksem. Czarne kropki to kropelki bez obecnego celu (nie wykryto fluorescencji), a niebieskie kropelki to kropelki z obecnym celem (wykryto fluorescencję). B) Przykładowa dwuwymiarowa, dwupleksowa reakcja. Czarne kropki to kropelki bez obecnego celu (brak wykrytej fluorescencji), niebieskie kropki to kropelki z obecnym pierwszym celem (wykryta fluorescencja z pierwszego barwnika reporterowego), zielone kropki to kropelki z obecnym drugim celem (wykryta fluorescencja z drugiego barwnika reporterowego), a pomarańczowe kropelki to kropelki z obecnymi oboma celami (wykryta fluorescencja z obu barwników reporterowych).
Studium przypadku
Znana firma zajmująca się narzędziami badawczymi w dziedzinie nauk przyrodniczych porównała wydajność naszych podwójnie znakowanych sond z różnymi zmianami konfiguracji, np. różnymi wygaszaczami, a także z sondami konkurencji w serii testów dPCR. Wszystkie eksperymenty zostały przeprowadzone na systemie Bio-Rad QX200 Droplet Digital™ PCR (ddPCR™).
Modyfikacje termiczne
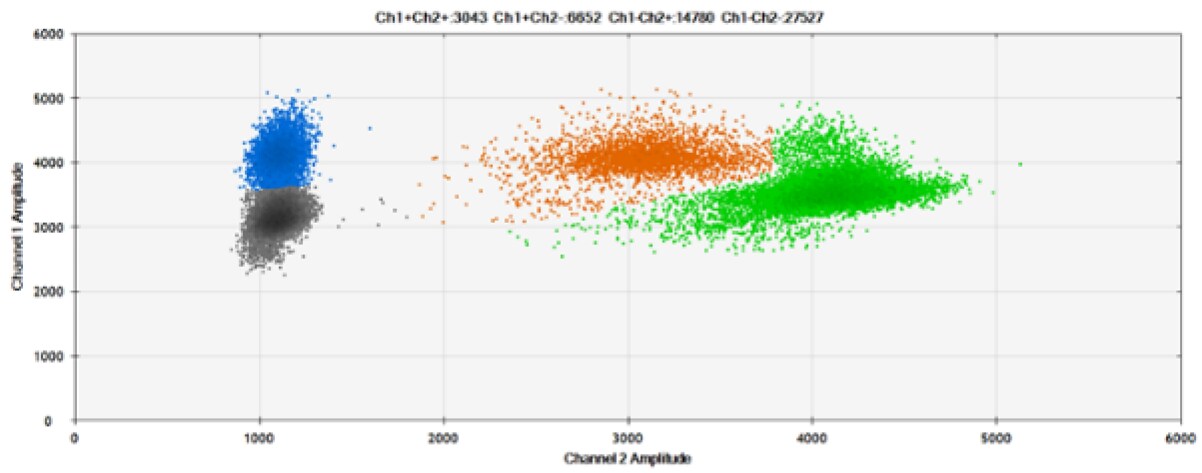
Pierwszy test (Rysunek 5) porównywał nasze sondy z i bez zablokowanego kwasu nukleinowego z sondami konkurenta znakowanymi inną modyfikacją termiczną, Minor Groove Binder (MGB).

A)

B)

C)
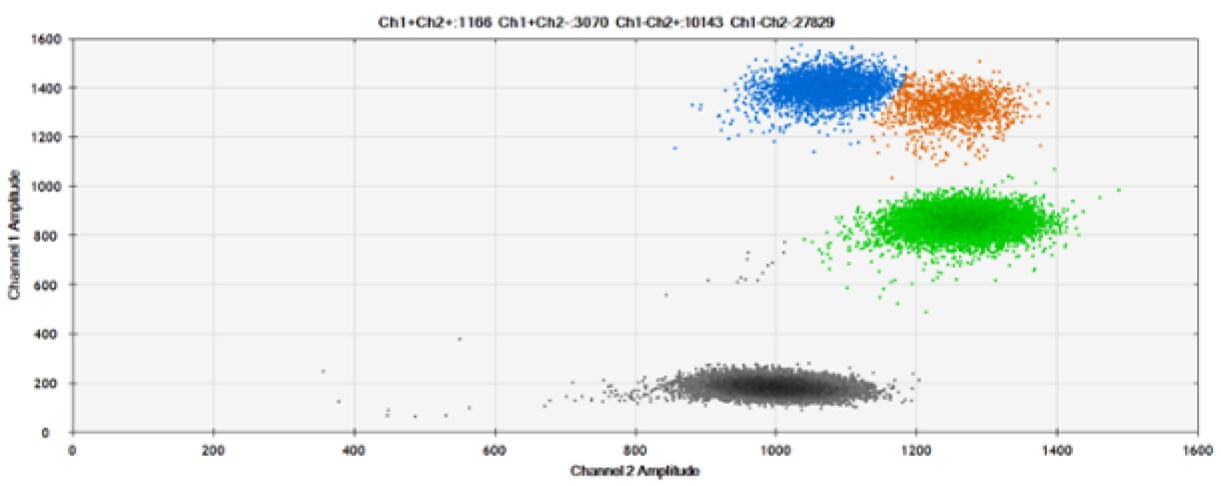
Rysunek 5. Wykrywanie typu dzikiego (L137) i zmutowanego (L137I) BRCA1. A Nasze sondy bez zablokowanego kwasu nukleinowego w reakcji dwupleksowej z typem dzikim i zmutowanym. B) Nasze sondy z zablokowanym kwasem nukleinowym w reakcji dwupleksowej z typem dzikim i mutantem. C) Konkurencyjne sondy z MGB w reakcji dwupleksowej z typem dzikim i mutantem. Czarne kropki to kropelki bez obecnego celu (brak wykrytej fluorescencji), niebieskie kropelki to kropelki z obecnym mutantem (6-FAM™/BHQ™-1, nasze sondy w A i B; 6-FAM/NFQ [wygaszacz niefluorescencyjny], sondy konkurencyjne), zielone kropki to kropelki z obecnością typu dzikiego (HEX™/BHQ™-1, nasze sondy; VIC™/NFQ, sondy konkurencyjne), a pomarańczowe kropelki to kropelki z obecnością zarówno mutanta, jak i typu dzikiego. Spadek sygnału w przypadku HEX wynika z faktu, że próbka została odczytana na kanale VIC.
Nasze sondy wykryły 113,3 kopii typu dzikiego i 343 zmutowane kopie na mikrolitr, podczas gdy sondy konkurencji wykryły odpowiednio 112,1 i 342. Równoważność naszych sond w porównaniu z sondami konkurencji jest oczywista, podobnie jak wymóg modyfikacji termicznej, takiej jak Zablokowany kwas nukleinowy, jest obecny w celu rozróżnienia między SNP.
HEX vs. ATTO-532
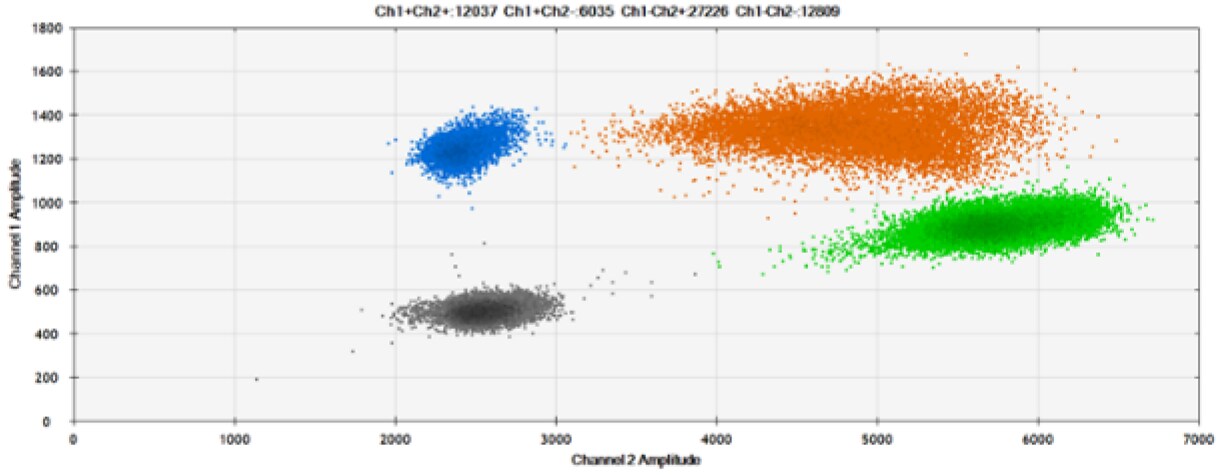
Drugi test (Rysunek 6, Tabela 2) porównał nasze sondy z barwnikiem reporterowym HEX i alternatywą dla HEX, ATTO-532.
.
A)
B)
C)
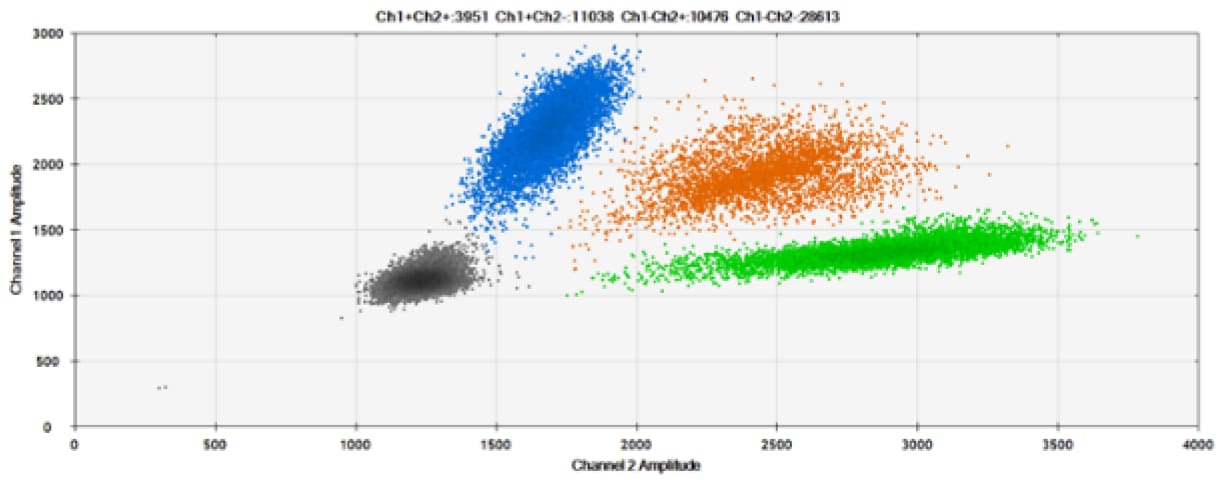
D)
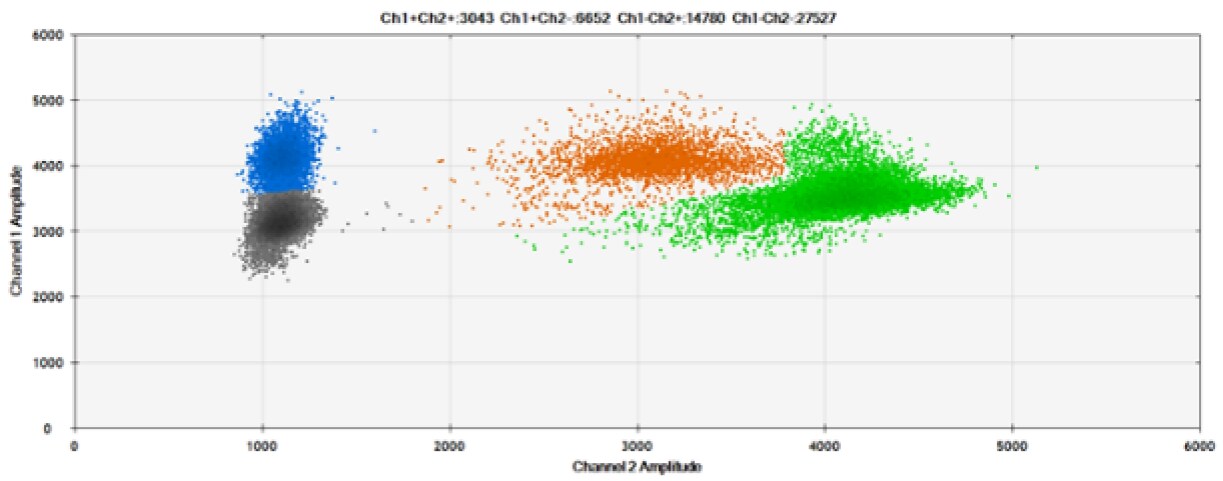
E)
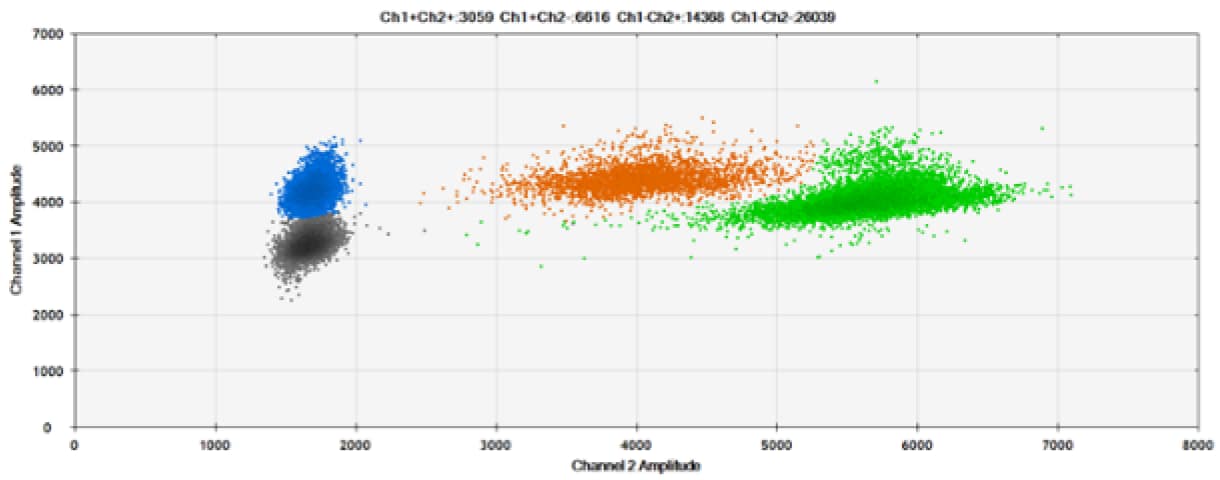
F)
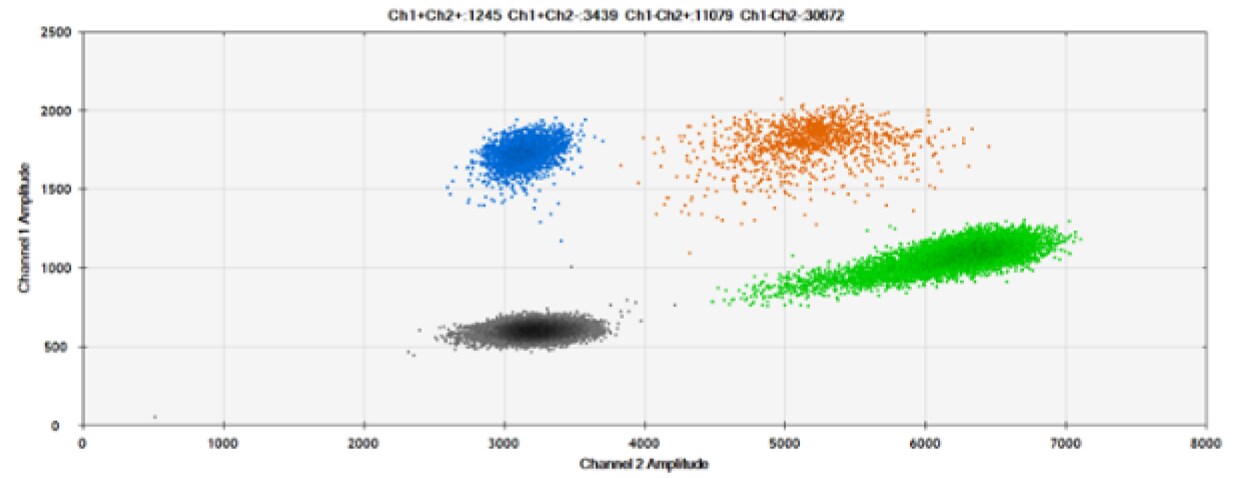
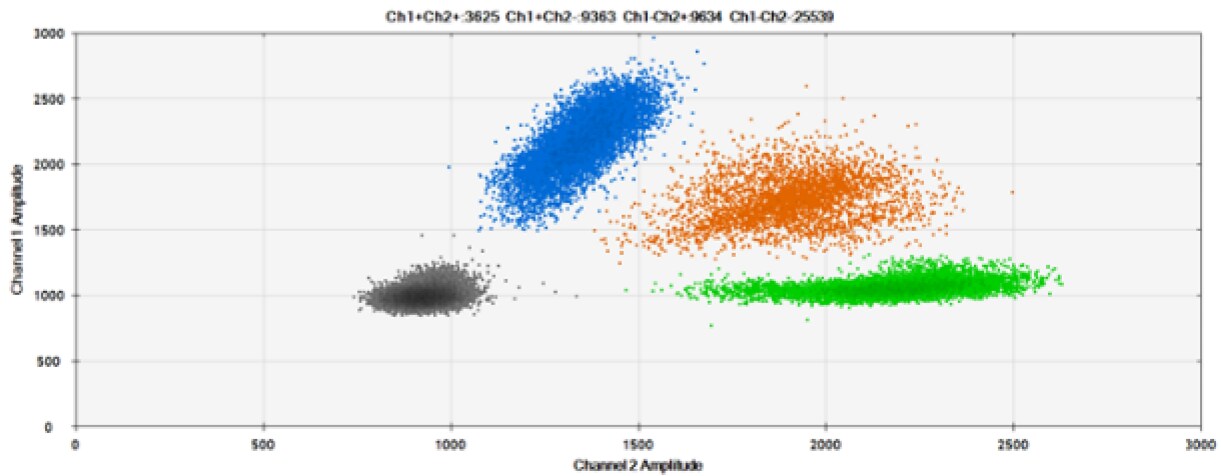
Rysunek 6. Wykrywanie BRCA1 L137I, ERBB2 A775_G776insYVMA i PDGFRA S566-E571>Rdel. A i B) Nasze sondy z HEX/BHQ™-1 i ATTO-532/BHQ™-1, odpowiednio, przeciwko BRCA1 L137I. C & D) Nasze sondy z HEX/BHQ™-1 i ATTO-532/BHQ™-1, odpowiednio, przeciwko ERBB2 A775_G776insYVMA. E i F) Nasze sondy z HEX/BHQ™-1 i ATTO-532/BHQ™-1, odpowiednio, przeciwko PDGFRA S566-E571>Rdel. Czarne kropki oznaczają kropelki bez obecności celu (brak wykrytej fluorescencji), niebieskie kropelki oznaczają kropelki z obecnością mutanta (6-FAM/BHQ™-1), zielone kropelki oznaczają kropelki z obecnością dzikiego typu (HEX/BHQ™-1 lub ATTO-532/BHQ™-1), a pomarańczowe kropelki oznaczają kropelki z obecnością zarówno mutanta, jak i dzikiego typu. Wszystkie sondy zawierają zablokowany kwas nukleinowy.
*Współczynnik zmienności
ATTO-532 wydaje się zapewniać podobną wydajność w porównaniu z HEX. Istnieje jednak ograniczona literatura z takimi porównaniami. Dlatego zalecamy, aby osoby zainteresowane wykorzystaniem ATTO-532 w dPCR przeprowadziły dodatkowe eksperymenty w celu ustalenia, czy spełnia on wymagania dotyczące wydajności.
Fluorescencyjny vs niefluorescencyjny wygaszacz
Trzeci test (Rysunek 7, Tabela 3) porównał nasze sondy z TAMRA i BHQ™-1 jako wygaszaczem, aby określić, czy jeden lub oba byłyby wystarczające w porównaniu z wydajnością konkurencyjnego NFQ (zwanego również DQ lub Dark Quencher).
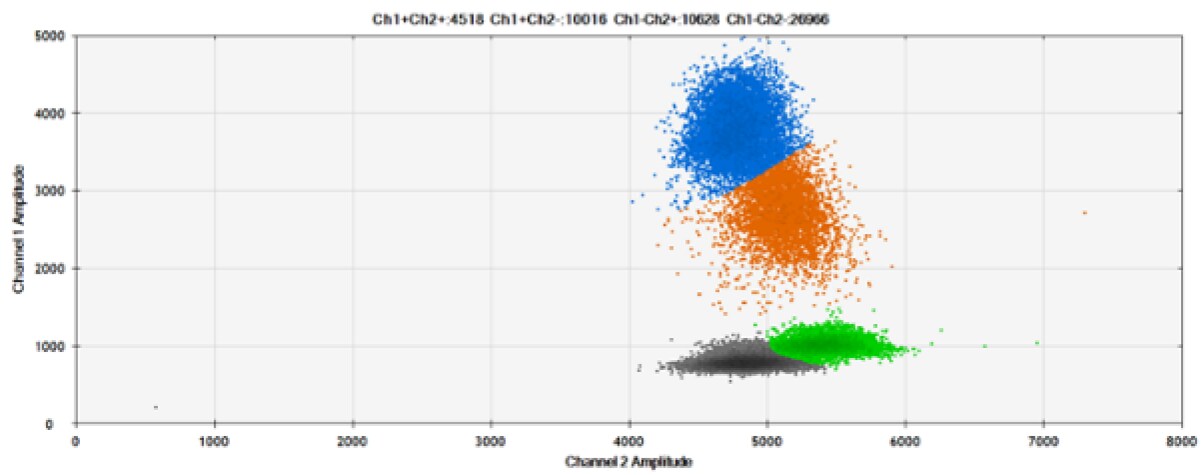
A)
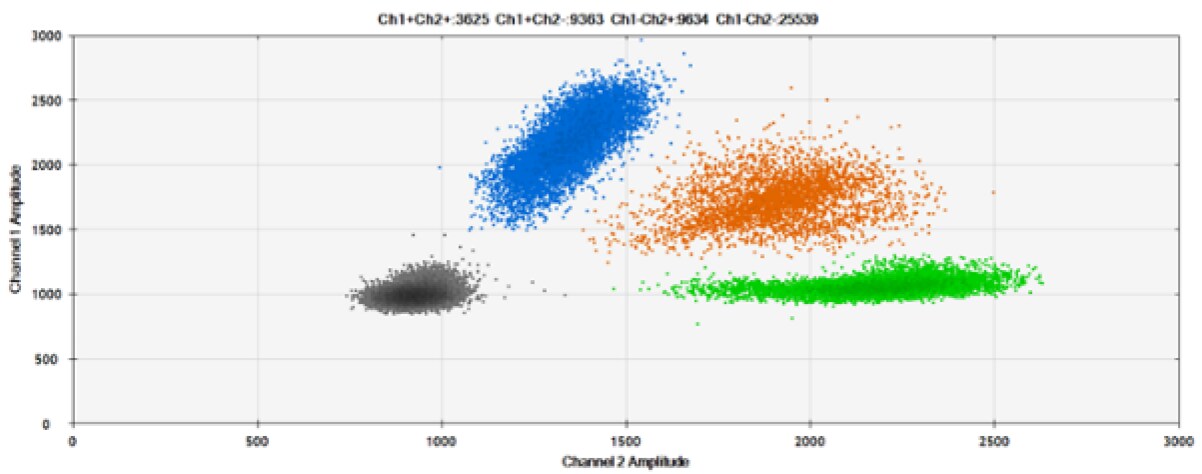
B)
C)
D)
Rysunek 7. Wykrywanie ERBB2 A775_G776insYVMA i PDGFRA S566-E571>Rdel. A i B) Nasze sondy z TAMRA i BHQ™-1, odpowiednio, przeciwko ERBB2 A775_G776insYVMA. C i D) Nasze sondy z TAMRA i BHQ™-1, odpowiednio, przeciwko PDGFRA S566-E571>Rdel. Czarne kropki to kropelki bez obecności celu (brak wykrytej fluorescencji), niebieskie kropki to kropelki z obecnością mutanta (6-FAM), zielone kropelki to kropelki z obecnością dzikiego typu (HEX), a pomarańczowe kropelki to kropelki z obecnością zarówno mutanta, jak i dzikiego typu. Wszystkie sondy zawierają zablokowany kwas nukleinowy.
*Współczynnik zmienności
Zgodnie z oczekiwaniami wynikającymi z opublikowanych eksperymentów qPCR i obserwowanymi tutaj, BHQ™-1 zapewnia lepszą separację sygnałów fluorescencji niż TAMRA w reakcjach dwupleksowych z 6-FAM i HEX.
Ciemny wygaszacz vs. Ciemny wygaszacz
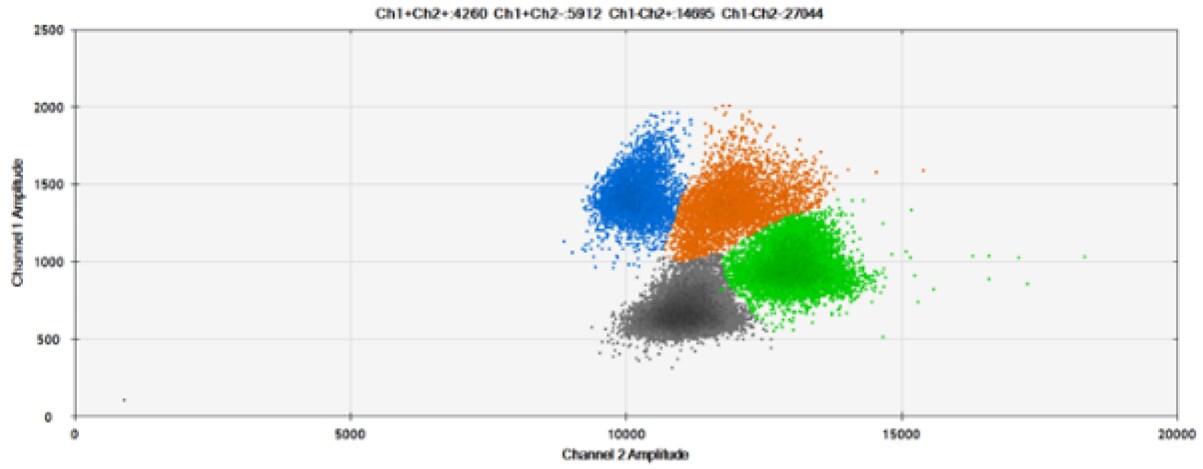
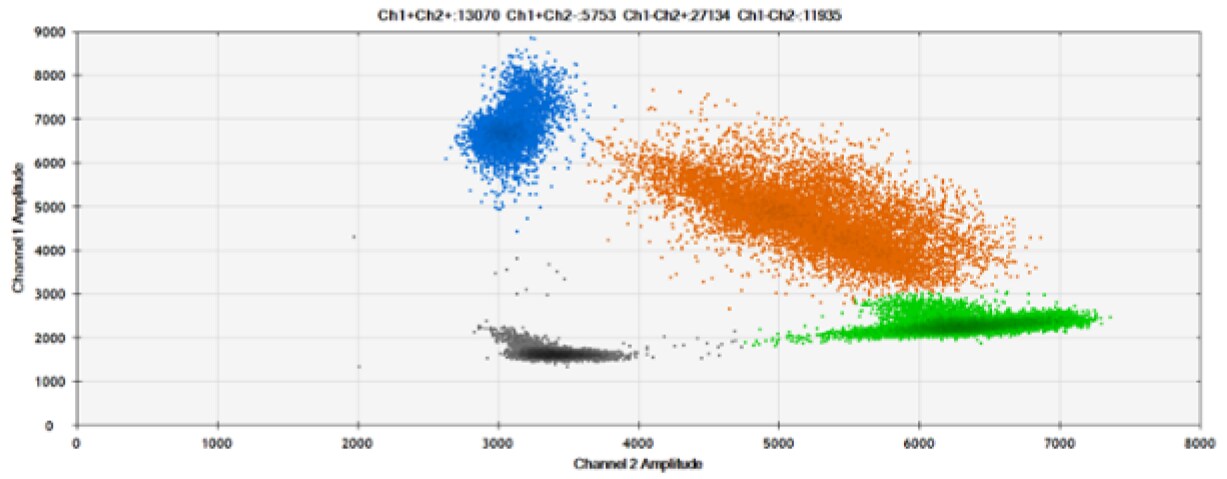
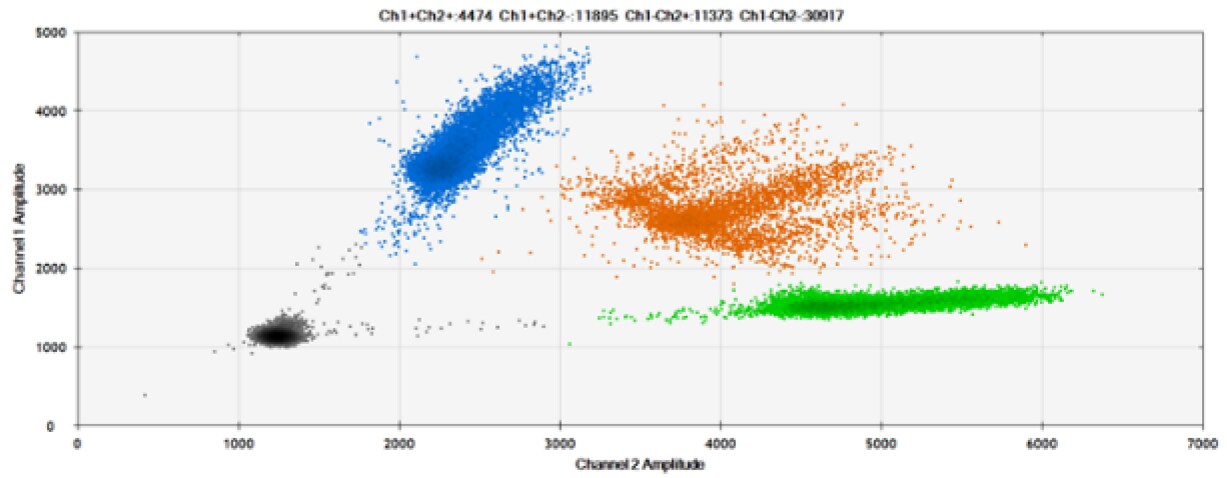
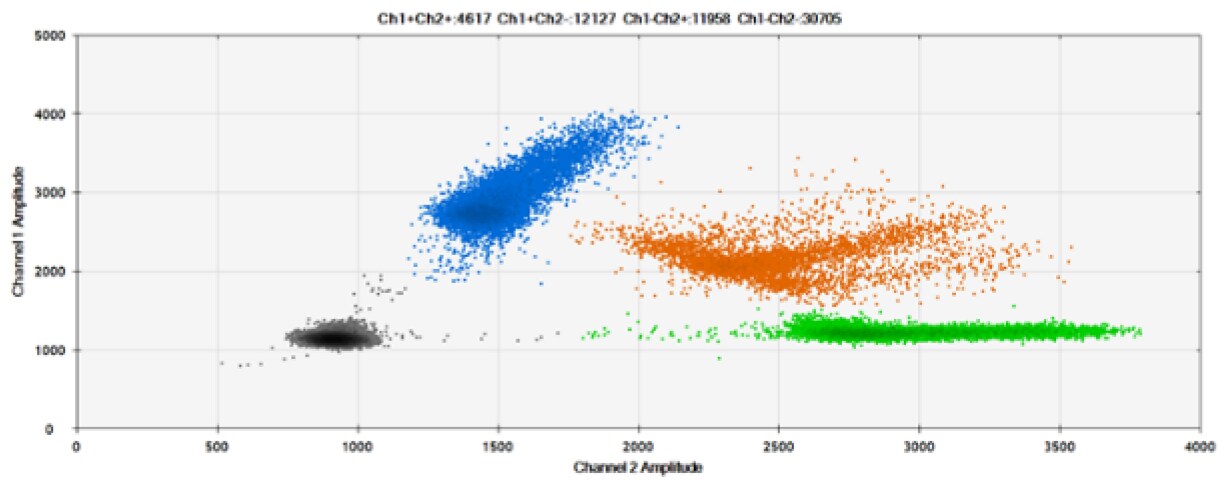
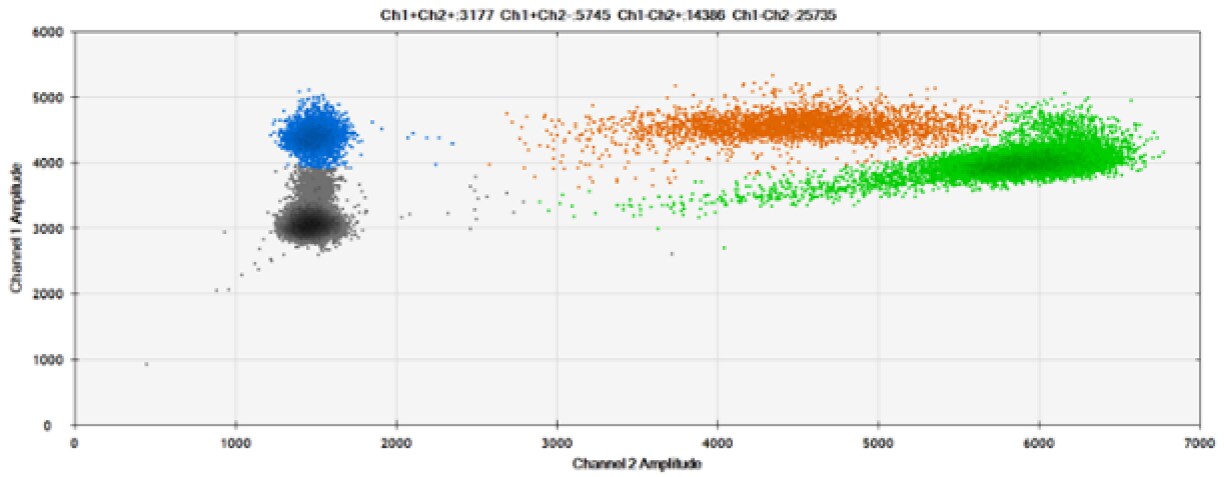
Czwarty test (Rysunek 8, Tabela 4) porównał nasze sondy z BHQ™-1 i OQ™ (Onyx Quencher™), nasza własna licencja i wolny od opłat licencyjnych niefluorescencyjny (ciemny) wygaszacz.
A)
B)
C)
D)
E)
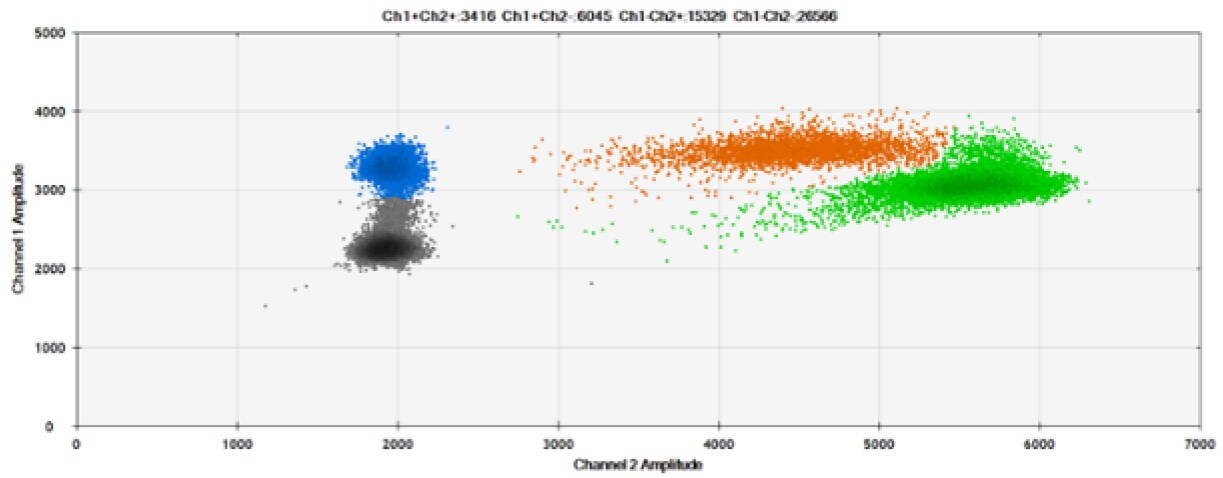
F)
Rysunek 8. Wykrywanie BRCA1 L137I, ERBB2 A775_G776insYVMA i PDGFRA S566-E571>Rdel. A i B) Nasze sondy z BHQ™-1 i OQA, odpowiednio, przeciwko BRCA1 L137I. C i D) Nasze sondy z BHQ™-1 i OQA, odpowiednio, przeciwko ERBB2 A775_G776insYVMA. E i F) Nasze sondy z BHQ™-1 i OQA, odpowiednio, przeciwko PDGFRA S566-E571>Rdel. Czarne kropki to kropelki bez obecności celu (brak wykrytej fluorescencji), niebieskie kropki to kropelki z obecnością mutanta (6-FAM), zielone kropelki to kropelki z obecnością dzikiego typu (HEX), a pomarańczowe kropelki to kropelki z obecnością zarówno mutanta, jak i dzikiego typu. Wszystkie sondy zawierają zablokowany kwas nukleinowy.
*Współczynnik zmienności
OQA zapewnia równoważną wydajność wygaszania w porównaniu z BHQ™-1. Biorąc pod uwagę, że OQ jest dostępny bez licencji i opłat licencyjnych, jest to atrakcyjna opcja do rozważenia, gdy celem jest komercjalizacja sond dPCR.
Wykrywanie SNP
Piąty i ostatni test (Rysunek 9 i powiązane tabele) porównał nasze sondy z sondami konkurenta do wykrywania SNP we wspólnych genach docelowych.
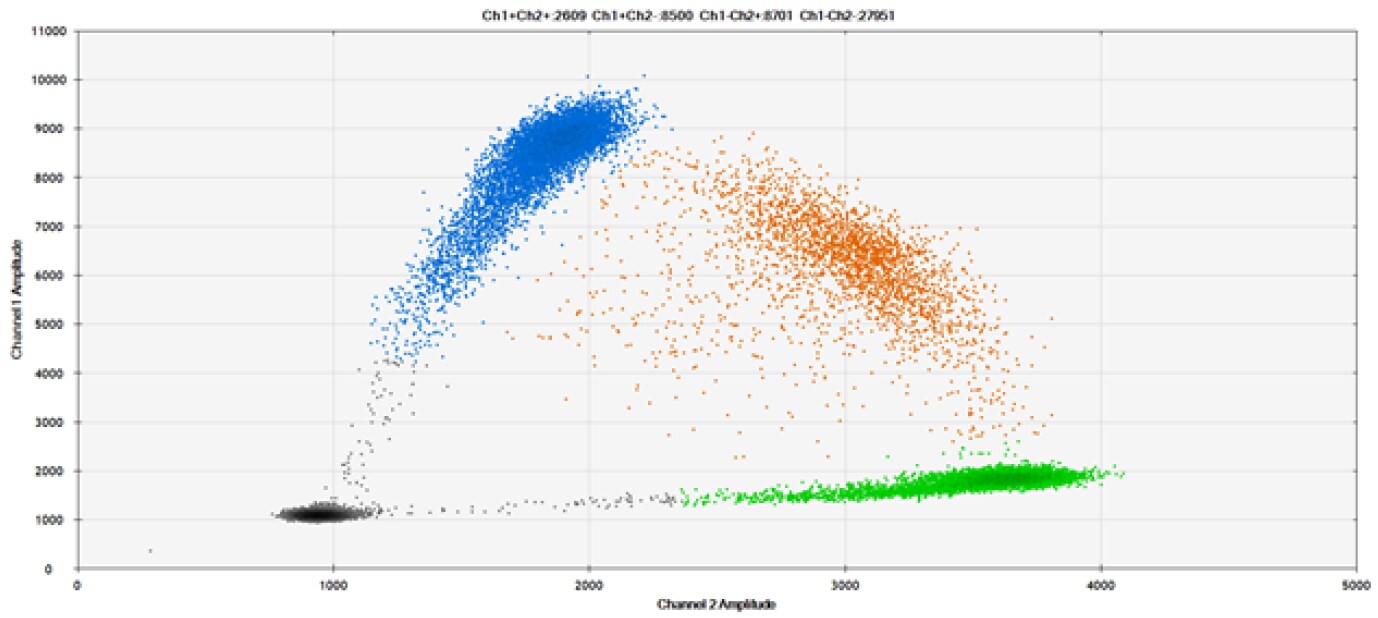
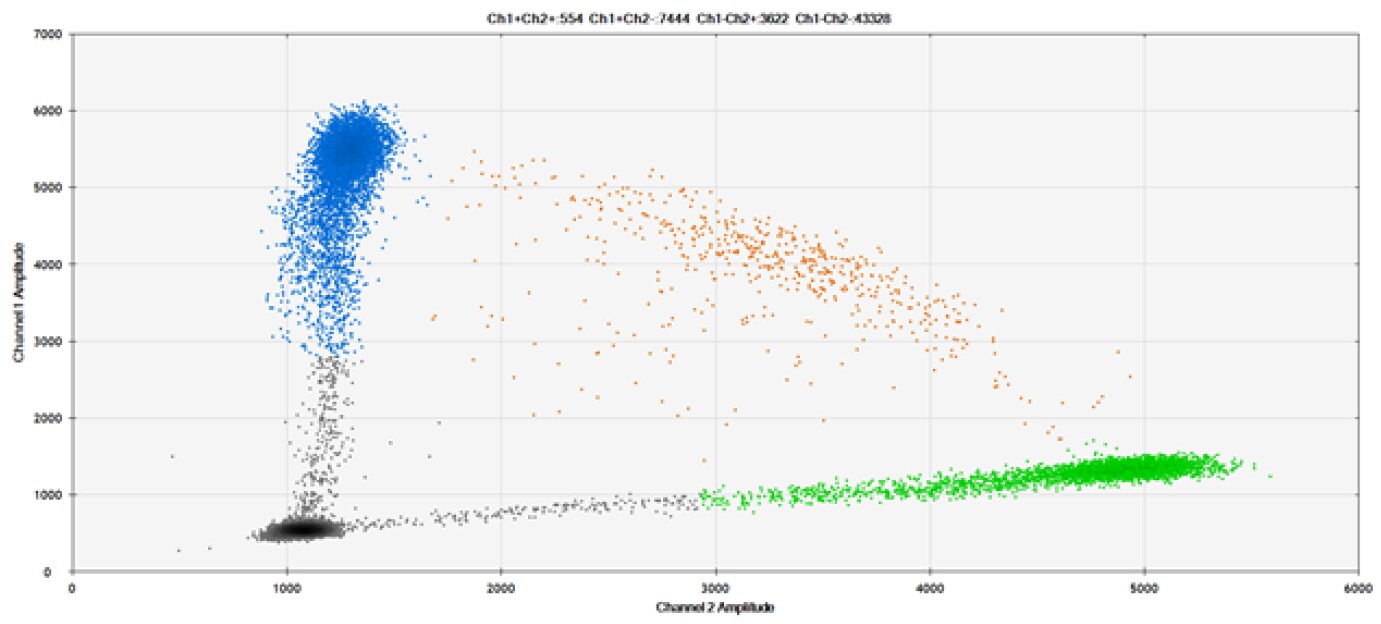
A)
*Współczynnik zmienności
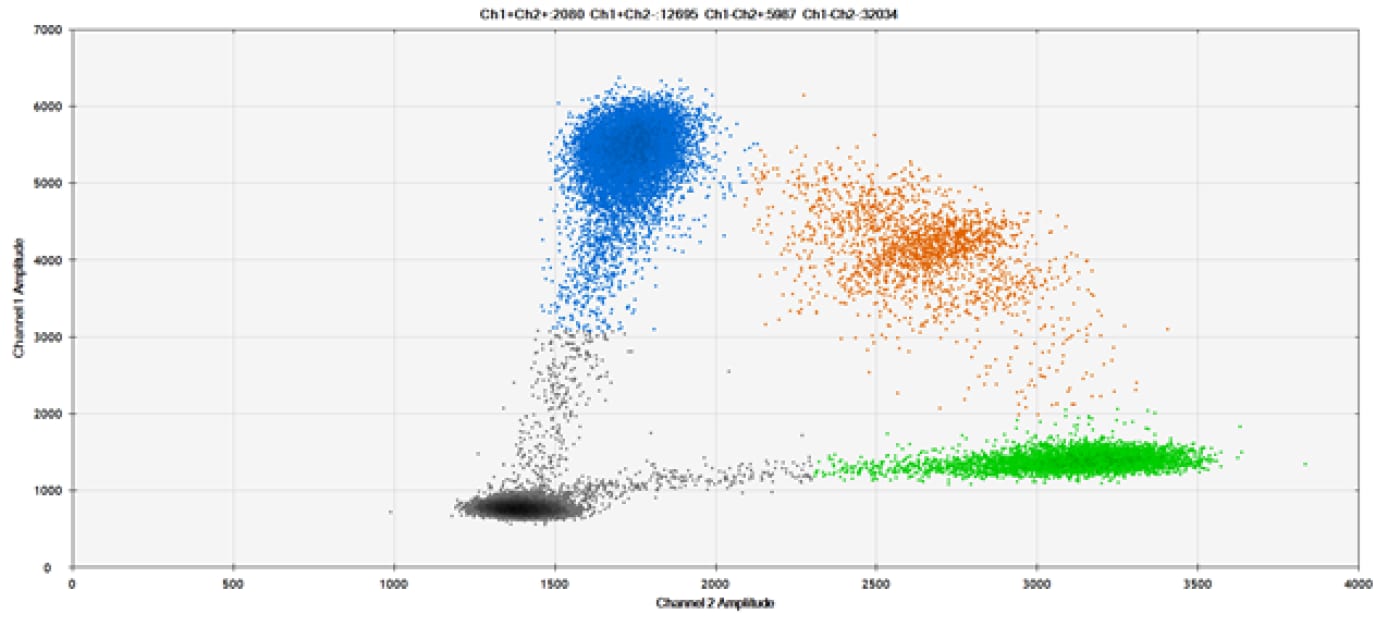
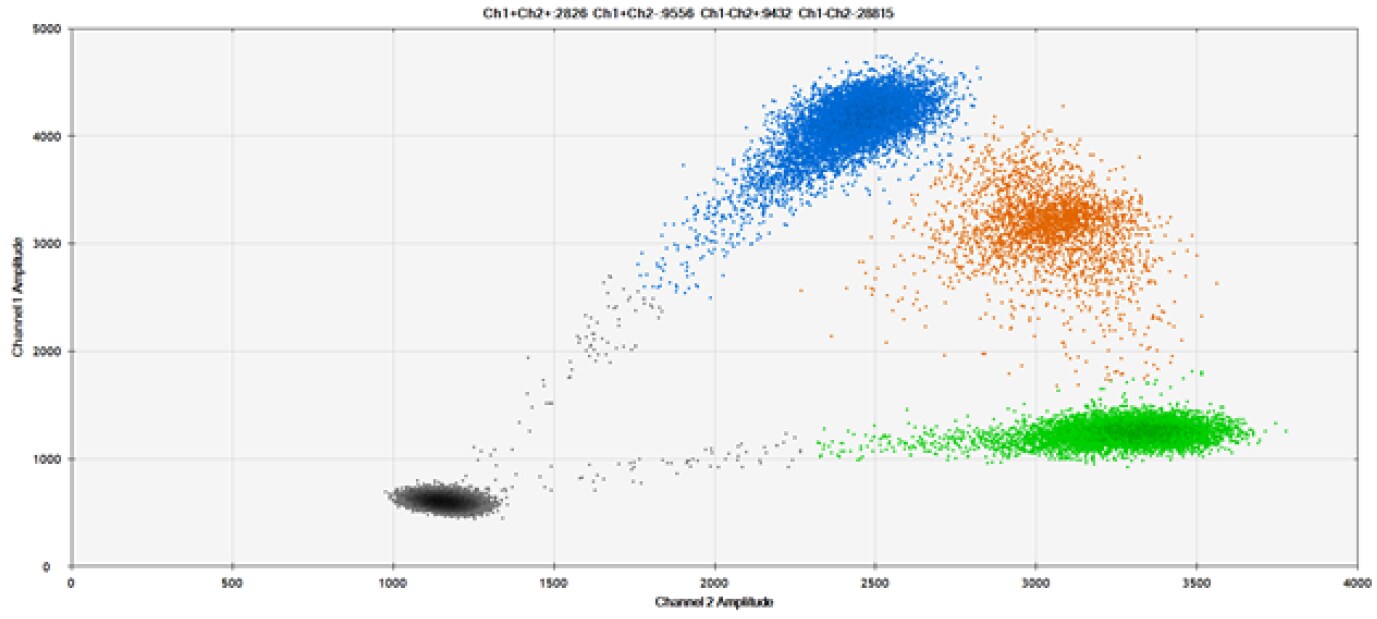
B)
*Współczynnik zmienności
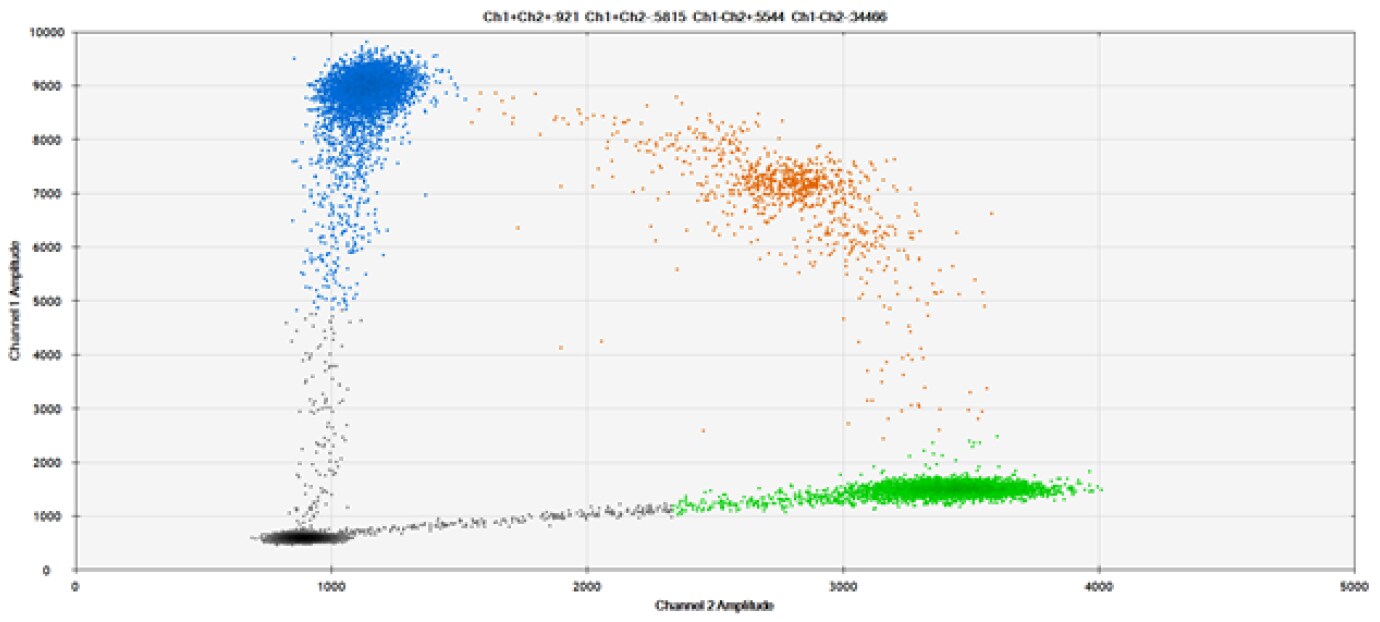
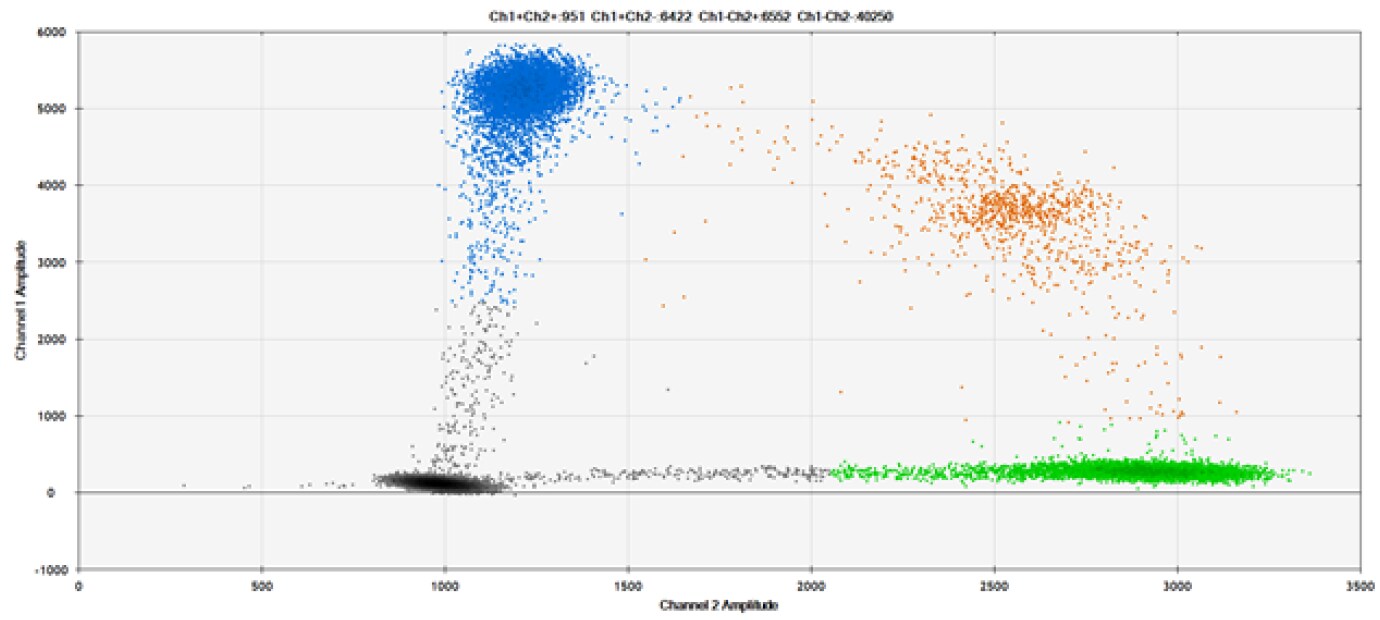
C)
*Współczynnik zmienności
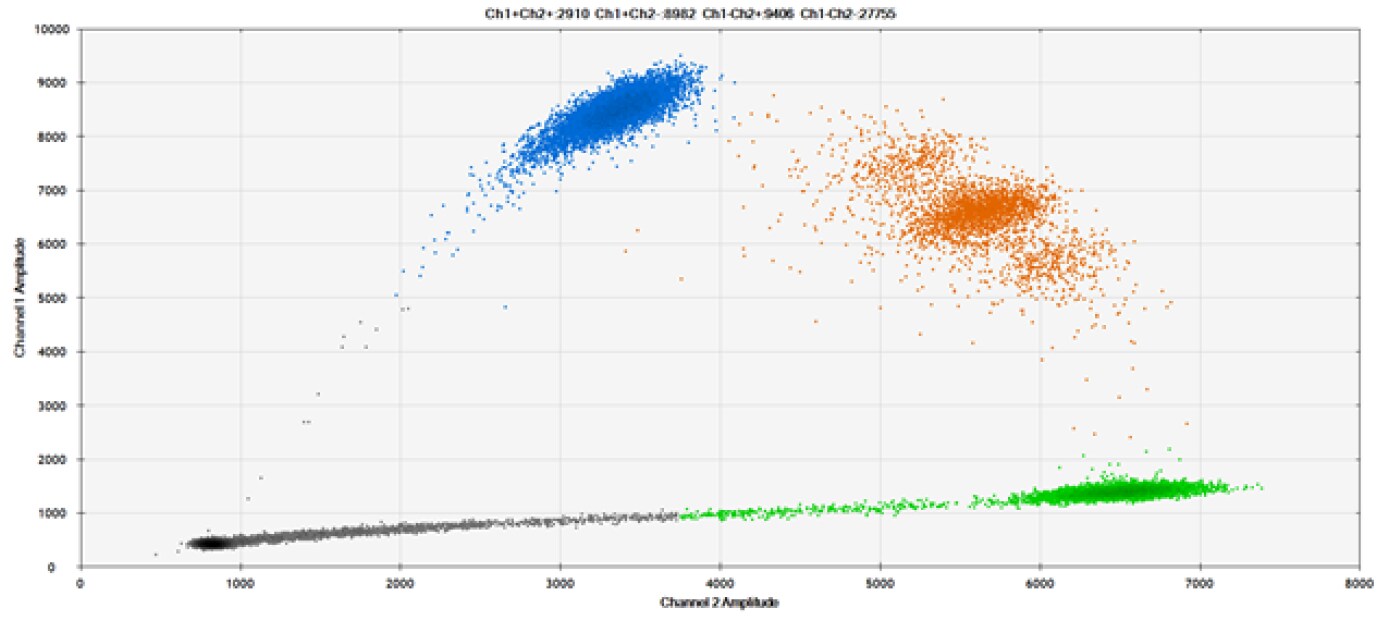
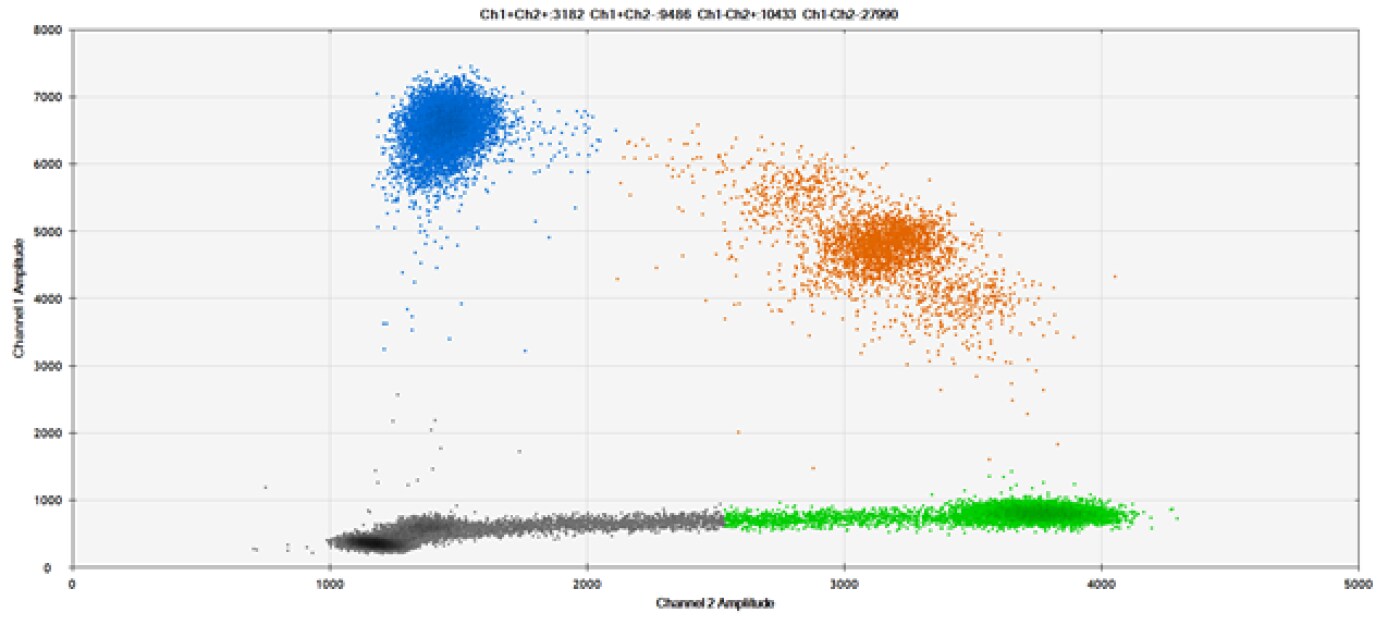
D)
*Współczynnik zmienności
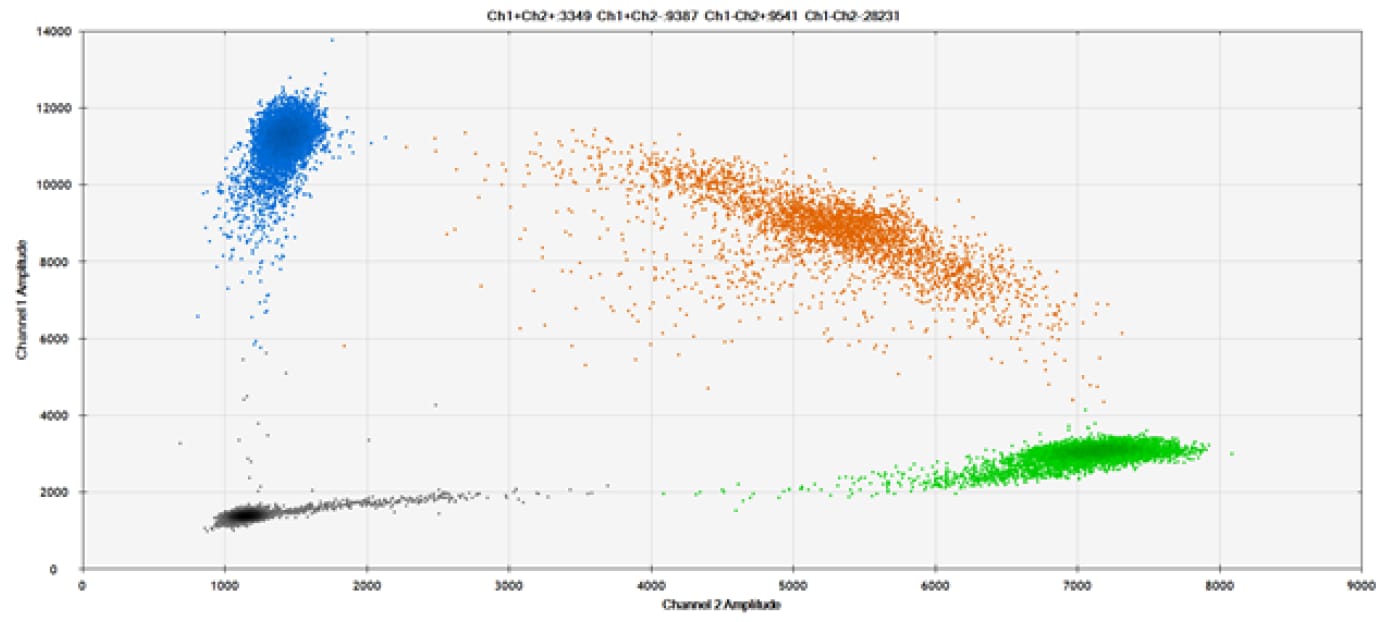
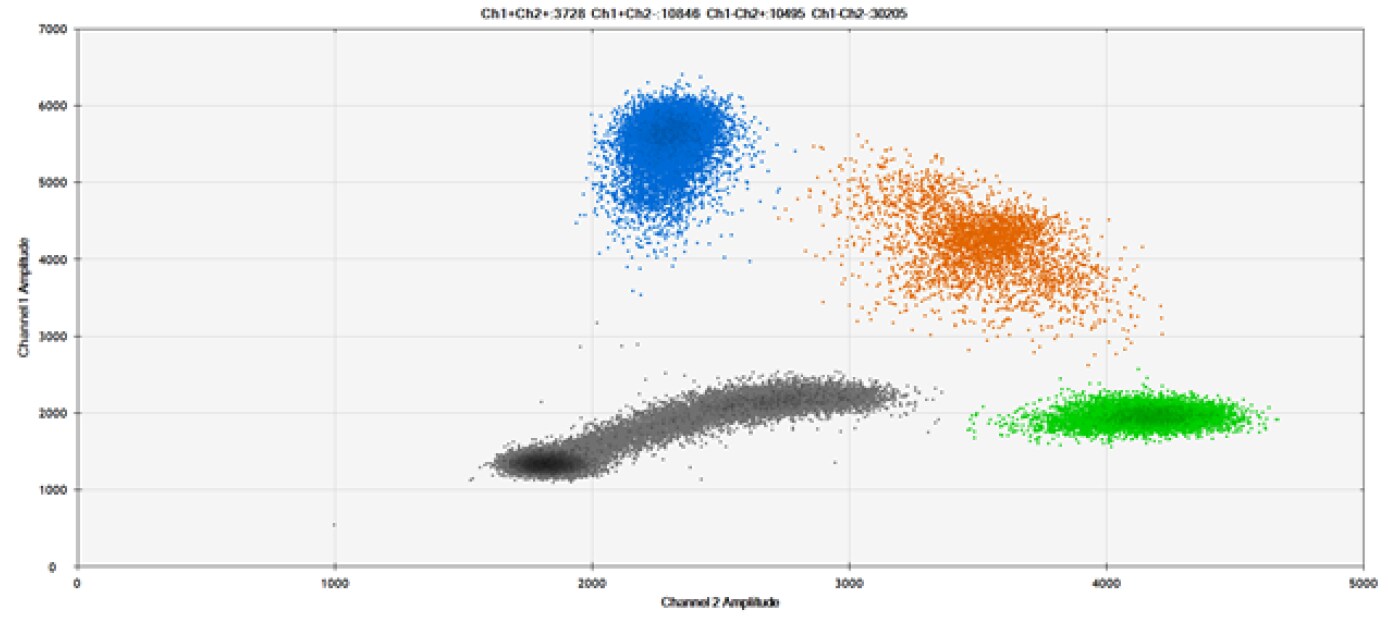
E)
Rysunek 9. Wykrywanie SNP w kilku popularnych genach docelowych. A) Nasze (górne) i konkurencyjne (dolne) sondy przeciwko AKT1 E17K B) Nasze (górne) i konkurencyjne (dolne) sondy przeciwko BRAF V600E C) Nasze (górne) i konkurencyjne (dolne) sondy przeciwko EGFR L858R D) Nasze (górne) i konkurencyjne (dolne) sondy przeciwko KRAS G12D E) Nasze (górne) i konkurencyjne (dolne) sondy przeciwko PIK3CA E545K. Tabele zawierają zmierzone stężenia i częstotliwość alleliczną (AF). Genomowe DNA ma 50% AF dla typu dzikiego i mutanta. Czarne kropki oznaczają kropelki bez obecności celu (brak wykrytej fluorescencji), niebieskie kropelki oznaczają kropelki z obecnością mutanta (6-FAM™/BHQ™-1, nasze sondy; 6-FAM/NFQ, sondy konkurencyjne), zielone kropelki oznaczają kropelki z obecnością typu dzikiego (ATTO-532/BHQ™-1, nasze sondy; VIC/NFQ, sondy konkurencyjne), a pomarańczowe kropelki oznaczają kropelki z obecnością zarówno mutanta, jak i typu dzikiego. Nasze sondy zawierają zablokowany kwas nukleinowy, a sondy konkurencyjne zawierają MGB.
*Współczynnik zmienności
Nasze sondy znakowane zablokowanym kwasem nukleinowym i alternatywnymi reporterami / wygaszaczami działają porównywalnie do sond popularnej marki.
Wnioski
Nasze podwójnie znakowane sondy działają dobrze w dPCR w porównaniu z popularną marką. Jeśli to możliwe, należy postępować zgodnie z tymi wskazówkami podczas konfigurowania eksperymentu:
- Zablokowany kwas nukleinowy jest wymagany do wykrywania SNP, ale może nie być konieczny do CNV (warianty liczby kopii) i analizy ekspresji genów
- ATTO-532 może być odpowiednim zamiennikiem HEX<
- TAMRA powinna być unikana jako wygaszacz
- OQ jest porównywalny do BHQ™ w wygaszaniu
Dostępne są zarówno opcje projektowania online, jak i konsultacyjne. Jeśli potrzebna jest dodatkowa pomoc, prosimy o kontakt z naszą grupą usług technicznych pod adresem oligotechserv@sial.com.
.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?