Kapa Biosystems Taq DNA Polymerase i zestawy do PCR
Odczynniki i zestawy polimerazy DNA Taq do ekstrakcji i amplifikacji DNA
Ekstrakcja i amplifikacja DNA są krytycznymi etapami w procesie biologii molekularnej i często wymagają oczyszczonego, wysokiej jakości DNA. Kapa Biosystems oferuje solidne portfolio odczynników polimerazy DNA i zestawów PCR do ekstrakcji i amplifikacji różnych materiałów genetycznych pochodzących od ssaków i roślin. Wykorzystując rewolucyjne termostabilne polimerazy, nasze odczynniki i zestawy Kapa Biosystems zostały zaprojektowane tak, aby umożliwić kolejne odkrycie z lepszą czułością i specyficznością dla wielu zastosowań.
Czytaj więcej o
KAPA Taq DNA Polymerase
Wysoka jakość i wydajność dla rutynowego PCR.
KAPA Taq DNA Polymerase jest oparta na pojedynczej podjednostce, dzikiej polimerazie Taq DNA termofilnej bakterii Thermus aquaticus. KAPA Taq i KAPA Taq HotStart DNA Polymerase mają aktywność polimerazy 5'-3' i egzonukleazy 5'-3', ale nie mają aktywności korekty egzonukleazy 3'-5'. Poziom błędu enzymu wynosi około 1 błędu na 2,2 x 105 włączonych nukleotydów.
Zestawy Taq DNA Polymerase firmy KAPA idealnie nadają się do:
- Standardowego PCR
- Znakowania DNA
- Sekwencjonowania DNA
- Licznych zastosowań, dla których wymagana jest wysokiej jakości, termostabilna polimeraza DNA
Korzyści obejmują:
- Ulepszona czułość, specyficzność i wydajność
- Nowa formuła buforu ułatwia specyficzne wyżarzanie starterów, prowadząc do wyższej wydajności określonego produktu
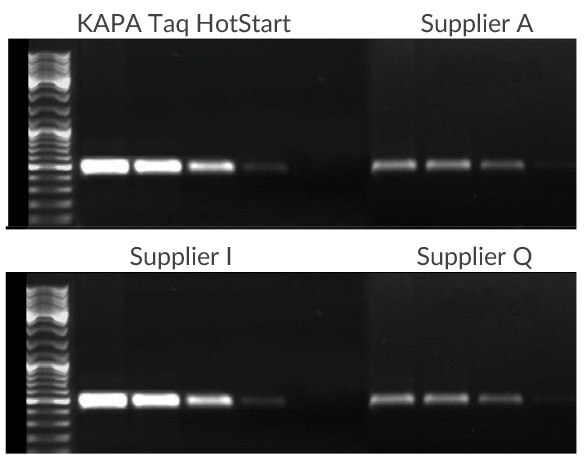
Rysunek 1.Fragment 500 bp genu CCR5 amplifikowano przy użyciu 100 ng, 10 ng, 1 ng lub 100 pg ludzkiego genomowego DNA jako matrycy. KAPA Taq HotStart wykazuje lepszą czułość, swoistość i wydajność w porównaniu z konkurencyjnymi produktami HotStart. Wszystkie reakcje zostały przeprowadzone przy użyciu protokołów zalecanych przez producenta.
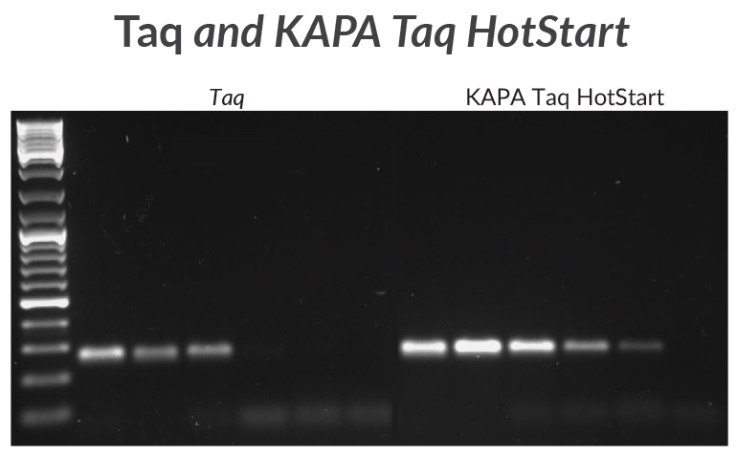
Rysunek 2.Amplikon 270 bp amplifikowano z DNA mykoplazmy za pomocą KAPA Taq lub KAPA Taq HotStart. Czułość testowano przy użyciu serii 10-krotnych rozcieńczeń matrycy, zaczynając od 1 ng DNA.
KAPA2G FAST MULTIPLEX
Szybki, wysokowydajny multipleksowy PCR.
Zestawy KAPA2G Fast Multiplex PCR zawierają enzym drugiej generacji (2G) uzyskany w procesie ukierunkowanej ewolucji. KAPA2G FAST HotStart DNA Polymerase to formuła gorącego startu za pośrednictwem przeciwciał, zaprojektowana z myślą o wyższej procesywności i szybkości, oferująca znacznie szybsze wydłużanie niż polimeraza Taq DNA typu dzikiego. Oprócz szybkości, KAPA2G Fast zapewnia wyższą wydajność i czułość niż konkurencyjne enzymy do wysoce multipleksowanej PCR.*
KAPA2G FAST Multiplex zapewnia:
- Jednolitą reprezentację wszystkich amplikonów
- Pomyślną multipleksową reakcję PCR z trudnymi, bogatymi w GC celami
- Skrócenie czasu cyklu PCR do 60%
- Skuteczną reakcję PCR z trudnymi, bogatymi w GC celami./li>
- Czas wydłużania nawet do 15 sekund
- Wysoka prędkość bez pogorszenia wydajności
- Minimalna optymalizacja składu mieszaniny wzorcowej
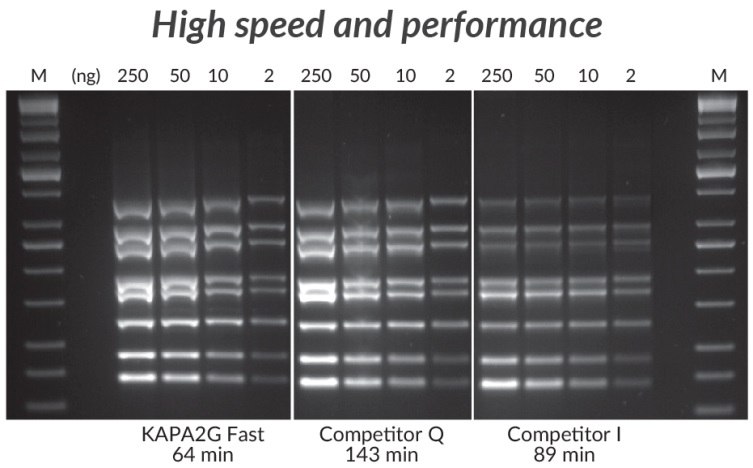
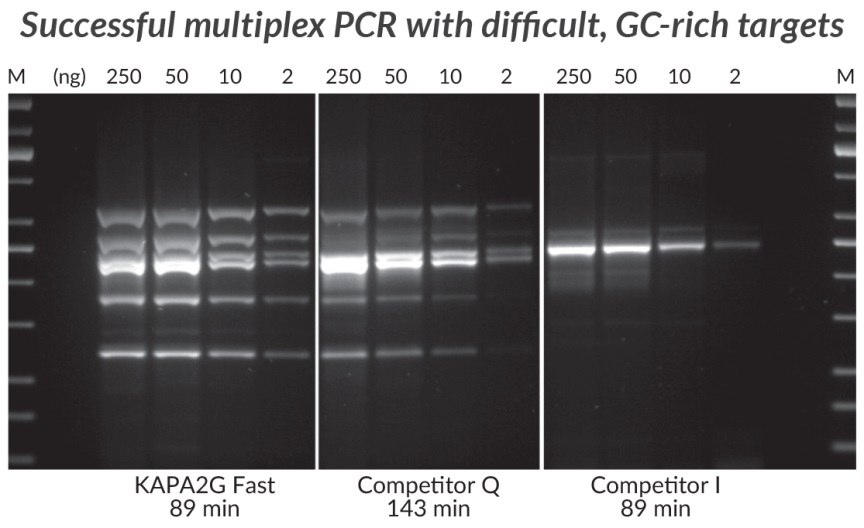
Rysunek 3 .*Reakcje (25 μL) zawierały 1X PCR Master Mix (KAPA i Competitor Q) lub 1X PCR Buffer, 3 mM MgCl2, 0,2 mM każdego dNTP i 1 U hot start Taq DNA Polymerase (domowe odczynniki multipleksowe, z Competitor I). Ludzkie genomowe DNA zostało użyte jako matryca (250 - 2 ng na reakcję), a startery zostały dostarczone w ilości 0,2 μM każdy. Cykle przeprowadzono zgodnie z zaleceniami producenta (30 cykli).
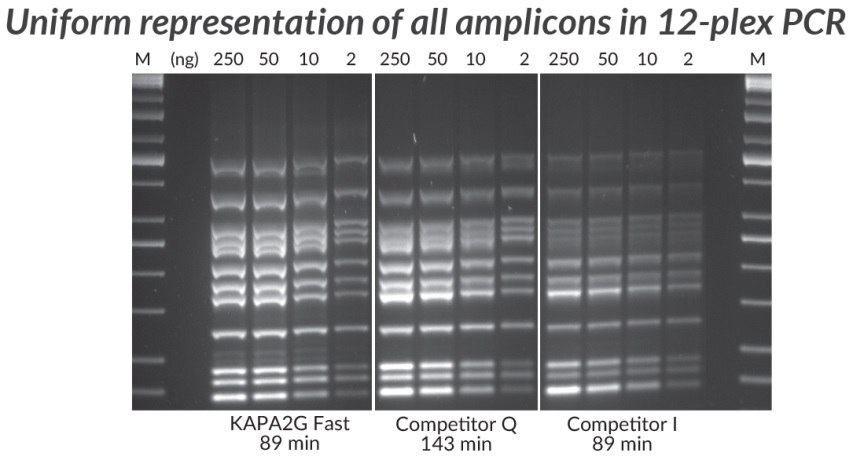
Rysunek 4.*Wielokrotna reakcja PCR (12-pleksowa) przeprowadzona przy użyciu zestawu KAPA2G Fast Multiplex PCR Kit, konkurenta Q i konkurenta I. Osiągnięcie jednolitej reprezentacji wszystkich amplikonów w złożonym teście multipleksowym jest wyzwaniem ze względu na tendencyjność amplifikacji - wynik różnic w długości amplikonu, strukturze drugorzędowej i wydajności primingu. Reakcje (25 μL) zawierały 1X PCR Master Mix (KAPA i Competitor Q) lub 1X PCR Buffer, 3 mM MgCl2, 0,2 mM każdego dNTP i 1 U hot-start Taq DNA Polymerase (domowe odczynniki multipleksowe, z Competitor I). Ludzkie genomowe DNA zostało użyte jako matryca (250 - 2 ng na reakcję), a startery zostały dostarczone w ilości 0,2 μM każdy. Cykle przeprowadzono zgodnie z zaleceniami producenta (30 cykli).
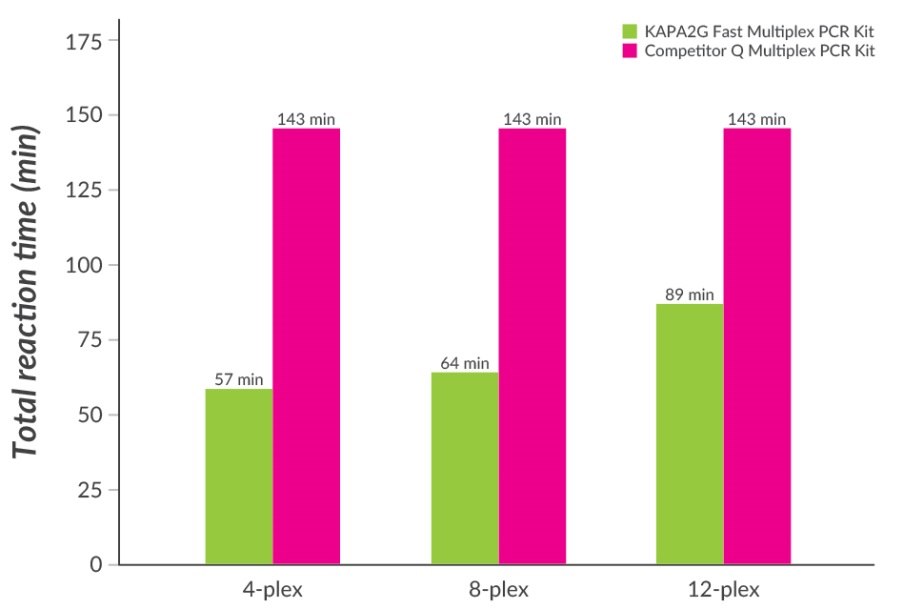
Rysunek 5.Szybki multipleksowy PCR z zestawami KAPA2G do szybkiego multipleksowego PCR. Multipleksowa reakcja PCR z użyciem Taq typu dzikiego zazwyczaj wymaga długich czasów wyżarzania i wydłużania, aby umożliwić wyżarzanie i wydłużanie wszystkich starterów w multipleksie. Przedstawiono całkowite czasy cykli PCR wymagane dla 4-pleksowych, 8-pleksowych i 12-pleksowych multipleksowych reakcji PCR (30 cykli, ustawionych zgodnie z zaleceniami producenta) z użyciem zestawów KAPA2G Fast Multiplex PCR i konkurencyjnego zestawu Q Multiplex PCR (który zawiera polimerazę Taq DNA typu dzikiego). Zestaw KAPA2G Fast Multiplex PCR Kit pozwala zaoszczędzić od 40 do 60% czasu.
Rysunek 6.GC-rich Multiplex PCR (6-plex) przeprowadzony przy użyciu zestawu KAPA2G Fast Multiplex PCR Kit, Competitor Q i Competitor I. Skuteczna multipleksowa reakcja PCR z Taq typu dzikiego jest ograniczona do łatwych, prostych celów, które można amplifikować z jednakową wydajnością. Ulepszona procesywność zmodyfikowanej szybkiej polimerazy DNA KAPA2G umożliwia jednolity multipleksowy PCR szerokiego zakresu trudnych celów. Reakcje (25 μL) zawierały 1X PCR Master Mix (KAPA i Competitor Q) lub 1X PCR Buffer, 3 mM MgCl2, 0,2 mM każdego dNTP i 1 U hot start Taq DNA Polymerase (domowe odczynniki multipleksowe, z Competitor I). Ludzkie genomowe DNA zostało użyte jako matryca (250 - 2 ng na reakcję), a startery zostały dostarczone w ilości 0,2 μM każdy. Do wszystkich reakcji dodano DMSO (5%) i KAPA Enhancer 1 (1X). Cykle przeprowadzono zgodnie z zaleceniami producenta (30 cykli). Wielkość amplikonów wynosiła od 241 do 642 bp, a zawartość GC od 72,7 do 83,8%.
KAPA2G ROBUST DNA POLYMERASE
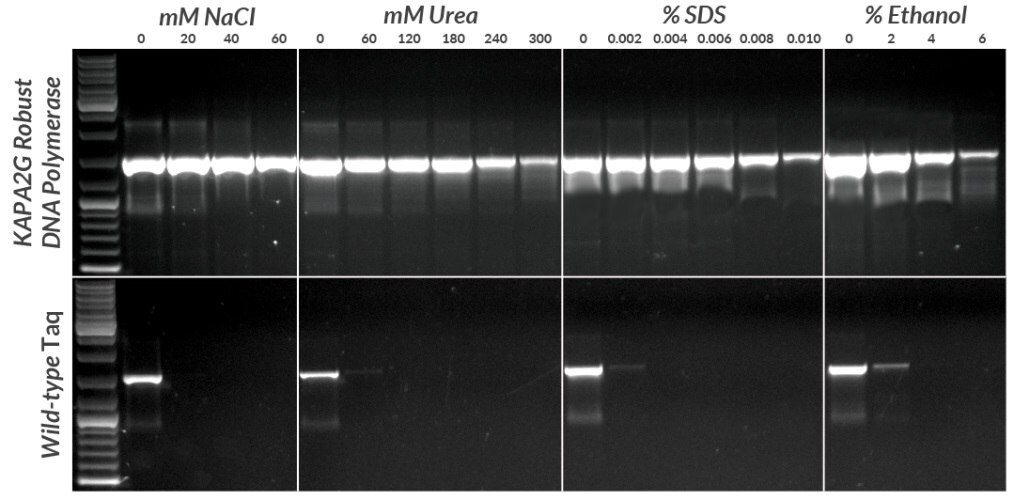
KAPA2G Robust DNA Polymerase została opracowana w celu rozwiązania problemu niespójnej amplifikacji w szerokim zakresie typów amplikonów (bogatych w GC i AT). Produkt ten zapewnia wyższą procesywność i specyficzną aktywność, co przekłada się na solidną wydajność PCR, wysoką czułość i lepszą tolerancję na typowe inhibitory. Ten wysokowydajny zestaw idealnie nadaje się do wymagających zastosowań PCR i trudnych próbek, eliminując potrzebę optymalizacji przy użyciu wielu enzymów i protokołów.
KAPA2G Robust DNA Polymerase zapewnia:
- Aplikacje wymagające wyższej wydajności na jednostkę enzymu
- Colony PCR
- Tolerancję na przenoszenie inhibitorów i surowe próbki PCR (np, FFPE)
- Rutynowa PCR przy użyciu formuły HotStart lub Readymix
Wydajna amplifikacja celów bogatych w GC i AT:
- Solidna wydajność w szerokim zakresie szablonów bogatych w GC i AT
- Zwiększony wskaźnik powodzenia PCR
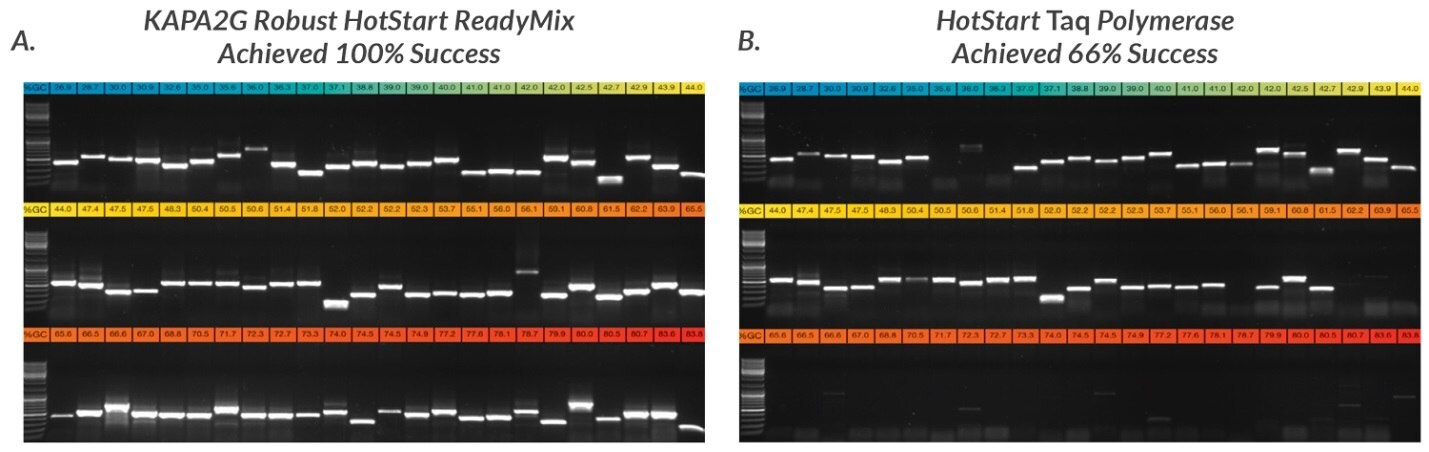
Rysunek 7.Połowę każdego z produktów PCR uzyskanych z 72 z 96 zestawów starterów użytych w tym badaniu poddano elektroforezie w 1% żelu TBE-agarozowym. Amplikony były ładowane w kolejności rosnącej zawartości GC, z najniższą zawartością GC (27%, niebieski) w lewym górnym rogu i najwyższą zawartością GC (84%, czerwony) w prawym dolnym rogu każdego złożonego obrazu żelu. Startery wybrane do tego badania miały zmienną długość, skład sekwencji, teoretyczne temperatury topnienia i inne cechy konstrukcyjne. Niektóre startery zawierały 5'-ogony do sekwencjonowania po PCR przy użyciu M13 lub innych standardowych starterów do sekwencjonowania. A. Reakcje KAPA2G Robust HotStart ReadyMix (25 μL) przeprowadzono zgodnie z instrukcją obsługi. B. Reakcje Taq typu dzikiego (25 μL, zawierające 0,5 U Taq na reakcję) przeprowadzono w buforze reakcyjnym Taq (1,5 mM MgCl2 przy 1X), stosując takie same końcowe stężenia starterów i dNTP jak dla KAPA2G Robust.
Wszystkie reakcje zawierały 25 ng ludzkiego genomowego DNA. 5% DMSO było zawarte we wszystkich reakcjach (KAPA2G Robust i Taq) ukierunkowanych na amplikony o zawartości GC >70%.
Zwiększona tolerancja na powszechne inhibitory PCR
- Wydajna amplifikacja z surowych próbek
- Wyższa wydajność i czułość na jednostkę enzymu
Rysunek 8.Amplifikacja fragmentu 1,5 kb z 1 pg plazmidowego DNA w obecności czterech powszechnie stosowanych inhibitorów PCR przy użyciu zestawu KAPA2G Robust HotStart PCR Kit i polimerazy Taq typu dzikiego. Wszystkie reakcje zawierały 0,5 jednostki enzymu na 25 μL reakcji. KAPA2G Robust HotStart Buffer B był używany przez cały czas, z dodatkiem KAPAEnhancer 1 dla reakcji zawierających SDS. Cyklowanie przeprowadzono za pomocą Eppendorf Mastercycler epgradient S, stosując standardowy 3-stopniowy profil cykliczny (35 cykli) z temperaturą wyżarzania 64ºC i 1,5 minuty czasu wydłużania na cykl dla wszystkich enzymów.
Niezrównana wydajność w PCR kolonii
- Wyższa wydajność i lepsza spójność PCR bezpośrednio z komórek E. coli i drożdży
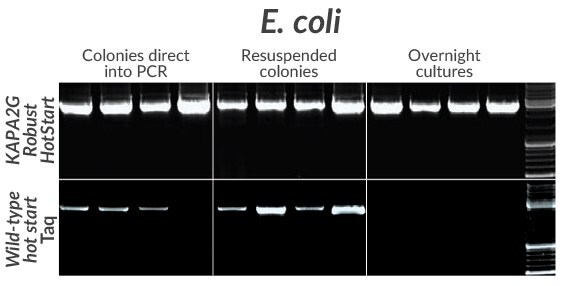
Rysunek 9.Amplifikacja sklonowanego insertu 2,7 kb z czterech powszechnie stosowanych szczepów E. coli (DH5a, DH10B, JM109 lub BL21) przy użyciu KAPA2G Robust HotStart (u góry) lub dzikiego Taq (u dołu). Kolonie (wyhodowane na płytkach LB-agar + Amp) zawieszono bezpośrednio w poszczególnych reakcjach PCR (po lewej) lub najpierw zawieszono w wodzie o czystości PCR, a następnie dodano do mieszanin reakcyjnych PCR (w środku). W przypadku hodowli nocnych (przygotowanych w LB + Amp), 1 μl dodawano bezpośrednio do mieszaniny PCR (po prawej).
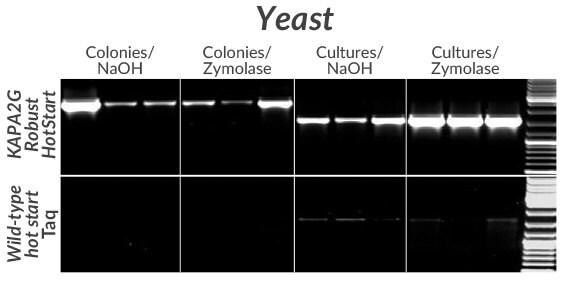
Rysunek 10.Amplifikacja fragmentu 2,5 kb (po lewej) lub 1,6 kb (po prawej) genu GSH1 z trzech powszechnie stosowanych szczepów S. cerevisiae (BY4742, FY23 i W303) przy użyciu KAPA2G Robust HotStart (u góry) lub dzikiego Taq (u dołu). Kolonie (z płytek YPD-agar) lub całonocne hodowle YPD były najpierw lizowane w objętości 50 μl za pomocą NaOH lub zymolazy (jak wskazano).
KAPA LONG RANGE PCR SYSTEM
KAPA Long Range PCR system to mieszanka polimerazy Taq DNA i zmodyfikowanej archealnej (z rodziny B) polimerazy DNA posiadającej zdolność korekty. Ten dwuskładnikowy system został zaprojektowany specjalnie do wspomagania PCR dalekiego zasięgu i/lub czułego PCR i charakteryzuje się 3-4-krotnie wyższą wiernością niż polimeraza Taq. W formule hotstart, KAPA Long Range jest połączona z opatentowanym przeciwciałem, które inaktywuje enzym do pierwszego etapu denaturacji, eliminując fałszywe produkty amplifikacji oraz zwiększając wydajność i czułość reakcji.
Zestawy KAPA Long Range PCR idealnie nadają się do:
- Amplifikacji PCR długich celów i/lub PCR przy użyciu małych ilości matrycowego DNA
- Standardowej amplifikacji PCR krótkiego i średniego zasięgu
- Produkcji produktów PCR, które mają być wykorzystane do ligacji w wektorach klonujących 3'-T-overhang
Korzyści obejmują:
- Amplifikacja celów z wysoką czułością i specyficznością do 15 kb
- Wysoka wydajność przy niskim wejściowym DNA
- Wykazuje wysoką wydajność, większą czułość i specyficzność tam, gdzie konkurencyjne systemy dalekiego zasięgu zawodzą*
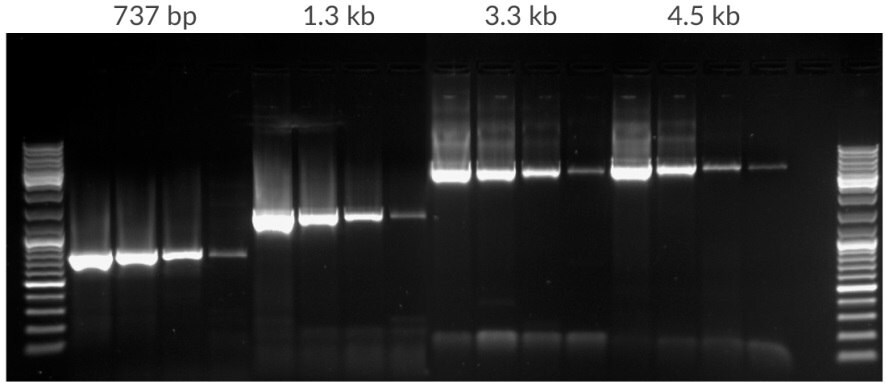
Rysunek 11.Amplifikacja rozcieńczenia matrycy ludzkiego genomowego DNA począwszy od 50 ng, 10 ng, 2 ng, 400 pg na 25 μL. Amplikony miały długość 737 bp, 1,3 kb, 3,3 kb i 4,5 kb. 35 cykli, temperatura wydłużania 72ºC.
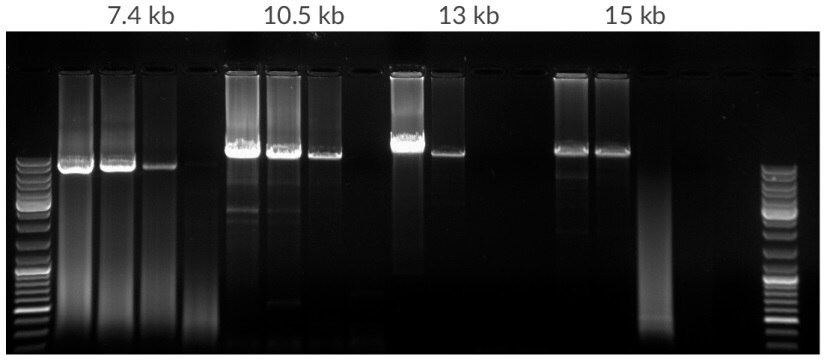
Rysunek 12.Amplifikacja rozcieńczenia matrycy ludzkiego genomowego DNA począwszy od 50 ng, 10 ng, 2 ng, 400 pg na 25 μL. Amplikony miały długość 7,5 kb, 10,5 kb, 13 kb i 15 kb. 35 cykli, temperatura wydłużania 68ºC.
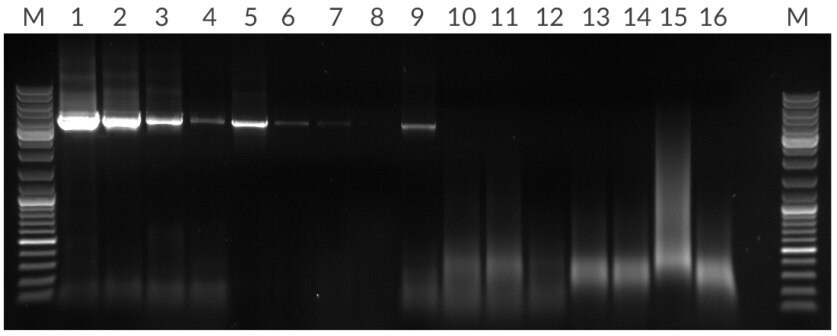
Rysunek 13.*Amplifikacja fragmentu 4,5 kb z 50 ng, 10 ng, 2 ng i 400 pg ludzkiego genomowego DNA na 25 μL reakcji. Pasy: (M) Marker, (1 - 4) KAPA Long Range HotStart, (5 - 8) Konkurent T, (13 - 16) Konkurent R.
KAPA MOUSE GENOTYPING
Przejdź od ogona do typu w 1 godzinę.
Zestawy do genotypowania myszy KAPA są przeznaczone do niezawodnej ekstrakcji i amplifikacji fragmentów DNA z tkanek myszy w ciągu zaledwie 1 godziny, w porównaniu do ≥ 1 dnia w przypadku konwencjonalnych protokołów. Zestawy obejmują KAPA Express Extract, nową termostabilną proteazę i system buforowy, który pozwala na ekstrakcję DNA gotowego do PCR z tkanki myszy w zaledwie 15 minut, oraz KAPA2G Fast Genotyping Mix z barwnikiem, zawierający polimerazę DNA zaprojektowaną w drodze ukierunkowanej ewolucji w celu uzyskania wysokiej procesywności i ekstremalnej prędkości.
Zestawy do genotypowania myszy KAPA zapewniają:
- 15-minutową ekstrakcję eliminującą rozpuszczalniki organiczne, neutralizację pH i wirowanie
- 45-minutową amplifikację z wysoką procesywnością i ekstremalną szybkością
- Wygodny format mieszaniny wzorcowej z barwnikiem ładującym zarówno w formulacjach z gorącym startem, jak i bez gorącego startu
Przewyższa surowe metody ekstrakcji i zestawy komercyjne
- Zwiększony czas realizacji i specyficzność
- Zmniejszony koszt

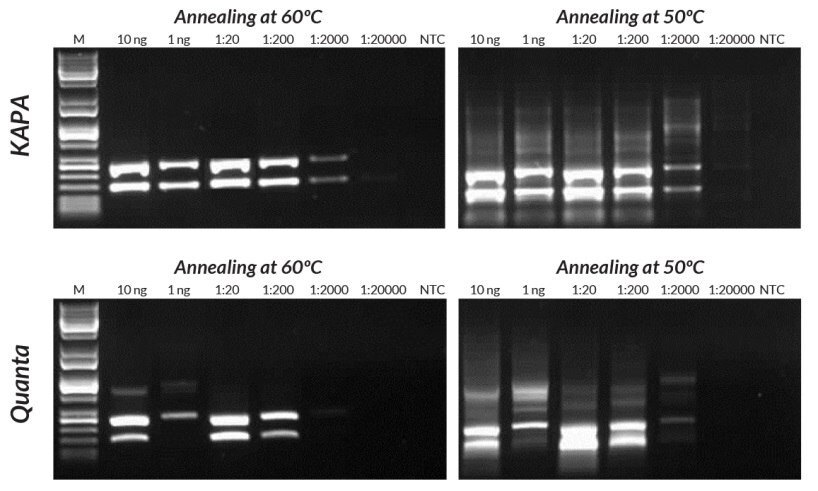
Rysunki 14 i 15.W porównaniu z zestawem do genotypowania myszy Quanta, zestawy do genotypowania myszy KAPA wykazują lepszą czułość, wydajność i swoistość w multipleksowej reakcji PCR. Reakcje przeprowadzono z wyżarzaniem w temperaturze 50°C lub 60°C, aby zademonstrować wpływ stosowania nieoptymalnych temperatur wyżarzania w testach genotypowania.
Zwiększona wydajność, czas realizacji i niezawodność
- DNA gotowe do PCR wygenerowane w zaledwie 15 minut
- Minimalna obsługa, zmniejszając tym samym ryzyko utraty lub zanieczyszczenia próbki
- Szablon wystarczający do wielu testów; łatwy do skalowania w celu obsługi próbek w formacie 96-dołkowym
- Szybka i niezawodna amplifikacja fragmentów DNA w szerokim zakresie długości amplikonów i zawartości GC
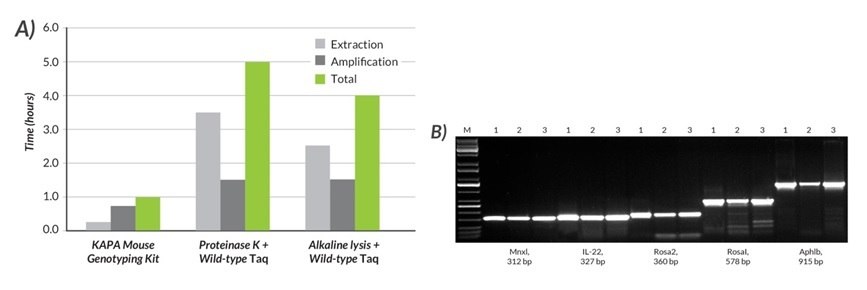
Rysunki 16 i 17.A) Porównano całkowity czas wymagany do ekstrakcji i amplifikacji DNA przy użyciu zestawu do genotypowania myszy KAPA, proteinazy K i Taq typu dzikiego oraz lizy alkalicznej i Taq typu dzikiego. B) Wyniki dla pięciu amplikonów (312 - 915 bp) wygenerowanych za pomocą zestawu do genotypowania myszy KAPA (1) porównano z wynikami uzyskanymi za pomocą dwóch powszechnie stosowanych metod (2 i 3). W przypadku zestawu do genotypowania myszy KAPA, lizaty DNA przygotowano z ogonów myszy za pomocą szybkiego (15 min), jednokanałowego systemu KAPA Express Extract. Amplifikacja za pomocą KAPA2G Fast HotStart ReadyMix z barwnikiem została zakończona w ciągu 45 minut. Natomiast lizaty DNA przygotowano za pomocą protokołu proteinazy K trwającego ~3,5 godziny (2) lub szybkiej (~2,5 godziny) metody lizy alkalicznej (3). W obu przypadkach amplifikację przeprowadzono przy użyciu Taq typu dzikiego (protokół cyklu 1,5 godziny). Wyniki uzyskane za pomocą zestawu do genotypowania myszy KAPA były takie same lub lepsze (bardziej specyficzne) niż te uzyskane innymi metodami, które (w zależności od zastosowanego protokołu ekstrakcji DNA) mogą trwać co najmniej cztery razy dłużej lub do 1 dnia.
KAPA EXPRESS EXTRACT
Szybka i wydajna ekstrakcja DNA
KAPA Express Extract to nowy termostabilny system proteazy i buforu, który pozwala na ekstrakcję DNA gotowego do PCR w zaledwie 15 minut. Ekstrakcje DNA są wygodnie przeprowadzane w pojedynczej probówce, co znacznie zmniejsza ryzyko utraty próbki i zanieczyszczenia. Połączenie KAPA Express Extract i KAPA2G Robust HotStart ReadyMix zapewnia wysokowydajne rozwiązanie do szybkiej ekstrakcji DNA i spójnej amplifikacji.
KAPA Express Extract zapewnia:
- Szybki protokół ekstrakcji z DNA gotowym do PCR w ciągu 15 minut dla spójnej amplifikacji wymagających typów próbek
- Wszechstronny, zoptymalizowany zestaw do skutecznej ekstrakcji DNA z różnych próbek i typów tkanek, w tym wymazów z policzka, mieszków włosowych, tkanek ryb i ptasich piór
- Reakcja w jednej probówce minimalizuje ryzyko zanieczyszczenia
- Wydajna ekstrakcja z kart "Guthrie", kart FTA® Elute i kart FTA

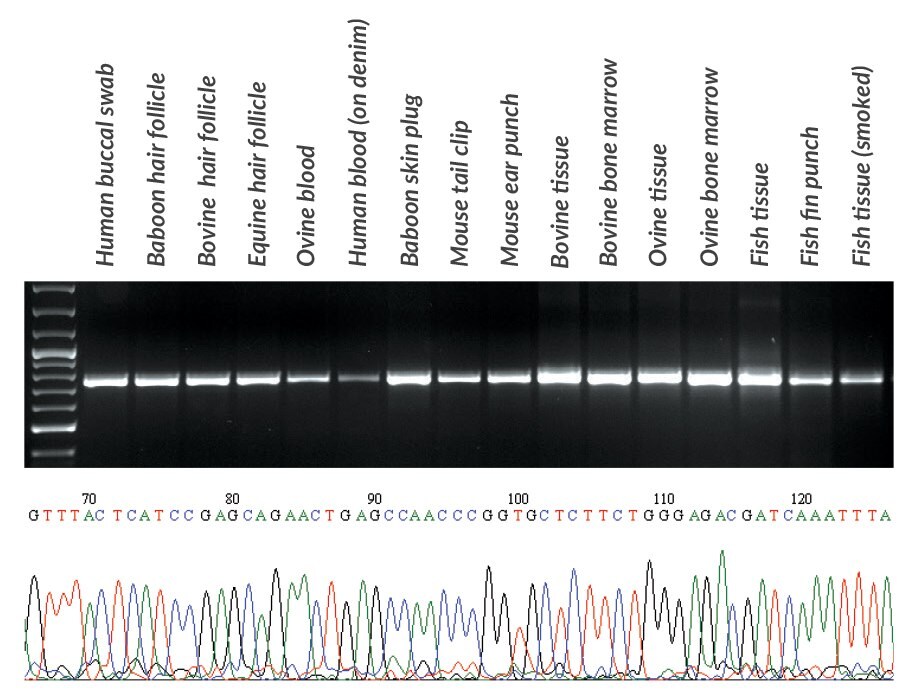
Rysunki 18 i 19.DNA ekstrahowano za pomocą KAPA Express Extract z różnych próbek uzyskanych od ssaków i ryb. Z każdego ekstraktu, 2 μL użyto bezpośrednio (bez kwantyfikacji) w PCR zawierającym KAPA2G Robust HotStart ReadyMix i startery dla fragmentu genu oksydazy cytochromu c I o wielkości ~650 bp powszechnie stosowanego w identyfikacji gatunków (Ivanova, et al., 2007). Produkty PCR (10 μl) analizowano w 1% żelu agarozowym. Pochodzenie i typ próbki są wyświetlane nad żelem. Produkty reakcji wykorzystano bezpośrednio w standardowych reakcjach sekwencjonowania Sangera przy użyciu zagnieżdżonych starterów M13 (2 μL produktu PCR na 10 μL reakcji sekwencjonowania). Dane sekwencyjne były wysokiej jakości i umożliwiły identyfikację każdego gatunku. Sekcja śladu sekwencji z tkanki Seriola lalandi (Yellowtail amberjack) jest przedstawiona w dolnym panelu.
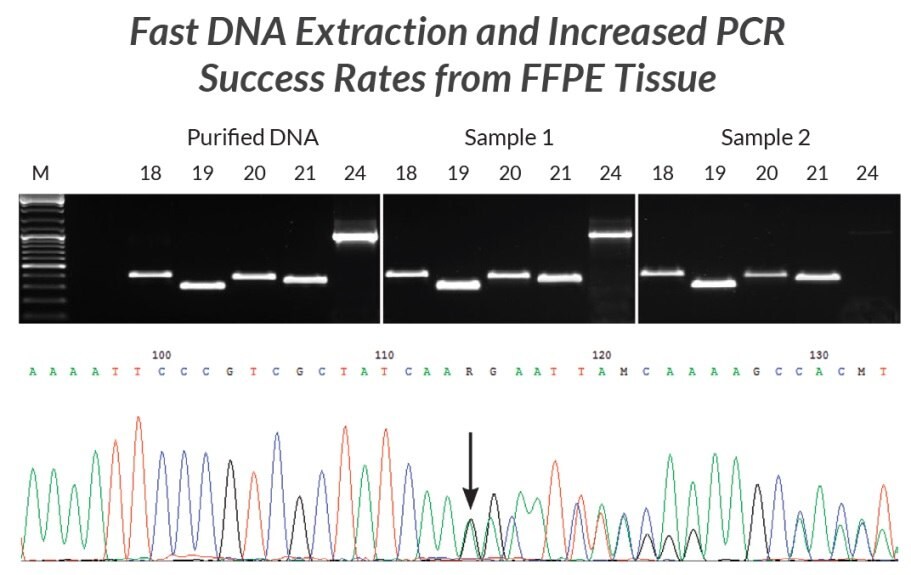
Rysunek 20.Ekstrakty DNA przygotowano z dwóch różnych próbek FFPE przy użyciu KAPA Express Extract. Każdy ekstrakt został użyty bezpośrednio (bez kwantyfikacji) w wielu reakcjach PCR zawierających KAPA2G Robust HotStart ReadyMix i startery dla pięciu różnych fragmentów (293 bp - 1 kb) genu EGFR (odpowiadających eksonom 18 - 21 i 24). Wyniki porównano z wynikami uzyskanymi przy użyciu tych samych warunków reakcji i cykli, ale przy użyciu 1 ng oczyszczonego ludzkiego genomowego DNA jako matrycy. Z wyjątkiem fragmentu eksonu 24 o długości 1 kb ze starszej próbki, wydajność i skuteczność reakcji były porównywalne między ekstraktami FFPE DNA i oczyszczonym genomowym DNA. Produkty PCR wygenerowane z próbki 1 rozcieńczono w stosunku 1:10 i wykorzystano bezpośrednio w standardowych reakcjach sekwencjonowania Sangera. Dane sekwencjonowania (dolny panel, fragment egzonu 19 próbki 1) były wysokiej jakości. Sekwencja mieszana rozpoczynająca się w pozycji oznaczonej strzałką potwierdziła obecność 15-nt delecji związanej z rakiem niedrobnokomórkowym zdiagnozowanym u pacjenta, od którego pobrano próbkę 1.
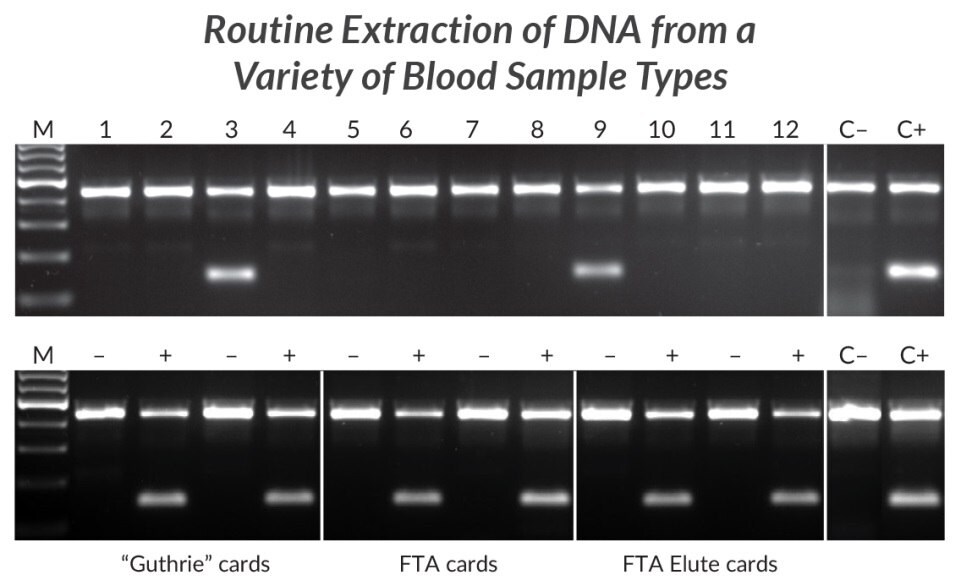
Rysunek 21.Ekstrakcja i amplifikacja DNA z różnych typów próbek krwi w celu wykrycia allelu HLA-B*27. DNA ekstrahowano z 12 ludzkich próbek krwi EDTA za pomocą ekstraktu KAPA Express (górny panel). 2 μL każdego ekstraktu dodano bezpośrednio do 25 μL PCR zawierającego KAPA2G Robust HotStart ReadyMix i dwa zestawy starterów. Zestaw starterów kontroli wewnętrznej jest ukierunkowany na fragment 429 bp genu beta globiny, podczas gdy drugi zestaw starterów jest ukierunkowany na fragment 141 bp locus HLA-B*27 w sposób specyficzny dla sekwencji. Dwie z 12 osób uzyskały pozytywny wynik testu na obecność allelu HLA-B*27 związanego ze zesztywniającym zapaleniem stawów kręgosłupa. Pasy C- i C+ reprezentują odpowiednio negatywną i pozytywną kontrolę HLA-B*27 (1 ng oczyszczonego ludzkiego genomowego DNA jako matryca). DNA wyekstrahowano z kart "Guthrie", kart FTA lub kart FTA Elute (dolny panel) splamionych krwią osób, u których potwierdzono, że są HLA-B*27 dodatnie (+) lub ujemne (-). Warunki ekstrakcji i amplifikacji DNA oraz kontrole (C- i C+) były takie same jak w przypadku panelu górnego.
KAPA3G PLANT PCR KITS
KAPA3G Plant PCR Kits są przeznaczone do wykorzystania oczyszczonego DNA lub surowego ekstrahowanego DNA do PCR DNA pochodzenia roślinnego. Zestaw zawiera polimerazę DNA KAPA3G, która została zaprojektowana z myślą o lepszej tolerancji na powszechne inhibitory PCR pochodzenia roślinnego, takie jak polifenole i polisacharydy.
Zestawy KAPA3G Plant PCR zapewniają:
- Szybki PCR bezpośrednio z tkanek roślinnych, takich jak krążki liściowe, nasiona i surowe ekstrakty roślinne
- Usprawnione przepływy pracy dla badań przesiewowych transgenów
- Poprawione wskaźniki sukcesu i powtarzalności PCR
Usprawnienie przepływu pracy i poprawa czasu realizacji
- Wykonaj PCR w czasie o połowę krótszym w porównaniu z enzymami typu dzikiego
- Eliminuj potrzebę czasochłonnych ekstrakcji DNA
- Nadaje się do bezpośredniego PCR
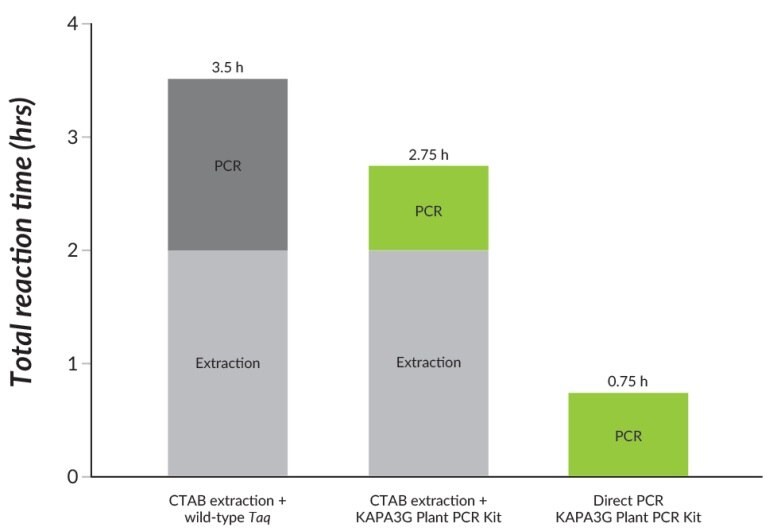
Rysunek 22.Bezpośrednia reakcja PCR z użyciem zestawu KAPA3G Plant PCR Kit przewyższa ekstrakcję CTAB i standardową reakcję PCR z użyciem Taq typu dzikiego, w znacznie krótszym czasie.
Amplikuj długie cele z surowych próbek i oczyszczonego DNA
- Amplikuj fragmenty do 5 kb
- Wysoka wydajność i specyficzność
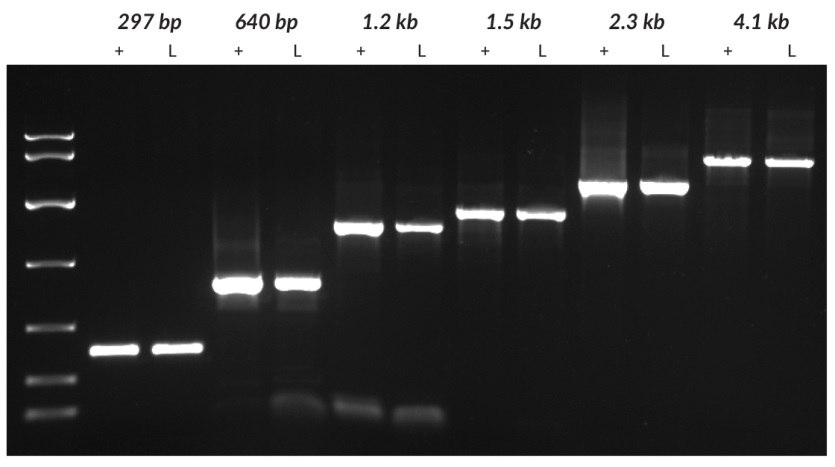
Rysunek 23.argety o różnej długości (297 bp i 4100 bp z tytoniu, 640 bp z pomidora, 1221 bp z winorośli oraz 1448 bp i 2249 bp z ziemniaka) amplifikowano z oczyszczonego DNA (+) lub krążków liściowych (L) przy użyciu zestawu KAPA3G Plant PCR Kit. Dla każdego gatunku genomowe DNA oczyszczono przy użyciu komercyjnego zestawu do oczyszczania DNA. Surowy materiał został pobrany przy użyciu narzędzia do pobierania próbek Harris Uni-Core™ o średnicy 0,5 mm. Oczyszczone DNA (1 - 10 ng na reakcję, w zależności od gatunku) i surowe próbki wykorzystano jako szablony w reakcjach PCR o objętości 50 μl, z 40 cyklami amplifikacji. Produkty reakcji analizowano na 1% żelu agarozowym. Jako markera MW użyto KAPA Express DNA Ladder (100, 200, 400, 800, 1600, 4000, 8000 bp).
Przeprowadź bezpośrednią reakcję PCR z różnych gatunków roślin i typów tkanek
- Bezpośrednia reakcja PCR z krążkiem liściowym lub nasionami jako matrycą
- Bez potrzeby czasochłonnej ekstrakcji DNA
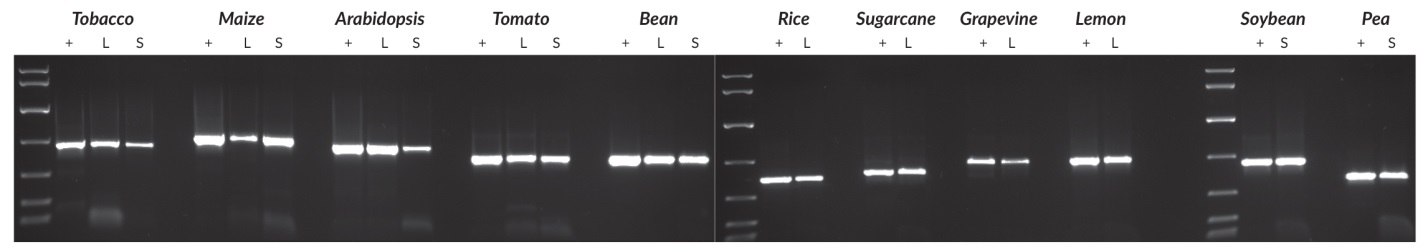
Rysunek 24.Genomowe DNA roślin oczyszczono ze wszystkich gatunków przy użyciu komercyjnego zestawu do oczyszczania DNA. Narzędzie do pobierania próbek Harris Uni-Core™ (o średnicy 0,5 mm) zostało użyte do pobierania próbek liści (wszystkie gatunki) lub nasion (wszystkie gatunki z wyjątkiem tytoniu i Arabidopsis; w tym przypadku na reakcję użyto jednego pokruszonego nasiona). Reakcje PCR (50 μL) zawierały surową próbkę lub 1-10 ng oczyszczonego DNA (w zależności od gatunku) i we wszystkich przypadkach wykonano 40 cykli. Cele mieściły się w zakresie od 500 do 900 bp, a produkty reakcji analizowano w 1% żelu agarozowym. Jako markera MW użyto KAPA Express DNA Ladder (100, 200, 400, 800, 1600, 4000, 8000 bp).
Popraw wskaźniki sukcesu dzięki nowemu przepływowi pracy PCR dla surowych próbek roślinnych
- Użyj buforu ekstrakcyjnego do przygotowania surowych ekstraktów do PCR roślinnego w zaledwie 5 minut
- Wysokie wskaźniki sukcesu nawet z najtrudniejszymi typami próbek
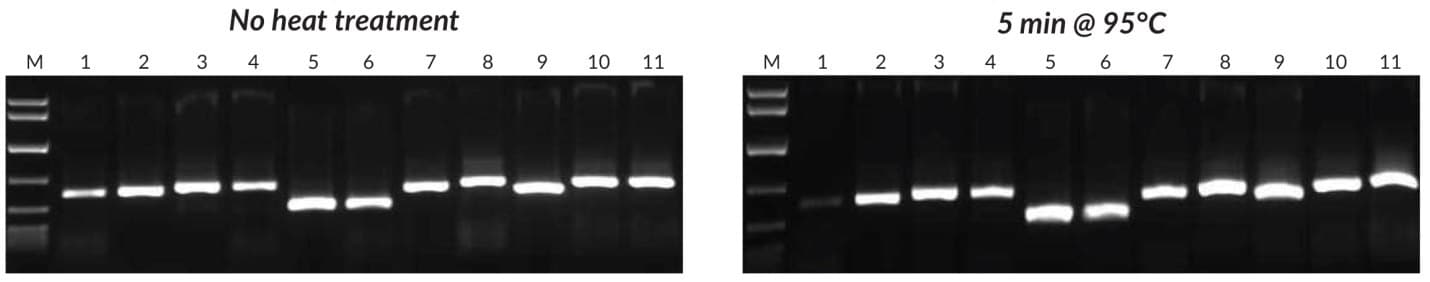
Rysunek 25.PCR surowej próbki z parą starterów Tab c/d przeprowadzono na liściach i/lub nasionach 8 roślin, z 1 μL surowego ekstraktu przygotowanego bez obróbki cieplnej (po lewej) lub 1 μL surowego ekstraktu przygotowanego z obróbką cieplną (po prawej). Reakcje ustawiono zgodnie z opisem w KAPA3G Plant PCR Kit TDS i wykonano 45 cykli PCR z wyżarzaniem w 55 ° C i 20-sekundowym przedłużaniem w 72 ° C na cykl. Jako markera MW użyto KAPA Express DNA Ladder (100, 200, 400, 800, 1600, 4000, 8000 bp). 1: Eukaliptus; 2: Winorośl; 3: Liść pszenicy; 4: Nasiona pszenicy; 5: Liść rzepaku; 6: Nasiona rzepaku; 7: Nasiona ryżu; 8: Nasiona jęczmienia; 9: Ziarno kukurydzy; 10: Nasiona bawełny; 11: Liść bawełny.
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?