Wszystkie zdjęcia(1)
Kluczowe dokumenty
MSAMPURO
Mouse Whole Genome SAM CRISPRa Pooled Lentiviral Library Kit Puro
Zaloguj sięWyświetlanie cen organizacyjnych i kontraktowych
About This Item
Kod UNSPSC:
41106621
NACRES:
NA.51
Polecane produkty
opakowanie
pkg of 40 vials (8x25μL aliquots for each of the 5 kit components)
pkg of
Zastosowanie
CRISPR
Warunki transportu
dry ice
temp. przechowywania
−70°C
Powiązane kategorie
Opis ogólny
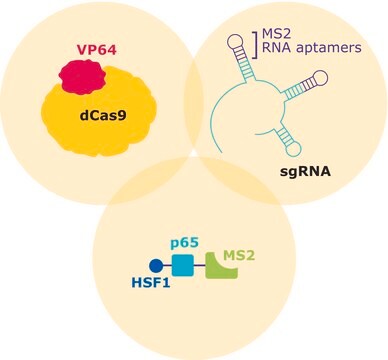
Aktywator transkrypcyjny VP64 połączony z nukleazą SpCas9 (dCas9) tworzy kompleks rybonukleoproteinowy (RNP) z RNA-przewodnikiem CRISPR (gRNA). Sekwencje łodygi i tetra-pętli w rusztowaniu gRNA zostały zmodyfikowane w minimalne aptamery RNA spinki do włosów, które selektywnie wiążą dimeryzowane białka płaszcza bakteriofaga MS2. Białko płaszcza MS2 jest połączone z podjednostką p65 NF-kappaB (NFκB) i domeną aktywacyjną ludzkiego czynnika szoku cieplnego 1 (HSF1). Prowadnica RNA zawiera dwa aptamery, z których każdy może wiązać dwa białka koaktywatora MS2, skutecznie rekrutując cztery koaktywatory dla każdego kompleksu aktywatora celującego CRISPR.
Konstrukty pomocnicze dCas9-VP64, MS2-p65-HSF1 i gRNA są dostarczane jako trzy oddzielne plazmidy/cząsteczki lentiwirusowe. W przeciwieństwie do macierzowych bibliotek gRNA zawierających każdy klon w osobnym dołku na płytce, zbiorcze biblioteki gRNA zawierają tysiące pojedynczych gRNA w jednej probówce, umożliwiając wydajne przesiewanie całego ludzkiego genomu (ponad 16 000 genów) na stole laboratoryjnym bez robotyki lub specjalistycznego sprzętu. Biblioteka SAM CRISPR jest dostarczana w 3 pulach. Każda pula zawiera około 23 500 gRNA, z 3 gRNA na RefSeqID. Biblioteka całego genomu zawiera łącznie około 70 000 gRNA ukierunkowanych na 19 000 unikalnych symboli genów (w tym alternatywne izoformy obejmujące 23 500 RefSeqID).
W każdej puli znajduje się 100 niecelujących (tj. negatywnych) kontrolnych gRNA do wykorzystania jako punkt odniesienia w statystycznej charakterystyce zmian częstotliwości gRNA mierzonych przez głębokie sekwencjonowanie. Negatywne kontrolne gRNA mają minimalną homologię z genomem docelowym i nie powinny podlegać znaczącym zmianom w reprezentacji podczas leczenia komórek kontrolnych. Ze względu na charakter eksperymentów przesiewowych z aktywatorami CRISPR, kontrole pozytywne muszą być określane empirycznie, biorąc pod uwagę specyficzny fenotyp, który ma być badany na ekranie. Łącznie kontrole te stwarzają wiele możliwości wcześniejszego projektowania eksperymentów i późniejszej analizy danych.
Konstrukty pomocnicze dCas9-VP64, MS2-p65-HSF1 i gRNA są dostarczane jako trzy oddzielne plazmidy/cząsteczki lentiwirusowe. W przeciwieństwie do macierzowych bibliotek gRNA zawierających każdy klon w osobnym dołku na płytce, zbiorcze biblioteki gRNA zawierają tysiące pojedynczych gRNA w jednej probówce, umożliwiając wydajne przesiewanie całego ludzkiego genomu (ponad 16 000 genów) na stole laboratoryjnym bez robotyki lub specjalistycznego sprzętu. Biblioteka SAM CRISPR jest dostarczana w 3 pulach. Każda pula zawiera około 23 500 gRNA, z 3 gRNA na RefSeqID. Biblioteka całego genomu zawiera łącznie około 70 000 gRNA ukierunkowanych na 19 000 unikalnych symboli genów (w tym alternatywne izoformy obejmujące 23 500 RefSeqID).
W każdej puli znajduje się 100 niecelujących (tj. negatywnych) kontrolnych gRNA do wykorzystania jako punkt odniesienia w statystycznej charakterystyce zmian częstotliwości gRNA mierzonych przez głębokie sekwencjonowanie. Negatywne kontrolne gRNA mają minimalną homologię z genomem docelowym i nie powinny podlegać znaczącym zmianom w reprezentacji podczas leczenia komórek kontrolnych. Ze względu na charakter eksperymentów przesiewowych z aktywatorami CRISPR, kontrole pozytywne muszą być określane empirycznie, biorąc pod uwagę specyficzny fenotyp, który ma być badany na ekranie. Łącznie kontrole te stwarzają wiele możliwości wcześniejszego projektowania eksperymentów i późniejszej analizy danych.
Zastosowanie
- Genomika funkcjonalna
- Walidacja celu
- Badanie przesiewowe zysku funkcji w całym genomie
Cechy i korzyści
- Wysoka specyficzność i aktywność
- Cząsteczki lentiwirusowe o wysokiej czystości i wysokim mianie, zweryfikowane pod względem sekwencji
- Aktywuje geny poprzez aktywację transkrypcyjną zamiast nadekspresji opartej na cDNA
Dowiedz się więcej o aktywatorach SAM CRISPR na stronie SigmaAldrich.com/CRISPRa.
Komponenty
This kit contains 5 components:
2 Helper Constructs with a minimum concentration of 1x106 VP/mL(via p24 assay)
3 Subpools with a minimum concentration of 5x108 VP/mL(via p24 assay)
2 Helper Constructs with a minimum concentration of 1x106 VP/mL(via p24 assay)
- 8 x 25μl vials of dCas9-VP64-Blasticidin SAM CRISPRa Helper Construct 1 Lentiviral Transduction Particles
- 8 x 25μl vials of MS2-P65-HSF1-Hygromycin SAM CRISPRa Helper Construct 2 Lentiviral Transduction Particles
3 Subpools with a minimum concentration of 5x108 VP/mL(via p24 assay)
- 8 x 25μl vials of Mouse SAM CRISPRa Library Pool 1 - Puro
- 8 x 25μl vials of Mouse SAM CRISPRa Library Pool 2 - Puro
- 8 x 25μl vials of Mouse SAM CRISPRa Library Pool 3 - Puro
Zasada
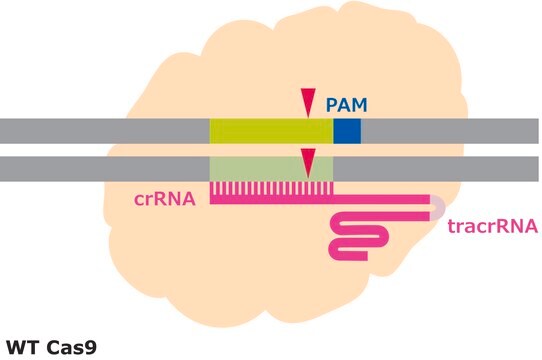
Systemy CRISPR/Cas są wykorzystywane przez bakterie i archeony do obrony przed inwazją wirusów i plazmidów. Niedawno system CRISPR/Cas typu II z bakterii Streptococcus pyogenes został zaprojektowany do funkcjonowania w systemach eukariotycznych przy użyciu dwóch składników molekularnych: pojedynczego białka Cas9 i niekodującego RNA (gRNA). Endonukleaza Cas9 może być nieaktywna (dCas9) z mutacjami w dwóch domenach białkowych, RuvC i HnH (odpowiednio D10A i H840A), które są odpowiedzialne za aktywność nukleazy. Białko z niedoborem nukleazy można następnie połączyć z aktywatorem transkrypcyjnym VP64 i wykorzystać w połączeniu z RNA-przewodnikiem zmodyfikowanym aptamerami MS2 RNA, które działają w celu rekrutacji dodatkowych koaktywatorów transkrypcyjnych p65 i HSF1. Zmontowany kompleks SAM jest następnie wykorzystywany jako system dostarczania ładunku do docelowych promotorów genów, umożliwiając specyficzną dla miejsca aktywację transkrypcji interesującego nas genu.
Definicja jednostki
VP/ml jest jednostką miary stężenia dla miana wirusa oszacowanego za pomocą testu p24.
Informacje prawne
Ta strona może zawierać tekst przetłumaczony maszynowo.
Kod klasy składowania
10 - Combustible liquids
Wybierz jedną z najnowszych wersji:
Certyfikaty analizy (CoA)
Lot/Batch Number
It looks like we've run into a problem, but you can still download Certificates of Analysis from our Dokumenty section.
Proszę o kontakt, jeśli potrzebna jest pomoc Obsługa Klienta
Masz już ten produkt?
Dokumenty związane z niedawno zakupionymi produktami zostały zamieszczone w Bibliotece dokumentów.
Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex
Konermann S, et al
Nature, 517, 583-588 (2015)
Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex
Konermann S, et al.
Nature, 517, 583-588 (2015)
Genome-scale CRISPR-Cas9 Knockout and Transcriptional Activation Screening.
Joung, S. et al.
Nature Protocols, 12, 828-863 (2017)
Julia Joung et al.
Nature protocols, 12(4), 828-863 (2017-03-24)
Forward genetic screens are powerful tools for the unbiased discovery and functional characterization of specific genetic elements associated with a phenotype of interest. Recently, the RNA-guided endonuclease Cas9 from the microbial CRISPR (clustered regularly interspaced short palindromic repeats) immune system
Nasz zespół naukowców ma doświadczenie we wszystkich obszarach badań, w tym w naukach przyrodniczych, materiałoznawstwie, syntezie chemicznej, chromatografii, analityce i wielu innych dziedzinach.
Skontaktuj się z zespołem ds. pomocy technicznej