Tkanka FFPE
Zarchiwizowane próbki tkanek utrwalone w formalinie i zatopione w parafinie (FFPE) są nieocenionymi zasobami do profilowania ekspresji genów i badania różnych chorób. Ponieważ zarchiwizowane DNA jest zwykle dostępne w ograniczonych ilościach, amplifikacja próbek jest niezbędna. Amplifikacja tkanki FFPE może być trudnym zadaniem ze względu na uszkodzoną matrycę, która wynika z procesu archiwizacji. Niniejszy protokół zapewnia wygodną metodę oczyszczania i amplifikacji genomowego DNA z tkanki FFPE. GenomePlex® Whole Genome Amplification został użyty do amplifikacji genomowego DNA ze szczurzych i ludzkich próbek tkanek FFPE.
Uwaga: Poniższy protokół opisuje proces oczyszczania DNA i późniejszej amplifikacji całego genomu. Znacznie usprawniliśmy ten proces dzięki naszemu zestawowi GenomePlex® Tissue Whole Genome Amplification Kit (Product No. WGA2+C4482). Zestaw ten zawiera zoptymalizowane odczynniki do rozbijania tkanek i lizy komórek, eliminując potrzebę żmudnych ekstrakcji organicznych w celu usunięcia nadmiaru parafiny lub oczyszczania DNA przed amplifikacją.
Wymagane produkty
Materiały dostarczane przez użytkownika
- Tkanka FPE
- Łaźnia wodna 37 °C lub blok cieplny
- Łaźnia wodna 55 °C lub blok cieplny
- 70 °C łaźnia wodna lub blok cieplny
- ksylen
- etanol (E7023)
- woda, odczynnik do biologii molekularnej (W4502)
- Mikrowirówka (z rotorem dla probówek 2 mL)
Ekstrakcja DNA z tkanki utrwalonej w formalinie i zatopionej w parafinie (FFPE)
Ten protokół został przeprowadzony na tkance wątroby szczura. Do tej procedury zalecany jest zestaw GenElute™ Mammalian Genomic DNA Miniprep Kit (G1N10) .
- Umieść mały wycinek (20 mg) tkanki zatopionej w parafinie w 2 ml probówce do mikrowirówki.
- Dodaj 1200 µL ksylenu i worteksuj przez 30 sekund.
- Wiruj z pełną prędkością przez 5 minut w temperaturze pokojowej.
- Usuń supernatant przez pipetowanie. Nie usuwaj osadu.
- Dodaj 1200 µl etanolu do osadu, aby usunąć resztki ksylenu. Wymieszać przez worteksowanie.
- Wirować z pełną prędkością przez 5 minut w temperaturze pokojowej.
- Ostrożnie usunąć etanol przez pipetowanie. Nie usuwaj żadnego osadu.
- Powtórz kroki 5-7 ponownie.
- Inkubuj otwartą probówkę mikrowirówki w temperaturze 37 °C przez 10-15 minut, aby usunąć resztki etanolu przez odparowanie.
- Traw tkanki: Zawiesić osad tkanki w 180 µL Roztworu do Lizy T.
- Dodać 20 µL Proteinazy K. Wymieszać przez worteksowanie. Inkubować w temperaturze 55°C przez noc lub do momentu całkowitej lizy tkanki. Worteksować od czasu do czasu podczas inkubacji.
Opcjonalna obróbka RNazą: Jeśli pozostałości RNA budzą obawy, dodać 20 µL roztworu RNazy A i inkubować w temperaturze pokojowej przez 2 minuty. - Liza komórek: Worteksować przez 15 sekund. Dodaj 200 µL roztworu lizującego C do próbki. Worteksuj dokładnie, ponieważ jednorodna mieszanina jest niezbędna do skutecznej lizy. Inkubować w temperaturze 70°C przez 10 minut.
- Przygotować kolumnę: Dodaj 500 µL roztworu przygotowującego kolumnę do każdej wstępnie zmontowanej kolumny wiążącej GenElute Miniprep i odwiruj z prędkością 12,000 × g przez 1 minutę.
- Przygotuj do wiązania: Dodaj 200 µL etanolu do zlizowanej próbki i wymieszaj przez worteksowanie.
- Załaduj lizat: Przenieś całą zawartość probówki z próbką do poddanej obróbce kolumny wiążącej z kroku 14. Wirować z prędkością ≥6500 × g przez 1 minutę. Wyrzucić probówkę zbierającą zawierającą ciecz przepływową i umieścić kolumnę wiążącą w nowej probówce zbierającej o pojemności 2 ml.
- Pierwsze płukanie: Przed pierwszym użyciem rozcieńczyć koncentrat roztworu płuczącego etanolem, jak opisano w instrukcji przygotowania. Dodać 500 µL roztworu płuczącego do kolumny wiążącej i wirować przez 1 minutę z prędkością ≥6 500 × g. Wyrzucić probówkę zbierającą zawierającą ciecz przepływową i umieścić kolumnę wiążącą w nowej probówce zbierającej o pojemności 2 ml.
- Drugie płukanie: Dodaj kolejne 500 µL roztworu płuczącego do kolumny wiążącej i wiruj przez 3 minuty z maksymalną prędkością (12 000-18 000 × g), aby osuszyć kolumnę wiążącą. Ważne jest, aby kolumna wiążąca była wolna od etanolu przed elucją DNA z kolumny. Jeśli widoczne są pozostałości etanolu, odwiruj kolumnę przez dodatkową minutę. Wyrzuć probówkę zbierającą zawierającą ciecz przepływową i umieść kolumnę wiążącą w nowej probówce zbierającej o pojemności 2 ml.
- Elucja DNA: Odmierzyć pipetą 200 µL roztworu elucyjnego bezpośrednio do środka kolumny wiążącej i inkubować w temperaturze pokojowej przez 5 minut. Wirować przez 1 minutę z prędkością ≥6500 × g w celu elucji DNA.
- Przechowywać próbki DNA w temperaturze -20 °C.
Uwaga: Protokół ten można wykonać bez użycia ksylenu, zaczynając od kroku numer 10. W wyniku pominięcia etapu obróbki ksylenem, ilość DNA zmniejszy się o około 50% w porównaniu do protokołu z etapem ksylenowym.
Dane aplikacyjne
GenomePlex® Whole Genome Amplification FFPE Tissue
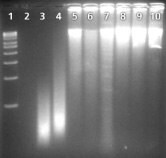
Legenda: Produkty amplifikowano przy użyciu zestawu do amplifikacji całego genomu GenomePlex®. Whole Genome Amplification Kit (WGA1) od nas, dostawcy A i dostawcy Q. Produkty rozdzielono na 1,5% żelu agarozowym. Do każdej studzienki dodano 5 µL amplifikowanego produktu. Produkty amplifikowane przy użyciu technologii GenomePlex miały mniejszą masę cząsteczkową, jak pokazano na żelu w porównaniu z dostawcami A i Q. Wynika to z losowej fragmentacji genomowego DNA przed amplifikacją. Nasze amplifikowane produkty są specyficzne i nie ma amplikonu widocznego w kontroli negatywnej (pas 2), co wskazuje, że amplifikowane jest tylko pożądane genomowe DNA. Zarówno dostawcy A, jak i Q dają niespecyficzny sygnał w kontroli negatywnej, który jest równy pod względem wielkości i intensywności sygnałowi dla kontroli pozytywnej dostawców.
| Lane 1-1 kb Marker | Lane 6-.Dostawca A 10 ng wejściowego DNA |
| Listwa 2-Nasza kontrola negatywna | Listwa 7-Dostawca A 100 ng wejściowego DNA |
| Listwa 3-.Nasze 10 ng wejściowego DNA | Linia 8-Dostawca Q Kontrola negatywna |
| Linia 4-Nasze 100 ng wejściowego DNA | Linia 9-Dostawca Q 10 ng wejściowego DNA |
| Linia 5 - Kontrola negatywna dostawcy A | Linia 10 - Dostawca Q 100 ng wejściowego DNA |

Legenda: DNA ekstrahowano z próbki utrwalonej w formalinie, zatopionej w parafinie wątroby szczura. 10 ng i 100 ng genomowego DNA amplifikowano przy użyciu GenomePlex® Complete WGA Kit (Nr produktu. WGA2) po czym następuje oczyszczanie przy użyciu GenElute™ PCR Clean-up Kit (NA1020). Ilościowy PCR (45 cykli) przeprowadzono na reakcji WGA przy użyciu 12 różnych zestawów starterów. GenomePlex wykazuje ~1000-krotnie (10 Ct) lepszą reprezentację w porównaniu do dostawców.
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?