Edycja genomu roślin za pomocą CRISPR/Cas9
Pomyślne celowanie w geny indukowane przez ZFN zostało opublikowane już w 2003 roku. Od tego czasu technologia ukierunkowanej edycji genomu szybko się rozwinęła i została udostępniona komercyjnie. Ostatnio odkrycie szlaku CRISPR/Cas9 przyspieszyło zainteresowanie tą dziedziną, otwierając nowe możliwości dla badań i rozwoju. Chociaż szlak CRISPR został zidentyfikowany w bakteriach jako część domniemanego adaptacyjnego układu odpornościowego, szybko został dostosowany do celów modyfikacji genomów eukariotycznych. Podczas gdy narzędzia takie jak ZFN położyły podwaliny pod dzisiejszą edycję genomu, istnieją ograniczenia tej założycielskiej technologii i innych jej podobnych: interakcja białko:DNA ZFN sprawia, że ich projektowanie jest złożone, montaż konstruktu ekspresji ZFN jest czasochłonny, a opcje celowania ZFN są ograniczone w wielu genomach roślin bogatych w A-T. Szlak CRISPR, który został przyjęty i zmodyfikowany do edycji genomu eukariotycznego, pokonuje wiele z tych przeszkód: opiera się na interakcji RNA:DNA w celu znalezienia celu genomowego, sekwencja rozpoznawania wymagana do tego wiązania to łatwo zmienione 18-20 par zasad, a jedynym wymogiem wiązania nukleazy jest obecność NGG obok miejsca docelowego. Pierwsze doniesienia o zastosowaniu CRISPR/Cas9 w roślinach w testach przejściowej ekspresji przy użyciu Agrobacterium pojawiły się w 2013 roku. Technologia CRISPR/Cas9 została z powodzeniem zastosowana w roślinach modelowych (Nicotiana benthamiana, Arabidopsis thaliana) i uprawnych (ryż, pszenica), a lista ta stale rośnie.
- CRISPR/Cas9: Co to jest i jak działa?
- Korzyści z używania CRISPR/Cas9 do edycji genomu
- Uproszczony przepływ pracy: CRISPR/Cas9 w roślinach
- Gotowe do użycia plazmidy ekspresyjne Cas9 i Guide RNA (gRNA) dla roślin jednoliściennych i dwuliściennych
- Niestandardowe produkty roślinne CRISPR/Cas9
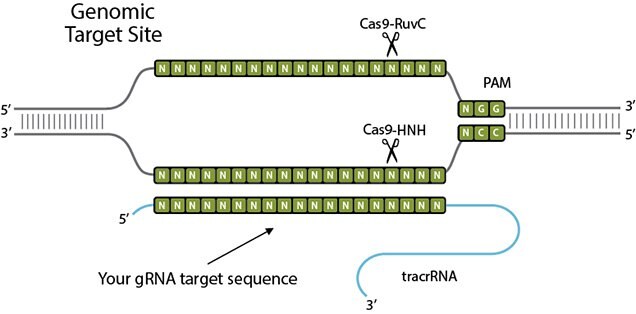
CRISPR/Cas9: Co to jest i jak działa?
CRISPR to skrót od Clustered Regularly Interspaced Short Palindromic Repeats. Odkrycie prokariotycznego "systemu immunologicznego" CRISPR typu II pozwoliło na opracowanie prostego, łatwego i szybkiego do wdrożenia narzędzia do edycji genomu sterowanego RNA. System CRISPR/Cas9 składa się z pojedynczego monomerycznego białka i chimerycznego RNA. Sekwencja 20-nt w gRNA nadaje specyficzność sekwencji, a w rozszczepieniu pośredniczy białko Cas9. Parowanie zasad Watsona-Cricka z docelową sekwencją DNA jest podstawą rozszczepienia opartego na gRNA, dzięki czemu zaawansowana inżynieria białek dla każdego celu jest zbędna. Jedynie 20 nt w gRNA musi zostać zmodyfikowane, aby ułatwić rozpoznanie innego celu.
CRISPR/Cas9 składa się z białka Cas9, CRISPR RNA (crRNA) i trans-aktywującego crRNA (tracrRNA). W zastosowaniach związanych z edycją genów, crRNA i tracrRNA są często łączone w pojedyncze RNA prowadzące (sgRNA). Rybonukleoproteina atakuje cel za pomocą sekwencji prowadzącej crRNA, tworząc hybrydę 20-bp RNA/DNA i wypierając przeciwną nić DNA po napotkaniu sąsiedniego motywu protospacer (PAM), takiego jak NGG. Endonukleaza Cas9 rozszczepia następnie komplementarną nić DNA (nić docelową) za pomocą domeny nukleazy HNH i przesuniętą nić DNA (nić niecelową) za pomocą domeny nukleazy podobnej do RuvC, tworząc podwójne przerwanie nici (DSB). Naprawa DSB przez komórkę gospodarza poprzez niehomologiczne łączenie końców (NHEJ) lub ścieżkę naprawy ukierunkowanej homologicznie (HDR) może być wykorzystana do znokautowania genu lub wprowadzenia określonej modyfikacji genetycznej poprzez homologiczną rekombinację z dawcą DNA.
RNA-guided endonucleases (RGENs) składające się z białka Cas9 pochodzącego ze Streptococcus pyogenes i guide RNA (gRNAs) można dostosować poprzez zastąpienie tylko składnika RNA, co prowadzi do zmniejszenia nakładu pracy i czasu w porównaniu z innymi metodami edycji genów. Używając bakterii Agrobacterium tumefaciens lub transfekując plazmidy, które je kodują, programowalne nukleazy mogą być dostarczane do komórek roślinnych, gdzie te nukleazy rozszczepiają chromosomalne miejsca docelowe w sposób zależny od sekwencji.Rezultatem są specyficzne dla miejsca pęknięcia dwuniciowe DNA (DSB), których naprawa przez systemy endogenne skutkuje ukierunkowanymi modyfikacjami genomu.
Korzyści ze stosowania CRISPR/Cas9 do edycji genomu
Główne zalety CRISPR/Cas9 to prostota, dostępność, koszt i wszechstronność.
System CRISPR/Cas9 nie wymaga żadnych etapów inżynierii białek, co znacznie ułatwia testowanie wielu gRNA dla każdego genu docelowego.
Tylko 20 nt w sekwencji gRNA musi zostać zmienione, aby nadać inną specyficzność docelową, co oznacza, że klonowanie jest również zbędne.
Dowolna liczba gRNA może być wytwarzana przez in vitro transkrypcję przy użyciu dwóch komplementarnych, wyżarzonych oligonukleotydów. Pozwala to na niedrogie tworzenie dużych bibliotek gRNA, dzięki czemu system CRISPR/Cas9 może być wykorzystywany do wysokowydajnych zastosowań genomiki funkcjonalnej.
Kolejną zaletą CRISPR/Cas9 w porównaniu do ZFN i TALEN jest łatwość multipleksowania. Jednoczesne wprowadzenie DSB w wielu miejscach może być wykorzystane do edycji kilku genów w tym samym czasie. Może to być szczególnie przydatne do eliminacji zbędnych genów lub równoległych ścieżek. Naukowcy mogą zaprojektować duże delecje genomowe lub inwersje, celując w dwa szeroko rozmieszczone miejsca rozszczepienia na tym samym chromosomie. Edycja multipleksowa za pomocą systemu CRISPR/Cas9 wymaga po prostu monomerycznego białka Cas9 i dowolnej liczby różnych specyficznych dla sekwencji gRNA. W przeciwieństwie do tego, multipleksowa edycja za pomocą ZFN lub TALEN wymaga oddzielnych białek dimerycznych specyficznych dla każdego miejsca docelowego.
System CRISPR/Cas9 może rozszczepiać metylowane DNA w ludzkich komórkach, umożliwiając modyfikacje genomowe, które są poza zasięgiem innych nukleaz. Chociaż nie zostało to szczegółowo zbadane w roślinach, uzasadnione jest przekonanie, że zdolność do rozszczepiania metylowanego DNA jest nieodłączną cechą systemu CRISPR/Cas9 i nie zależy od docelowego genomu.
Uproszczony przepływ pracy: CRISPR/Cas9 w roślinach
Biotechnologia roślin wkracza w nową erę wraz z wprowadzeniem technologii edycji genomu, która umożliwia precyzyjną manipulację określonym genomem, zastępując tym samym starsze metody losowej mutagenezy, takie jak mutageneza EMS i sekwencje promieniowania g. Roślinne produkty CRISPR/Cas9 są przeznaczone do transformacji roślin za pośrednictwem bakterii Agrobacterium lub biolistycznego bombardowania mikrocząsteczkami lub transformacji protoplastów. Produkty są oparte na CRISPR/Cas9 typu IIA pochodzącym z Streptococcus pyogenes. Natywna sekwencja kodująca Cas9 została zoptymalizowana pod kątem ekspresji odpowiednio u roślin jednoliściennych i dwuliściennych. Konstrukty monocot Cas9 zawierają promotor monocot U6 do ekspresji sgRNA, a konstrukty dicot Cas9 zawierają promotor dicot U6. Markery selekcji roślin obejmują gen oporności na higromycynę B, gen fosfotransferazy neomycyny oraz gen bar (acetylotransferaza fosfinotrycyny).

Proces generowania zmutagenizowanej linii roślin CRISPR/Cas9. c, kontrola; m, zmutagenizowany; RE, enzym restrykcyjny. CELI i T7E1 to endonukleazy DNA używane w teście ankietowym.
Gotowe do użycia plazmidy ekspresyjne Cas9 i Guide RNA (gRNA) dla roślin jednoliściennych i dwuliściennych
- Nasze roślinne produkty CRISPR/Cas9 są przeznaczone dla transformacji roślin za pośrednictwem Agrobacterium lub biolistycznego bombardowania mikrocząsteczkami lub transformacji protoplastów
- Zoptymalizowane kodonowo białko Cas9 i gRNA są wyrażane z jednego wektora i dostarczane jako gotowe do użycia DNA o jakości transfekcji.

Podstawowa struktura konstruktów CRISPR/Cas9 do transformacji za pośrednictwem Agrobacterium.

Podstawowa struktura konstruktów CRISPR/Cas9 dla biolistyki lub transformacji protoplastów.
Lista produktów CRISPR/Cas9 dla roślin |
|---|
Niestandardowe wektory CRISPR GUS |
|---|
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?