PCR技術をベースに構築された定量PCR(正式には、定量リアルタイムPCR、qPCR)検出は、サンプル中の出発物質の量の推定を可能にします。反応が進むにつれて生成物が検出されるため、qPCRの分析のダイナミックレンジは従来のエンドポイントPCRよりはるかに広く、1コピーから10程度11のコピーまでを1回の実行で検出できます。定量リアルタイムPCRとその後に実施されるアンプリコン検出は、ゲル電気泳動などのPCR後の操作を不要にして、交差汚染のリスクを大幅に低減する閉管形式で行われます。
このページではqPCRの基本原理について説明します。また、一般的な検出化学の仕組みについては定量PCRおよびデジタルPCR検出法で説明します。
基本原理
qPCRを実行すると、各増幅サイクルの間に存在する核酸量の間接指標として蛍光レポーター色素が使用されます。蛍光シグナルは、反応の反復フェーズの間に指数関数的に蓄積されるPCR生成物分子(アンプリコン)の量に比例して増加します(ポリメラーゼ連鎖反応を参照)。レポーター分子は、二本鎖DNA(dsDNA)結合色素、プライマー結合色素、または色素標識オリゴヌクレオチドに分類され、これらはプローブと呼ばれます(定量PCRおよびデジタルPCR検出法)。
SYBR® Green IなどのDNA結合色素の使用は、最も簡単な検出化学の代表的な形式です。溶液中に拡散する、または一本鎖DNA(ssDNA)のみが存在するSYBR® Green I色素は、低シグナル強度で発光します。PCRが進行し、dsDNAの量が増加すると、より多くの色素がアンプリコンに結合するため、シグナル強度が増加します。
その他に、プライマー間に存在するテンプレートの特定の領域に結合するプローブ(または検出化学に応じた2つのプローブの組み合わせ)を使用する方法があり、検出の特異性をdsDNA結合色素よりも高いレベルまで引き上げることができます。最も一般的に使用されるプローブの形式は、両末端標識プローブです(DLP、加水分解プローブまたはTaqMan®プローブとも呼びます)。DLPは、6-FAM™および3’クエンチャーなどの5’蛍光標識の付いたオリゴヌクレオチドで、例えばいずれかのダーククエンチャー(BHQ®1またはOQ™)です(定量PCRおよびデジタルPCR検出法)。2つのプライマー間のテンプレートにハイブリダイズするように設計されたこれらのプローブは、固有の5’~3’エクソヌクレアーゼ活性を有するDNAポリメラーゼと組み合わせて使用されます。DLPが溶液中に拡散している場合は、レポーター色素はクエンチャー部分に接近しているためシグナル強度は低レベルです。反応中にはより多くのテンプレートが生じるため、より多くのプローブがテンプレートにハイブリダイズされて、進行するDNAポリメラーゼの5’~3’エクソヌクレアーゼ活性によって順次切断されます。蛍光シグナルの強度は、5’レポーター色素が溶液中に放出されるのに従い増加します。異なるレポーター色素で標識付けされたプローブを使用すると、1回の(マルチプレックス)反応で複数の標的を同時に検出および定量化できます。
代表的なqPCRランは、複数の温度下での反応の反復サイクルからなります(サイクル手順3.1)。このプロファイルは、dsDNA結合色素、Molecularビーコン、またはScorpion®プローブをqPCRの検出化学として選択した場合によく使用されます。プライマー伸長法は、ほとんどのDNAポリメラーゼの処理能力が最適になる72℃のときに最も効果的です。72℃では、1秒あたり約100塩基の速さで重合が起こります。ただし、短いテンプレートを増幅する場合は、さらに低い温度でも処理能力は十分です。qPCRアンプリコンは一般に、従来のPCR生成物よりも短いため(200塩基未満)、両末端標識プローブを使用する場合の伸張には、60℃のシングルステップアニーリングと組み合わせることがほとんどです(サイクル手順3.2)。
サイクル手順3.1
dsDNA結合色素、Molecular ビーコン、またはScorpion®プローブの検出を用いた標準的なqPCRプロファイルの例を説明します。
- 初期の変性:反応温度を95℃まで上昇させ、サンプルを2~10分間(時間はポリメラーゼ酵素のホットスタートの機序に依存)培養し、すべての複合標的(dsDNA)が分離されて一本鎖になり、増幅に使用できることを確認します。
- サイクル:
- 変性:反応温度を95℃まで上昇させて10秒間維持し、すべてのdsDNAを融解させます。
- アニーリング:温度を60℃まで降下させて30秒間維持し、プライマーとプローブ(含まれる場合)のテンプレートへの結合を促進します。
- 伸張:続いて、TaqDNAポリメラーゼの処理能力が最適になる72℃まで温度を上昇させると伸張が起こります。伸張の時間はアンプリコンの大きさによって異なります(1 kbあたり30秒)。望ましいアンプリコンの長さと使用する酵素によっては、期間を延長します。qPCRアンプリコンは短いため、通常は5~30秒です。
- 変性:反応温度を95℃まで上昇させて10秒間維持し、すべてのdsDNAを融解させます。
- 反復:ステップ1~3を繰り返します。通常は40サイクルです。
サイクル手順3.2
DLP検出のための2ステップサイクルqPCRのプロファイルの例を説明します。
- 初期の変性:温度を95℃まで上昇させて、反応を2~10分間(時間はポリメラーゼ酵素のホットスタート特性に依存)インキュベートします。
- サイクル:
- 変性:反応温度を95℃まで上昇させて10秒間維持し、すべてのdsDNAを融解させます。
- アニーリング・伸張:温度を60℃まで降下させて30秒間維持し、プライマーのテンプレートへの結合を促進します。この温度はDNAポリメラーゼの活性に十分なため、続いて伸張が起こります。
- 反復:ステップ1~2を繰り返します。通常は40サイクルです。
反応の全過程にわたる蛍光の変化が、リアルタイムPCRサーマルサイクラーによって測定されます。専用のリアルタイムPCR装置は、温度サイクル機能と光学ユニットを組み合わせたものです。光学ユニットは、蛍光色素分子を励起するために適した波長の光を出力して、結果として生じる発光を検出します。最近の装置にはディスプレイ画面を搭載しているものや、コンピューター用モニターとともに使用するものが多く、反応の進行の追跡が可能です(図3.1)。
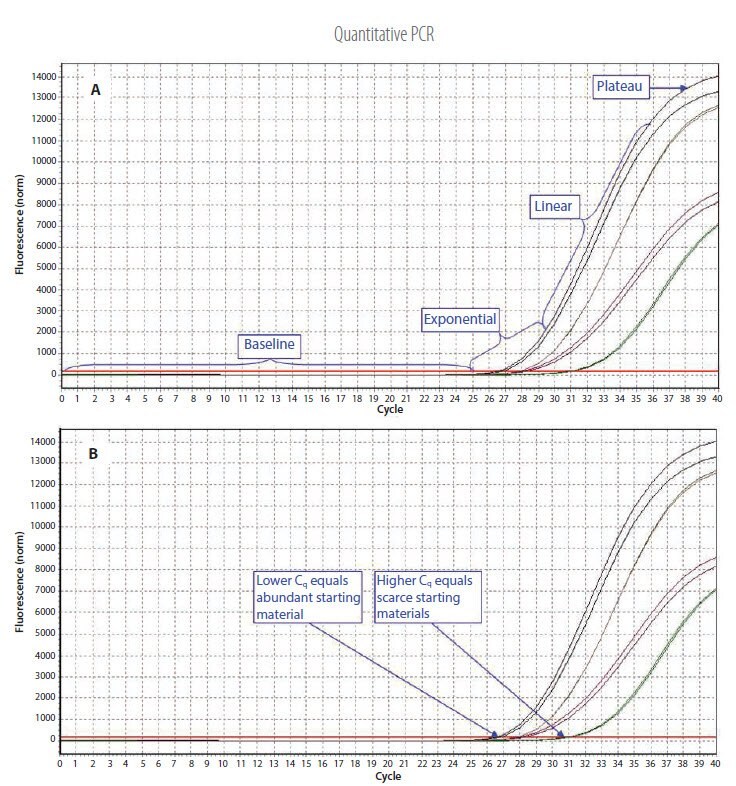
図3.1.qPCRアッセイデータの例。A) 各反応フェーズ。ベースライン:テンプレートの初期濃度が低いため、蛍光強度は検出できないほど低く、バックグラウンドシグナルのみが見られる。指数上昇期:標的の収量が検出閾値(赤色で示す閾値ライン)に達すると、反応の過程は指数上昇期に入る。直線期:テンプレートの濃度が上昇するにつれて利用可能なDNAポリメラーゼの濃度が低下し、反応速度は減少する。プラトー期:反応は最大収量に達する。B) 各反応は、蛍光が上昇して閾値を初めて超えるサイクル値(定量サイクル(Cq)と呼ばれる)によって特徴づけられる。出発物質が豊富にある場合は、早いサイクルで増幅が観察されてCqは低くなる。出発物質が少ない場合は、遅いサイクルで増幅が観察されてCqは高くなる。蛍光量、つまりCq値と増幅生成物とのこの相関関係から、広いダイナミックレンジにわたってテンプレートを定量できる。
gDNA標的の定量・分析
定量リアルタイムPCRは、gDNA標的の分析に容易に適用できます。この種の研究には、遺伝子型決定/SNP
決定、メチル化分析、遺伝子組換え配列のスクリーニング、または挿入と欠失のモニタリングなどがあります。
mRNA転写の定量・分析
qPCRの一般的なアプリケーションには、例えば対照サンプルと処理サンプル間で対象遺伝子のmRNA濃度を比較する、遺伝子発現解析があります。mRNAは、2ステップまたは1ステップRT-qPCR処理によって定量できます(RT反応の詳細は逆転写定量的リアルタイムPCRを参照)。
2ステップRT-qPCR
個別のチューブで逆転写とqPCR反応を順次行います。全RNAか、またはそれほど一般的ではない
ポリ(A)+ RNAが出発物質として使用され、逆転写酵素を用いたオリゴ-dTプライマー、ランダムプライマー、オリゴ-dTプライマー/ランダムプライマーの混合、
または遺伝子特異的プライマーからの伸張によってcDNAが産生されます。その後、この反応のアリコートをqPCRに添加します。
1ステップRT-qPCR
実験セットアップは、逆転写およびqPCR反応を1本のチューブの中で組み合わせるだけです。
これ以上のサンプル処理は不要のため、汚染が入り込む可能性が低く抑えられます。
ncRNA転写の定量・分析
ほとんどのノンコーディングRNAは100ヌクレオチドより長いため、mRNA解析の場合と同じ技術を用いたRT-qPCR
によって検出および定量できます。一方で、マイクロRNAやpiwi RNAなどの短いノンコーディングRNAの基本長は、単独のPCRプライマーの長さと同等です。そのため、qPCRを実行する前にこれらの短いRNAを伸ばす技術が必要です。市販されているシステムでは、主に次の2つの手法が採用されています:1) ステムループRTプライマーの使用、2) ポリAテール法の後オリゴ-dTアダプタープライマーによるRT(図3.2)。Casoldiらが開発した別の手法(市販されていません)では、サンプル中の全RNAへのアダプター分子の連結反応と特異的なプライマー設計を使用して、所望の標的同士を区別します1,2。
ステムループRTプライマーの手法は、Applied Biosystems(現在のLife Technologies/Thermo)がTaqMan®プローブベースqPCRの前処理のために開発し、販売しています3。図3.2Aに示すように、RTプライマーの5’-末端は、自身の3’-末端に含まれるいくつかのヌクレオチドの領域と塩基対を形成することで、不対ループによって分離された塩基対形成ステムを生成できます。RTプライマーの3’-末端に位置する最後の数個を除くすべてのヌクレオチドは多用途です。すなわち、これらはすべてのmiRNAプライマーの中に同じ配列を有します。ステムから3’を伸張している最後の数個のヌクレオチドは、標的miRNAの3’-末端に対して相補的です。miRNAテンプレートに沿ったプライマーの伸張は、
miRNA特異的フォワードプライマーおよびユニバーサルリバースプライマーによって増幅できるcDNAを生成し、後者のプライマーは
ステムループRTプライマーの5’-末端に対して相補的です。
MystiCq®ブランドを含む、その他のすべての市販されているmiRNA qPCRの方法は、ShiおよびChiangが述べているようにポリAテールをmiRNAの伸張に使用します4。テンプレート非依存性酵素のポリ(A)ポリメラーゼ(PAP)は、ATPからRNAの3’-末端へのアデノシン残留物の転写を触媒します。その後、図3.2Bに示すようにオリゴ-dTプライマーを用いてRTを実行できます。5’-末端にアダプター配列を含むオリゴ-dTプライマーは、その後のqPCRを、特異的miRNAに対して相補的なフォワードプライマーと、アダプター配列に対して相補的なリバースプライマーにより実施できるようにします。
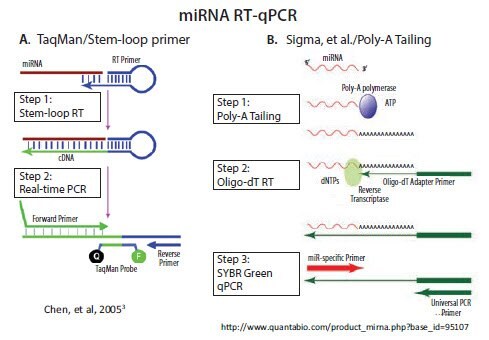
図3.2.miRNAのRTおよびqPCRの市販されている方法は2つあります。A) ハイブリダイゼーション後のRTステップをmiRNAの3’末端にプライミングするために、ループプライマーを使用する。ループによるPCR伸張により、結合miRNAと、増幅に十分な長さを有するユニバーサル配列が得られる。 B) ポリAテールをmiRNAに添加することにより、オリゴ-dT領域とユニバーサルプライミング配列からなるプライマーのプライミング部位が得られる。Oxford University Pressによる核酸の研究。Oxford University Pressから許可を得た上で、Copyright Clearance Centerから入手した書籍/電子書籍の再利用の形式で複製。
市販されているいずれの手法にも、メリットとデメリットがあります。ステムループプライミングでは2ステップRT-qPCRが可能ですが、ほとんどのポリAテール法はqPCRの前にRTのための追加ステップが必要です。ポリAテール法とRTを単一反応に組み合わせた.2ステップPAPテール/RT-qPCRの製品もあります。しかし、この手法では検出感度が低くなるおそれがあります(社内結果は未公開)。反対に、ポリAテール法とRTを順次行う手法はcDNAの安定したプールを生成します。これを用いればマイクロRNA、mRNAなどのあらゆるRNAをすぐに、または後でいつでも検出できます。ステムループ/TaqMan®の手法では、各miRNAを検出するために異なるプライマーが必要です。複数のステムループプライマーを同じRT反応に添加でき5、また複数のmiRNAのためのRTプライマーのプールをLife Technologiesから購入すればマルチプレックスRTを実行できます。しかし、最近発見されたmiRNAのqPCR検出を可能にするオプションはなく、同じcDNAからmRNAを検出するためのqPCRを可能にするオプションもありません。Castoldiらが述べているシステム1,2は、mRNA、前躯体、および十分に処理されたmiRNAの分子を同じ全RNAサンプルから検出する方法が得られるという点で独特です。
qPCRの要件
装置
特定の範囲のアプリケーションをサポートするqPCR装置が数多く設計されています。例えば、複数の384ウェルプレートの自動装填機能を用いるABi 7900の高スループットな装置と、Illuminaが製造・販売している単独の48ウェルプレートをサポートするEco装置では、性能が大きく異なります。従って、最適な装置は研究のニーズに適合する装置です。また、装置に付属するソフトウェアは使いやすく、必要な機能をほとんど実行でき、また下流の統計学的解析ソフトウェアパッケージでの操作を可能にする柔軟なデータ出力を備えるものを選択すると良いでしょう。これにより、トレーニングに要する時間、すなわち初めて結果が得られるまでの時間が短縮されます。その他の必要な特性には、絶対的な均一性(96ウェルの反復全体で1 Cqの最大絶対偏差が2倍)を有するPCRブロック、および広範囲の波長にわたりできるだけ高感度かつ均一に発光を励起および検出できる光学システムがあります。これにより、蛍光色素分子の選択肢が広がり、マルチプレックスが可能になります。考慮すべきその他の特性は、特定の消耗品にかかる運用コストです。例えば、反応のために標準のマイクロタイタープレートを使用しない場合もあれば、非標準型の装填プレート/チューブが便利な場合もあります。
テンプレート
qPCRを開始するために必要な標的核酸のコピーは、ごくわずかです(約100 pgのgDNAまたはcDNAに相当)。反応阻害物質による汚染を最小限に抑えるために、開始テンプレートの数量を、正確な定量を得るための必要最小量にしておく必要があります。出発物質がRNAの場合には、プライマーの設計とDNアーゼIの処理によってgDNA汚染が生じ、シグナルが減少するおそれがあります。
プライマー
dsDNA結合色素を使用するか、またプローブベースの検出化学を使用するかに関わらず、高品質プライマーの設計はqPCRにおける最も重要な実験前ステップの1つです。アッセイ設計の詳細については、PCR・qPCR・dPCRアッセイ設計をご覧ください。感度と特異度を最大限に高めるには、Locked Nucleic Acidなどの必要な修飾を含めなければなりません(qPCR検出化学)。
プローブ
アンプリコン検出の手法としてプローブを用いる場合、プライマーダイマーや非特異的生成物は検出されませんが、反応効率が低下するおそれがあるため、これらを避ける必要があります。感度と特異度を最大限に高めるには、アプリケーションに適したプローブの種類を選択し、さらにLocked Nucleic Acidなどの必要な修飾を含めなければなりません(qPCR検出化学)。
dNTP
標準のPCR・qPCRマスターミックスには、dATP、dCTP、dGTPおよびdTTPが含まれています。ただし、ミックスの中にはdTTPの代わりにdUTPを含むものもあります。dUTPによって実行した予備反応からの生成物には、チミンではなくウラシルが含まれています。そのため、これらの生成物はウラシル-DNA-グリコシラーゼ(UNG)による切断に耐性がありません。従って、その後の反応をUNGにより事前培養することで、反応間での汚染の持ち越しが回避されます。効率を上げるには、ラボ内のすべてのPCRにdUTPを使用する必要があります。
マグネシウム
逆転写酵素、Taq DNAポリメラーゼおよびTaq DNA 5’~3’エクソヌクレアーゼ活性には、塩化マグネシウム(MgCl2)が必要です。DLPを含む反応に最適なMg2+濃度は、ほとんどの場合3~6 mMです。主なマスターミックスにはMgCl2が含まれていますが、濃度の最適化が必要な場合があるため、マスターミックス製品にはMgCl2の追加チューブが含まれていることがほとんどです(アッセイの最適化・バリデーションを参照)。場合によっては、低濃度の使用を可能にするためにMgCl2を含まない反応ミックスが必要なこともあります。例えば、Scorpions®プローブ検出を使用する場合などが含まれます。
逆転写酵素
RT-qPCRで成果を上げるには、高収率のcDNAを確保しながら高温での活性を維持する逆転写酵素が不可欠です。高温での性能は、重要な二次構造を有するRNAの領域を不安定化して、ハイブリダイゼーションとその後の増幅に利用できるようにするために役立ちます。1ステップRT-qPCRを実行する場合は、高温性能により溶融温度(Tm)の高い遺伝子特異的プライマーの使用が可能になり、反応の特異性が向上します。2ステップの手順を実行する場合は、酵素によって、直線的に比例するcDNA収量がRNAから得られるようにすることが重要です(RT評価の詳細は逆転写を参照)。
Taq DNAポリメラーゼ
適切なポリメラーゼ酵素の選択は、RTに最適な逆転写酵素の選択と同じように重要です。自然のTaq DNAポリメラーゼに伴う基本的問題は、低温では酵素に活性が残る点にあります。非特異的プライマー結合は、この残留ポリメラーゼ活性の結果として非特異的生成物の形成を引き起こします。抗体ブロックまたは化学的ブロックのTaq DNAポリメラーゼ(「ホットスタート」)が、高温で変性ステップが始まるまで酵素活性を回避することによってこの状況を是正するために有効です。
バッファー
バッファーや反応マスターミックスには通常、dNTP、Taq DNAポリメラーゼ、MgCl2および安定化剤が含まれています。検出化学、装置および反応要件によっては、SYBR® Green I色素、ROX™、フルオレセインおよびインサートローディングダイを含めることもあります(Loading Control Dyes)。PCRバッファーの成分と安定化剤は、メーカーの独自技術によるものがほとんどです。別々に購入すれば各構成要素を反応のなかで個別に最適化できるため、最大の柔軟性が得られます。しかし、これとは反対に、構成要素をマスターミックスとしてまとめて購入すれば柔軟性は低下しますが、バッチの一貫性と利便性が高まるとともに、ピペット操作が減るため操作ミスや汚染の危険性が減ります。
Loading Control Dyes
一部のリアルタイムPCRサーマルサイクラーでは、光学システムの変動を制御するため、およびシグナル強度の違いを標準化するために、ROX™などの loading dyeを各反応に含める必要があります。同じように、SYBR® Green I色素アッセイ(このバックグラウンドは非常に低い)を用いる場合に、仮想バックグラウンドを生成するために初期のフルオレセインシグナルを要するサーマルサイクラーもあります。これらは、マスターミックスに含めて供給されるか、または個別のコンポーネントとして供給され、適切な濃度の使用を可能にします。
MIQEガイドライン
qPCRを使用すると、大量のデータを極めて容易に生成して、これを定量結果としてすぐに解釈することができますが、これは明らかに問題を引き起こします。つまり、アッセイの設計や実行、またはデータ解析が不適切な場合にはアーチファクトを招き、これを識別するための品質管理や追加分析が必要になるため、これが誤りの元になるおそれがあります。適切な品質管理を伴わない一見したところ意味のあるようなデータを発表すれば、その結果としてのレポートは生物学的または化学的意味をなさないか、最悪の場合は誤った内容になります。
サンプル収集からqPCRデータ解析に至るqPCR実験工程の各ステップでは、容易にデータ変動が起こります6。そのため、サンプルの選択と処理、核酸の抽出(品質の差異を招き、qPCR標的検出の再現性の低下を引き起こすおそれがある)、アッセイの設計と最適化(アッセイの効率と感度の差異を引き起こす)およびデータ解析(若干の主観的な解釈と、適切な統計学的手法の適用を要する)の際に、ばらつきが生じるおそれがあります。科学者である読者に提供される情報には、批評を受けるに値する適切な実験情報が含まれていることが極めて重要ですが7、当然のことながら常にこれが実施できているとは限りません8。この問題に対処するのが「Minimum Information for Publication of Quantitative Real-time PCR Experiments」(MIQE:リアルタイム定量PCR実験のための最小限の情報)ガイドライン9です。MIQEガイドラインの目的は、アッセイに関する必要最小限の情報を指定することにより透明性を向上させて読み手が技術的妥当性を評価できるようにすること、および新しいqPCRアッセイの設計、最適化およびバリデーションのガイドラインを示すことにあります。
MIQEは、実験計画、サンプル、核酸、逆転写、標的、プライマーとプローブ、アッセイの詳細、PCRサイクル、およびデータ解析の9セクションからなります(MIQEページからダウンロードできます、表1)。すべてのパラメータは、実験計画、最適化およびバリデーションの過程で取得する必要のある情報と関連性があります。市販のアッセイや、文献に発表されているアッセイを用いる場合でも、最適化とバリデーションを行う必要があります。MIQEのチェックリストで「必須」に分類されている要素は、qPCRアッセイを十分に説明するために不可欠と考えられる情報です。一方で、「推奨」に分類されているその他の要素は、有用な情報ですがこれらがなくてもアッセイは再現可能で、公表されるデータは評価に値することを示します。重要なことは、PCRの効率、分析感度、および個々のアッセイの特異性の報告は必要不可欠であり、研究者がラボ内の条件を使用してこれらを定めるべきであると認識することです。デジタルPCR(デジタルPCRを参照)を実行する場合には特に考慮すべき事項があり、これらは最近公表されたデジタルPCR MIQEで取り扱われています10。
MIQEとの適合
Minimum Information for Publication of Quantitative Real-Time PCR Experiments(リアルタイム定量PCR実験のための最小限の情報)
aAmerican Association For Clinical Chemistry, Inc.による臨床化学Copyright 2009。American Association For Clinical Chemistry, Inc.の承諾を得てCopyright Clearance Centerのインターネット掲示の形式で複製。
bすべての必須情報を原稿で提出する必要があります。推奨情報は、必要に応じて提出してください。RTPrimerDBに収録されているプライマーの場合は、RTPrimerDBからqPCRターゲット、オリゴヌクレオチド、プロトコル、およびバリデーションに関する情報が得られます。
c非逆転写アッセイによるDNA不在評価は、RNAを初めて抽出する場合に必須です。サンプルがDNAフリーとして検証されている場合、非逆転写制御の実施は推奨されますが、必須ではありません。
dプローブ配列の開示が非常に望ましく、強く推奨されます。しかし、市販されている設計済みアッセイのすべての供給業者がこれらの情報を提供しているとは限らないため、必須要件ではありません。このようなアッセイは使用しないようお勧めします。
参考文献
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?