遺伝子発現解析へのアプローチの一つは、遺伝子のmRNA濃度の測定です。この解析には、異なる転写物間の半減期の差、転写物の時間的なパターン、mRNAとタンパク質との間の相関の欠落など、いくつもの課題があります。PCRによる方法を用いてRNAを解析するため、cDNAが逆転写(RT)により産生される必要があります。このプロセスは逆転写酵素およびdNTPsを利用します。
RTのステップは、多数の転写物の全体的cDNA representationが産生されるように(通常、2ステッププロトコルを介して)または対象RNAのみがcDNA(通常、1ステッププロトコルに従う)に変換されている遺伝子特異的なアプローチで、全RNAに対し実行されます。
2ステップRT反応はインプットRNAおよびcDNA量について必ずしも線形性ではないことは示されているため1、RT反応において抽出され含まれているトータルRNA量を測定および制御することが重要です。RNA濃度の測定は不確定であり、絶対値は測定に使用する機器またはシステムに依存しています。 図8.1でみられるように、Nanodrop、従来のUV分光測色法、Ribogreen、the Agilent 2100 BioAnalyzer、またはBio-Rad Experionを用いたときの、5つのRNAサンプル(A~E)の濃度測定値の間には大きな変動がみられます(サンプル精製および品質評価参照)。下流のRTおよびPCRを阻害するEDTAの存在(サンプルDおよびE)、および分解(サンプルC)は、使用するシステムに応じて濃度測定に異なる影響を及ぼすことに注意します。この観察結果は、下流の反応でサンプルを使用する前に追加の品質管理ステップを実施する重要性を示しています(サンプル精製および品質評価参照)。
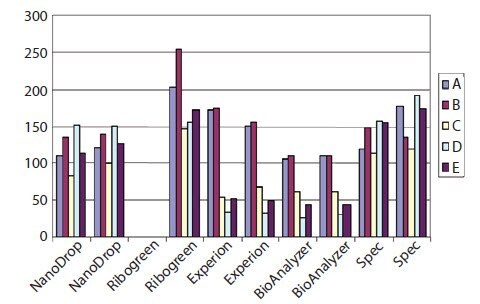
図8.1.5つのサンプルのトータルRNA(A~E)濃度を、Nanodrop、従来のUV分光測色法、Bio-Rad Experion、the Agilent 2100 BioAnalyzer(すべて2連測定)またはRibogreen(1連測定)を用いて測定しました。以上のように、絶対濃度と相対濃度はサンプル間で異なりました。これは、サンプルCが劣化しサンプルDおよびEにEDTAが含まれたためであり、これによりExperionおよびBioAnalyzerシステムでの測定が不正確だったためです。
逆転写の直線性
トータルRNAの相対濃度は、RTの効率性および既定の転写物から産生されたcDNAの濃度に影響を与える可能性があります。したがって、RTシステムが線形応答を有することが確認されていない限り、すべての2ステップのcDNA合成反応に同じまたはほぼ同等の濃度のRNAを含めることが望ましいです。図8.2でみられるように、従来のRTプロトコルを用いると、インプットRNAの100倍希釈は、検査したテンプレートに対するcDNA収量において100倍相当の差をもたらしません。興味深いことに、このデータは2連RT反応での2連qPCRランで示されています。示されているとおり、直線性の欠落は2つのRT反応の間で再現性があります。

図8.2.トータルRNAは100倍希釈され、2ステップのランダムプライミングにより逆転写されており、2つの独立したRT反応が行われました。各RT反応の2連qPCRでβ-アクチンが検出されました。このRTは再現性がありますが、cDNA収量はインプットRNA濃度に比例しません。したがって、実験上の制約によりRNA濃度の変動がRTに含まれるよう要求される場合、プロトコルおよび試薬の組み合わせが線形応答をもたらすことの確認は重要です。
図8.3に示す例では、ReadyScript® RT試薬(RDRT)を用いて、2倍および10倍の段階希釈から、2ステッププロトコルならびにオリゴ-dT(O4387)およびランダムプライミング(以下に示す)の組み合わせにより、トータルRNAを逆転写しました。両方の希釈系列からCANX遺伝子が検出され、インプットRNA濃度に正比例していました。

図8.3.ReadyScript® RT試薬(RDRT)を用いて、2倍および10倍の段階希釈から、トータルRNAを逆転写しました。両方の希釈系列からCANX遺伝子が検出され、インプットRNA濃度に正比例していました(EMBL Advanced qPCR Workshopに参加した学生グループのデータ)。
逆転写のプライミング
逆転写の開始のために使用されるプライマーの選択は、RT-qPCRの結果に大きく影響する可能性があります。1ステップ
RT-qPCRでは、遺伝子特異的プライマーを使用します。2ステップアッセイを実施する場合、逆転写用遺伝子特異的プライマー、オリゴ-dT(O4387)、ランダムヘキサマー、ノナマー、デカマー、ドデカマー、ペンタデカマー2 またはオリゴ-dT(O4387)およびランダムプライマーの組み合わせを使用することができます。遺伝子特異的プライミングは通常、各ターゲットRNAに対し別々の反応で行います。これらの別々の反応は効率性が異なる可能性があるため、RNA濃度の比較を複雑にします。一方、遺伝子特異的プライマーを使用する場合、すべてのRT産物は標的遺伝子をコードし、非特異的RTプライマーにより検出できなかったきわめて少量のmRNAを定量化できる可能性があります。遺伝子特異的プライマーにより発生しうるRTにおける高いアッセイ間変動を回避するため、非特異的プライマーを使用してcDNAプールを生成することができます。その後、cDNAプールからのアリコートを用いて、各ターゲットに対し別々のqPCRアッセイを実施することができます。すべてのqPCRターゲットがポリアデニル化mRNAの3’-末端付近である場合、オリゴ-dT(O4387)は適切なプライマー選択です。一方、qPCRターゲットが3’末端から数キロベース以上離れている場合、またはRNAがポリアデニル化されていない場合、ランダムプライマーがより信頼性の高い検出をもたらします。qPCRターゲットの相対的な3’位が変動する、または目的の転写物にポリアデニル化RNAおよび非ポリアデニル化RNAの組み合わせが含まれている場合、オリゴdT(O4387)およびランダムオリゴマーの混合物により最良の結果が得られます
2ステップRT反応に対する逆転写のプライミング
2ステップRTプライミングには、2つのアプローチ、すなわちオリゴ-dT(O4387)およびランダムプライミングが汎用されています。オリゴ-dT(O4387)法は、mRNA分子の大多数の3’末端に存在する、poly-A tailへのオリゴdT(通常15mer)のハイブリダイゼーションに依存しており、mRNAをプライミングし選択的に逆転写します。
概念的には大変シンプルですが、このアプローチは以下の課題を伴います:オリゴ-dT(O4387)プライマー結合は、RTに使用する反応温度ではmRNAに特異的ではないため、オリゴ-dT(O4387)はRNAの他の領域に非特異的に結合します。さらに、これらの分子のATrich領域はオリゴ-dT(O4387)によりプライミングされているため、rRNAの伸長も検出されています。一部のmRNAには、ヒストンをコードするmRNAのように、poly A tailが含まれておらず、その結果生じたcDNA内に示されない可能性があります。
第2の方法はランダムプライミングを使用します。ランダムプライマーはランダム配列で構成されており、多くはヘキサマー(6mer)またはノナマー(9mer)で構成されます。これらはRT反応をプライミングするのに使用されており、様々な長さのcDNAフラグメント合成をもたらします。これらは元のRNAを示します。ランダムプライマーは転写物の長さに沿ってハイブリダイゼーションし、オリゴ-dT(O4387)または遺伝子特異的プライミングと比べて二次構造に許容性を示す傾向があります。
それぞれの技術の利点から利益を得るため、一部のプロトコルは両方のタイプのプライマーの組み合わせを要求しています。また、ターゲット配列に対する特異的なプライマーは、2ステップRTプロトコルにも使用することができますが、1ステップで使用されることのほうが一般的です(下記参照)。
1ステップRT反応の逆転写のプライミング
1ステップRTプロトコルでは、遺伝子特異的プライマーが単独のターゲットを逆転写するのに使用されます。遺伝子特異的プライマーのデザインは重要です。RT反応温度で予測した場合、mRNAターゲットのオープンでアクセス可能な領域内にある必要があります。これらの状況下では、インプットRNAとcDNAとの間に直線関係が存在します(図8.4)。このプライマーはPCRプライマーに共通している可能性があります(通常は共通しています)。

図8.4.トータルRNAは連続10倍希釈系列で希釈され、GAPDH検出のために1ステップRT-qPCRが実施されました。各反応は3回実施しました。この結果、RNA濃度とcDNA収量との間に直線関係が認められました(Prof. Stephen Bustin, Anglia Ruskin University, UKのご厚意により画像を掲載)。
逆転写は変動が大きいプロセスであるため、すべてのステップは反応要素をできる限り安定に保つよう考慮する必要があります。1ステップRT-qPCR反応の実施により、必要なピペッティングステップの数を減少させ、エラーを減らすことができます。したがって、小さな差を測定する必要があり精度が最優先である場合、要求された結果を達成するのに最も良い方法です。しかしながら、反対の考察としては、標的遺伝子(GOI)と参照遺伝子の比率の決定(データ分析参照)には、2ステップ反応からの単一のcDNAではなく、2つの別々の1ステップRT反応が必要です。2つのターゲットは、マルチプレックスqPCRアプローチを使用して検出することはできません。
逆転写の効率
一般的に、RT反応におけるすべてのRNA/mRNAはcDNAに変換され、すべての転写物は1:1の比率で、または開始RNA濃度に比例して変換されると想定されています。近年、これらの仮定のそれぞれを調査するために研究が行われています。cDNAに変換されるRNA量はきわめて変動が大きいことは明らかです。2ステップRTプロセスは変動が大きく、特にRNA濃度、酵素、バッファー組成およびプライミングプロトコルに依存しています。このプロセスは変動が大きいため、できる限り多くの一定の条件を維持することが重要です1,3,5。
2ステップRT反応では、一般的に、同じインプットRNA濃度を目指し、プライミング条件、RT酵素およびバッファーを一定に維持することが必要です。一定のインプット濃度を決定できない場合、1ステッププロセスを使用する、ポリエチレングリコール(PEG)6などのキャリアを含める、またはReadyScript® RTのような線形応答をもたらすことが検証済みの市販キットを選択することが望ましいです。
プライミング戦略の選択は、トータルRNAサンプルからの絶対収量および異なるcDNAターゲットの比率の両方に影響を与えます。図8.5 は、制御された酵素分解を受けたRNAサンプル中の3つの異なる遺伝子についてcDNAコピー数の収量を示しています(ヒストグラムの色で表示)。様々なプライミング法が様々な絶対的なコピー数およびサンプル間の比をもたらすことは明らかです。さらに、遺伝子特異的なプライミングはオリゴ‑dTプライミングと比べてRNA分解に耐性があるようにみえます。

図8.5.トータルRNAを、所定の時間(1、2.5、5および7.5分)の間、手でインキュベートしました。遺伝子特異的、ランダム、またはオリゴ-dT(O4387)プライミングにより、cDNAを調製しました。3つの遺伝子のコピー数を測定し、紫色、青緑色、橙色のヒストグラムで示しました。様々なプライミング戦略がオリゴ-dT(O4387)プライミングを用いた各遺伝子検出に影響し、遺伝子3(橙色)の不検出をもたらしたことは明らかです。このことはRNAサンプルにおいて遺伝子特異的プライミングを用いて検出されているため明白です(Prof. Stephen Bustin, Anglia Ruskin University, UKにより提供されたデータ)。
RT反応に使用した温度は、特異性、特に遺伝子特異的プライマーをハイブリダイゼーションする場合に影響を及ぼす可能性があります。強力な3’-二重鎖を形成することができるプライマーは、低い温度であるほどより速やかにハイブリダイゼーションすると考えられます。RT酵素はDNAテンプレート上でDNAプライマーから延伸するため、プライマー-ダイマー形成がRTステップ時に開始する可能性があります。RTインキュベーション温度を酵素が完全な活性を示す最高温度まで上昇させる、または高温耐性酵素を使用することにより、プライマー-ダイマー産物量を減少させることがあります。図8.6 で使用されたプライマーは、45℃でモロニーマウス白血病ウイルス逆転写酵素(MMLV-RT)を用いてRTを実施したところ、37℃でRTを実施した場合(図8.6A)と比べて、1ステップRT-qPCR後に非特異的な産物と比べて有意に特異的な産物をもたらしました(図8.6B)。同様に、RTのために非特異的プライマーおよびqPCRのためにHot Start Taqポリメラーゼを用いた2ステップRT-qPCR の実施は、3’-二重鎖を形成することができる遺伝子特異的プライマーを用いた1ステップRT-qPCRと比べて、プライマー-ダイマー産物(図8.6D)の減少をもたらしました(図8.6B)。
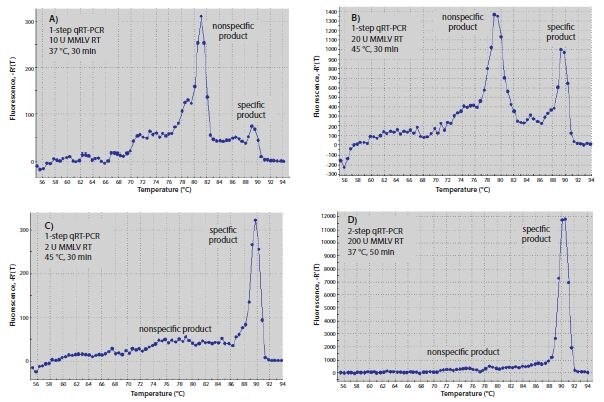
図8.6.RTの最適化。(A~C)1ステップRT-qPCRを用いて産生したRT-qPCR産物の解離曲線または(D)2ステップRT-qPCRを用いて産生したRT-qPCR産物の解離曲線。反応A~Cは各々、10 μL SYBR® Green JumpStart™ Taq ReadyMix™、0.02 μL標準色素、0.4 μMの両方の遺伝子特異的プライマー、および10 ngヒトトータルRNAを最終容量20 μLに含有しています。遺伝子特異的プライマーは、c-fos(Accession番号:NM_005252)に対する5’-CGGGCTTCAACGCAGACTA-3´および5´-CTGGTCGAGATGGCAGTGA-3´でした。また、反応AおよびBは、20ユニットのMMLV-RTを含有していますが、反応Cは2ユニットです。反応AはqPCR前に37℃で30分インキュベートしましたが、BおよびCはqPCR前に45℃で30分インキュベートしました。Dでは、RT反応は、1x MMLVバッファー(製品番号D8559)、0.5 mM dNTPs、1 μM オリゴ-dT(O4387)、0.8ユニット/μL RNアーゼインヒビター、200 ユニット MMLV-RT、10 ngヒトトータルRNAを最終容量20 μLに含有しています。反応は25℃で10分、37℃で50分、80℃で10分、インキュベートしました。RT反応産物2 μLをqPCR(1ステップ反応では10 μL SYBR® Green JumpStart™ Taq ReadyMix™、0.02 μL標準色素、0.4 μMの両方の遺伝子特異的プライマー含有)に添加しました(A~C)。すべてのqPCR反応は94℃で3分インキュベートすることにより変性させ、その後、94℃で15秒、および60℃で1分、40サイクル実施しました。
また、反応あたりのRT酵素量はRT-qPCRの結果に影響を及ぼすことができます。図8.6に示す通り、2ユニットのMMLV-RTを用いた1ステップ反応(図8.6C)は、20ユニットを用いた反応と比べて特異的です(図8.6B)。RTにオリゴ-dT(O4387)またはランダムプライマーを用いた2ステップRT-PCRは1ステップRT-PCRと比べて高い特異性をもたらしました(図8.6D)。これは、低温RT反応中に遺伝子特異的プライマーが存在しないため、非特異的産物の形成が防止されるという事実に起因する可能性があります。RTの濃度が高いほど、2ステップ反応で優れた結果が得られる可能性がありますが、RT酵素がTaq DNA ポリメラーゼ活性を妨げる可能性があるため7、qPCRに転送されるRT産物量は、最終反応容量の10%を超えないよう制限します。この推奨の例外はReadyScript使用時であり、PCR容量の25%がPCR効率に影響しないRT反応である可能性があります。
RTプロセスに固有である変数についてのこの説明は、RTからの遺伝子
発現の測定値が、テンプレートの量に加え、使用したRT法およびサンプルの量・質に依存していることを示唆しており、MIQEガイドライン8(定量的PCR参照)に記載のとおり、慎重に報告する必要があります。
参考文献
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?