Endonukleazy restrykcyjne - nożyczki molekularne
Tło
Termin "enzym restrykcyjny" wywodzi się z badań faga λ Enterobacteria (faga lambda) w laboratoriach Wernera Arbera i Matthew Meselsona. Zbadano zdolność niektórych szczepów E. coli do hamowania aktywności faga lambda poprzez enzymatyczne rozszczepienie DNA faga, a enzym odpowiedzialny za to ograniczenie wzrostu nazwano enzymem restrykcyjnym.1, 2, 3
Werner Arber, Daniel Nathans i Hamilton O. Smith otrzymali Nagrodę Nobla w dziedzinie fizjologii lub medycyny w 1978 roku za odkrycie i scharakteryzowanie enzymów restrykcyjnych, co doprowadziło do rozwoju technologii rekombinacji DNA.
Wprowadzenie
Enzymy restrykcyjne są również nazywane "nożyczkami molekularnymi", ponieważ rozcinają DNA w lub w pobliżu określonych sekwencji rozpoznawczych znanych jako miejsca restrykcyjne. Enzymy te wykonują jedno nacięcie na każdej z dwóch nici DNA i są również nazywane endonukleazami restrykcyjnymi.4
Wirusy infekują komórki gospodarza poprzez wstrzyknięcie swojego DNA do komórek. To wirusowe DNA przejmuje maszynerię komórki gospodarza do reprodukcji wirusowego potomstwa, powodując śmierć komórki gospodarza. Aby przezwyciężyć infekcję wirusową, wiele bakterii i archeonów wykształciło kilka mechanizmów. Główny mechanizm ochronny polega na wykorzystaniu enzymów restrykcyjnych do degradacji inwazyjnego wirusowego DNA poprzez rozszczepienie go w określonych miejscach restrykcyjnych. W tym samym czasie komórka gospodarza chroni własne DNA przed rozszczepieniem, stosując inne enzymy zwane metylazami, które metylują zasady adeniny lub cytozyny w sekwencjach rozpoznawczych gospodarza. Dla każdego enzymu restrykcyjnego komórka gospodarza wytwarza odpowiadającą mu metylazę, która metyluje i chroni DNA gospodarza przed degradacją. Enzymy te tworzą systemy modyfikacji restrykcyjnej (R-M).
Enzymy restrykcyjne katalizują hydrolizę wiązania między atomem 3'-tlenu a atomem fosforu w szkielecie fosfodiestrowym DNA. Do swojej aktywności enzymy te wymagają Mg2+ lub innych jonów dwuwartościowych.
Nomenklatura
Smith i Nathans zaproponowali wytyczne dotyczące nazewnictwa endonukleaz restrykcyjnych w 1973 roku. Zgodnie z tymi wytycznymi, nazwy enzymów zaczynają się od trzyliterowego akronimu zapisanego kursywą. Pierwsza litera wskazuje pierwszą literę rodzaju bakterii, z którego enzym został wyizolowany, a kolejne dwie litery pochodzą od gatunku bakterii. Po nich mogą następować dodatkowe litery lub cyfry wskazujące serotyp lub szczep. Po tym następuje spacja i cyfra rzymska wskazująca chronologię identyfikacji. Na przykład, Hind III był trzecim z czterech enzymów wyizolowanych z Haemophilus influenza serotypu d.6
Rodzaje enzymów restrykcyjnych
.W oparciu o skład, charakterystykę miejsca rozszczepienia i wymagania dotyczące kofaktorów, endonukleazy restrykcyjne są podzielone na cztery grupy: typ I, II, III i IV.
Miejsce rozszczepienia jest oznaczone czerwoną strzałką w tabeli.
Czynniki wpływające na aktywność enzymów restrykcyjnych
W zależności od DNA substratu i warunków reakcji, enzymy restrykcyjne wykazują dużą zmienność rozszczepiania i możliwą aktywność gwiazdy. W celu uzyskania pożądanego rozszczepienia, ważne staje się kontrolowanie następujących czynników:
- Aktywność gwiaździsta: W nieoptymalnych warunkach reakcji, niektóre enzymy restrykcyjne rozszczepiają sekwencje zasad w miejscach innych niż zdefiniowana sekwencja rozpoznawania. Innymi słowy, rozszczepiają one w niespecyficznych miejscach. Zjawisko to nazywane jest aktywnością gwiazdy. Niektóre z czynników indukujących aktywność gwiezdną to wysokie stężenie soli i glicerolu, obecność zanieczyszczeń, nadmierna ilość enzymu w porównaniu do DNA substratu, wydłużony czas inkubacji lub niekompatybilny bufor i kofaktor.
- Metylowane DNA: Kilka cząsteczek DNA jest metylowanych w miejscu rozpoznawania, co czyni je odpornymi na rozszczepienie przez niektóre enzymy restrykcyjne. Na przykład większość szczepów E. coli wykazuje metylotransferazy Dam lub Dcm, które metylują określone miejsca rozpoznawania, tworząc odpowiednio G6mATC i C5mCA/TGG. G6mATC jest odporna na rozszczepienie przez Mbo I.
- Temperatura: Większość endonukleaz optymalnie trawi docelowe DNA w temperaturze 37 °C. Istnieją jednak pewne wyjątki z niższymi lub wyższymi optymalnymi temperaturami. Na przykład, Taq I optymalnie trawi w temperaturze 65 °C, a Apa I (nr katalogowy 10899208001) trawi w temperaturze 25 °C.
Isoschizomery i Neoschizomery6
Isoschizomery to enzymy restrykcyjne o tej samej sekwencji rozpoznawania i miejscach rozszczepienia. Przykład: Sph I (CGTAC/G) i Bbu I (CGTAC/G)
.
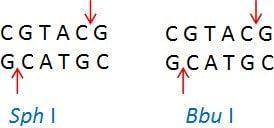
Neoschizomery to enzymy restrykcyjne o tej samej sekwencji rozpoznawania, ale rozszczepiające DNA w innym miejscu w tej sekwencji. Przykład: Tai I (ACGT/) i Mae II (A/CGT)
.

Produkty trawienia restrykcyjnego
Trawienie restrykcyjne dwuniciowego DNA wytwarza dwa rodzaje końców: lepkie i tępe.
Tępe końce posiadają grupę 5'-fosforanową, która sprzyja ligacji. Są one uniwersalnie kompatybilne z innymi tępymi końcami DNA.
Tępe końce generowane przez EcoR V

Kleiste końce to małe odcinki jednoniciowego DNA zdolne do samoligacji lub ligacji z komplementarnym regionem innej cząsteczki DNA. Lepkie końce posiadają 3'- lub 5'-overhangs o długości 1-4 nukleotydów.
5' kohezyjny koniec generowany przez Bln I (nr kat. 11558170001)

3' Spójny koniec generowany przez Kpn I

System buforów
Nasza kolekcja enzymów restrykcyjnych została zoptymalizowana do trawienia przy użyciu pięciu unikalnych buforów. Podczas trawienia DNA przy użyciu pojedynczego enzymu należy użyć buforu dostarczonego z enzymem. Do podwójnego trawienia DNA należy użyć buforu, w którym oba enzymy wykazują 100% aktywności. Alternatywnie, optymalny bufor można określić na podstawie wykresu typowych podwójnych trawień. W niektórych przypadkach zalecane jest trawienie sekwencyjne ze względu na niekompatybilność buforu (skład lub temperatura). Protokoły trawienia restrykcyjnego pojedynczym enzymem, dwoma enzymami restrykcyjnymi i sekwencyjnego trawienia DNA można znaleźć na stronie Protokół trawienia enzymami restrykcyjnymi. Wybór odpowiedniego buforu ma kluczowe znaczenie dla uzyskania wysokiej aktywności obu enzymów i uniknięcia aktywności gwiezdnej. Wybór pięciu buforów pozwala użytkownikowi wybrać kompatybilne bufory do pożądanego trawienia. Większość trawień wymaga 1-2 buforów, co staje się opłacalne.
Zastosowania
Zdolność endonukleaz restrykcyjnych do rozszczepiania DNA w określonych miejscach rozpoznawania umożliwiła szerokie zastosowanie tych enzymów jako podstawowych narzędzi w kilku technikach biologii molekularnej. Niektóre z głównych zastosowań wyjaśniono poniżej:
- Klonowanie molekularne: Popularnym zastosowaniem enzymów restrykcyjnych było generowanie rekombinowanych cząsteczek DNA. Proces ten obejmuje cięcie DNA dawcy (zwykle plazmidu) i DNA wektora (zwykle genu z innego organizmu) przez enzym restrykcyjny w celu uzyskania zgodnych końców. Końce te mogą być "tępe" lub "lepkie". Dwa rozszczepione DNA są łączone ze sobą za pomocą enzymu zwanego ligazą DNA w celu wygenerowania rekombinowanej cząsteczki DNA. Ten rekombinowany DNA może być następnie wprowadzony do organizmu gospodarza w celu replikacji. Więcej szczegółów można znaleźć w Restriction Enzyme Cloning Manual
- Mapowanie DNA, zwane również mapowaniem restrykcyjnym, obejmuje wykorzystanie endonukleaz restrykcyjnych w celu uzyskania informacji strukturalnych fragmentu DNA lub genomu. Mapowanie obejmuje określenie kolejności miejsc enzymów restrykcyjnych w genomie. DNA będący przedmiotem zainteresowania, którego struktura ma zostać określona, jest rozszczepiany za pomocą szeregu endonukleaz restrykcyjnych w celu wytworzenia fragmentów DNA o różnej wielkości. Fragmenty te są rozdzielane na żelu agarozowym w celu określenia struktury interesującego DNA.
W oparciu o znane miejsca enzymów restrykcyjnych określonego fragmentu DNA, endonukleazy restrykcyjne mogą być wykorzystane do weryfikacji tożsamości tego fragmentu DNA. - Skanowanie genomu za pomocą punktów restrykcyjnych to metoda analizy genomu, która wykorzystuje kombinację enzymów restrykcyjnych do wizualizacji różnic w poziomach metylacji w genomie danego organizmu. Jest to przydatna technika do identyfikacji odchyleń od normy w dowolnym DNA. Jest bardzo skuteczna w wykrywaniu hiper/hipometylacji w nowotworach, delecji lub amplifikacji genów lub zmian w ekspresji genów podczas rozwoju organizmu.8
- Sekwencjonowanie genów: Duża cząsteczka DNA może być sekwencjonowana poprzez trawienie jej enzymami restrykcyjnymi i przetwarzanie powstałych fragmentów przez sekwenator DNA.
- Polimorfizm długości fragmentów restrykcyjnych (RFLP) obejmuje trawienie próbki DNA za pomocą enzymów restrykcyjnych, oddzielanie tych fragmentów na podstawie długości za pomocą elektroforezy żelowej i przenoszenie ich na membranę. Fragmenty te są następnie wiązane z radioaktywną lub fluorescencyjną znakowaną sondą ukierunkowaną na określone sekwencje, które są umieszczone w miejscach enzymów restrykcyjnych. RFLP występuje, gdy wynikowe długości fragmentów różnią się między osobnikami. Każda osoba ma unikalny wzór zwany "biologicznym kodem kreskowym". Technika ta była pierwszą techniką profilowania DNA stosowaną w mapowaniu genów, lokalizacji genów zaburzeń genetycznych, określaniu ryzyka choroby i testowaniu ojcostwa.9
- Elektroforeza żelowa w polu impulsowym obejmuje rozdzielanie dużych fragmentów DNA, głównie fragmentów powstałych w wyniku trawienia genomu bakterii rzadkim enzymem restrykcyjnym. Unikalny wzór jest wykorzystywany do rozróżniania różnych szczepów bakterii. Może to być przydatne do identyfikacji konkretnego szczepu jako przyczyny powszechnej choroby.10, 11
- Seryjna analiza ekspresji genów (SAGE) to technika, która obejmuje ilościową i jednoczesną analizę dużej liczby transkryptów w postaci małych znaczników. Enzymy restrykcyjne są wykorzystywane w tej technice jako enzymy kotwiczące i znakujące.12
- Integracja za pomocą enzymów restrykcyjnych (REMI) obejmuje wykorzystanie enzymów restrykcyjnych do wytworzenia kompatybilnych spójnych końców w genomie w celu wprowadzenia mieszaniny plazmidowego DNA, który został zlinearyzowany enzymem restrykcyjnym. Plazmidowe DNA jest transformowane do komórki gospodarza wraz z enzymem restrykcyjnym, który ułatwia integrację DNA z miejscami restrykcyjnymi w chromosomach. Technika ta jest przydatna do badań genetycznych oraz do wprowadzania markerów genetycznych i molekularnych w określonych punktach genomu w celu identyfikacji interesujących genów na podstawie ich zmutowanych fenotypów.13
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?