Walidacja edycji genów za pomocą CRISPR/Cas9
Test T7E1 a metody sekwencjonowania i kiedy ich używać
Przeprowadzenie eksperymentu CRISPR obejmuje wiele etapów, a każdy z nich ma opcje do rozważenia. Po wybraniu genu docelowego, zaprojektowaniu odpowiedniego RNA prowadzącego (gRNA) i zamówienie prowadnic i niezbędnych odczynników, kolejnym etapem jest sprawdzenie, czy prowadnice skutecznie wycelowały w interesujący gen i wprowadziły pożądaną zmianę w ekspresji (Rysunek 1).

Rysunek 1A.Zaprojektuj gRNA, wybierz nukleazę Cas i wybierz metodę transfekcji.
Rysunek 1B.Wprowadzenie komponentów CRISPR przy użyciu wybranej metody transfekcji w celu edycji docelowych genów w pożądanej linii komórkowej.

Rysunek 1C.Analiza wyników edycji genów w celu potwierdzenia dokładności i wydajności za pomocą T7E1, sekwencjonowania Sangera, NGS, Western/Elisa, FACS.
Przedstawiamy tutaj przegląd dostępnych metod walidacji, w tym zarówno ich mechanizmów, jak i względnych zalet, aby pomóc w wyborze przepływu pracy.
Znaczenie kontroli w walidacji CRISPR
Ponieważ walidacja jest tak ważną częścią eksperymentu edycji genów, posiadanie właściwa kontrola jest niezbędna, zapewniając podstawę do rzetelnej analizy. Typowa kontrola negatywna składa się z gRNA, które nie jest ukierunkowane na żadną znaną sekwencję w systemie eksperymentalnym i jest wprowadzane do komórek przy użyciu tych samych odczynników, co gRNA ukierunkowane na interesujący gen. Pozwala to na przypisanie każdego zaobserwowanego fenotypu specyficznym skutkom utraty funkcji genu, a nie jakimkolwiek efektom poza celem odczynników użytych do wygenerowania nokautu.
Składające się z co najmniej jednego, wstępnie zwalidowanego, wysokowydajnego gRNA w identycznych warunkach eksperymentalnych jak walidowane gRNA, kontrole pozytywne są również integralną częścią każdego eksperymentu edycji genów. Często w tym celu wykorzystywane są geny gospodarza. Kontrole pozytywne są szczególnie ważne, gdy obserwuje się brak cięcia, aby upewnić się, że nie jest to wynikiem błędu w procedurze eksperymentalnej.
Ocena celowania CRISPR
Istnieją dwie kategorie powszechnie stosowanych metod wykrywania, czy dany gen został pomyślnie edytowany w eksperymencie CRISPR. Pierwszy typ wykorzystuje zdolność enzymów do namierzania i rozszczepiania niedopasowanego DNA w celu sprawdzenia obecności zmutowanych alleli. Drugi typ opiera się na sekwencjonowaniu, umożliwiając bezpośrednią ocenę samego DNA. Oba rodzaje walidacji mogą być przydatne w różnych częściach procesu CRISPR.
Test rozszczepiania niedopasowanych enzymów do walidacji pierwszego etapu CRISPR: Test T7E1
Techniki rozszczepiania niedopasowania enzymatycznego (EMC) są generalnie niedrogie i łatwe do przeprowadzenia przy użyciu standardowego sprzętu laboratoryjnego, dlatego są często wykorzystywane do badania komórek pod kątem udanej edycji genów. Endonukleaza T7 I (T7E1)1 jest pochodzącym z bakteriofaga enzymem specyficznym dla niedopasowania, powszechnie stosowanym do walidacji CRISPR i można go znaleźć w zestawie T7E1 firmy Sigma-Aldrich®.
Pierwszym krokiem walidacji za pośrednictwem T7E1 jest pobranie genomowego DNA i amplifikacja regionu otaczającego miejsce docelowe gRNA metodą PCR (Tabela 1). Jeśli mutacja została pomyślnie wprowadzona do jednego allelu przez niehomologiczne łączenie końców (NHEJ) po rozszczepieniu za pośrednictwem CRISPR/Cas9, skutkuje to amplifikacją zarówno sekwencji typu dzikiego, jak i zmutowanych. Polimeraza DNA o wysokiej wierności, taka jak AccuTaq™ enzym, ma kluczowe znaczenie na tym etapie, aby zapobiec fałszywie dodatnim wynikom w wyniku mutacji wprowadzonych na etapie amplifikacji, a nie przez edycję genów za pośrednictwem CRISPR.
Po amplifikacji, powstałe DNA jest podgrzewane i schładzane w celu denaturacji i reanalizowania, generując niektóre produkty, które są heterodupleksami amplikonów typu dzikiego i zmutowanego, niektóre całkowicie typu dzikiego, a niektóre całkowicie zmutowane. Wszystkie z nich są następnie inkubowane z enzymem T7E1, a produkty reakcji są nanoszone na żel agarozowy w celu analizy (Rysunek 2). Niedopasowane sekwencje heterodupleksów zmieniają strukturę DNA, czyniąc je celem rozszczepienia przez T7E1 i generując specyficzny i przewidywalny rozmiar produktów rozszczepienia w oparciu o startery PCR i miejsca wiązania gRNA. Stosunek intensywności wyciętych i niewyciętych pasm na żelu agarozowym może być wykorzystany jako odczyt wydajności edycji genów.
Przegląd procesu walidacji
Walidacja eksperymentu edycji genów przy użyciu T7E1

Krok 1
Izolacja genomowego DNA - zmodyfikowany Iocus

Krok 2
Aplifikacja PCR

Krok 3
Denaturacja i wyżarzanie - możliwe produkty ponownego wyżarzania

Krok 4
Trawienie niedopasowanych dupleksów - endonukleaza T7 I

Krok 5
Elektroforeza w żelu agarozowym
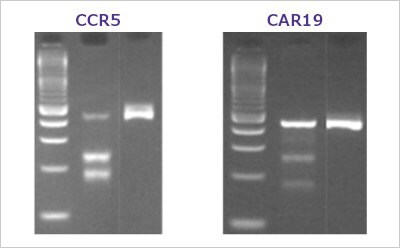
Rysunek 2. Typowy wynik testu T7E1Komórki HEK293 transfekowano różnymi kompleksami Cas9 i sgRNA RNP ukierunkowanymi na geny CCR5 i CAR19. Po trzech dniach transfekowane komórki zbierano i pobierano genomowe DNA. Geny docelowe amplifikowano metodą PCR przy użyciu polimerazy DNA AccuTaq™ LA (D8045), a edycję analizowano przy użyciu zestawu T7 Endonuclease I Gene Editing Detection Kit. Pasy są uporządkowane według drabiny, komórki edytowane sgRNA, kontrola negatywna.
Prostota przepływu pracy EMC sprawiła, że jest to popularna metoda walidacji CRISPR. Jest łatwa w analizie, zapewniając wyniki tego samego dnia bez konieczności sekwencjonowania lub skomplikowanego oprogramowania. Metoda ta nie jest jednak w stanie określić konkretnych mutacji i może generować fałszywie pozytywne wyniki w wyniku naturalnie występującego polimorfizmu.
Metody oparte na sekwencjonowaniu do walidacji CRISPR
Podczas gdy metody EMC mogą przeprowadzić skuteczną analizę pierwszego przejścia edycji genów w przepływie pracy CRISPR, wyniki te często muszą zostać potwierdzone przez sekwencjonowanie, aby upewnić się, że wprowadzono pożądaną mutację. Jest to niezbędny krok, ponieważ gRNA może wykazywać silne cięcie w teście EMC, ale może nie wprowadzać mutacji przydatnej do nokautu białka, ponieważ na przykład jest w ramce i wytwarza białko, które może zachować swoją funkcję. Chociaż może się wydawać, że użycie sekwencjonowania jako wstępnego narzędzia przesiewowego byłoby korzystne, ponieważ jest bardziej szczegółowe niż analiza EMC, wyższy koszt i dłuższy czas realizacji uniemożliwiają wykorzystanie sekwencjonowania w ten sposób.
Sekwencjonowanie Sangera i śledzenie indeli przez dekompozycję (TIDE)
Sekwencjonowanie Sangera jest tradycyjną i popularną techniką walidacji edycji genów.3 W tej metodzie DNA jest amplifikowane przy użyciu wyspecjalizowanego typu PCR zwanego PCR z terminacją łańcucha, w którym niskie stężenie fluorescencyjnie znakowanych, zmodyfikowanych nukleotydów, które kończą wydłużanie DNA - dideoksyrybonukleotydów (ddNTP) - jest mieszane z dNTP w celu wygenerowania łańcuchów DNA o każdej możliwej długości. Użycie innego fluoroforu dla każdej z zasad umożliwia określenie sekwencji po rozdzieleniu mieszaniny łańcuchów DNA według wielkości za pomocą elektroforezy w żelu kapilarnym.
Sekwencjonowanie Sangera jest bardzo niezawodne, ale ma wady. Po pierwsze, jest czasochłonne ze względu na wiele etapów eksperymentalnych. Nie nadaje się również do analizy wysokowydajnej, ponieważ sekwencjonuje tylko pojedynczy fragment DNA na raz. Ogólnie rzecz biorąc, nie stanowi to problemu w przypadku walidacji udanej edycji pojedynczego genu, ale nie pozwala na określenie efektów poza celem w tej samej reakcji.
TIDE to metoda analizy oparta na sekwencjonowaniu Sangera, w której mieszana populacja edytowanych i nieedytowanych komórek jest porównywana przez algorytm oprogramowania w celu ujawnienia obecności wcięć i ich częstotliwości.4 Jest to szybki, czuły i opłacalny sposób na znalezienie mutacji, który nie wymaga klonalnej populacji komórek do przeprowadzenia, ale charakter algorytmu uniemożliwia identyfikację sekwencji bez bardziej szczegółowych metod.
Sekwencjonowanie następnej generacji (NGS)
NGS to wysoce czuła metoda zdolna do identyfikacji mutacji o niskiej częstotliwości w populacji komórek.3 Podobnie jak sekwencjonowanie Sangera, analizuje fragmenty DNA, ale różni się tym, że robi to za pomocą masowo równoległej technologii sekwencjonowania dla znacznie większej przepustowości. Ponieważ może analizować tak wiele fragmentów równolegle, może być wykorzystywana do oceny efektów poza celem w eksperymentach CRISPR. Chociaż ma ona istotne zalety w porównaniu z innymi metodami walidacji, jest również podatna na błędy i jest najbardziej kosztowną z omawianych tutaj technik.
Potwierdzenie utraty ekspresji
Oprócz walidacji udanej edycji genów poprzez potwierdzenie sekwencji DNA, krytyczne jest również potwierdzenie, że pożądana modyfikacja wystąpiła w produkcie białkowym. Podczas oceny nokautu, wprowadzenie insercji i delecji niekoniecznie prowadzi do całkowitego zakłócenia ekspresji: niektóre geny mogą nadal wytwarzać skrócone lub częściowe białka. Potwierdzenie można przeprowadzić na poziomie mRNA przy użyciu technik takich jak RT-PCR w celu oceny dokładnej integracji mutacji lub SNP, podczas gdy potwierdzenie wyeliminowania genu nie jest możliwe.knockout i dodanie elementu fuzyjnego takiego jak GFP należy potwierdzić za pomocą elektroforezy żelowej i Western blot przy użyciu jednego lub więcej zatwierdzonych przeciwciał.
Assessing CRISPR/Cas9 Gene Editing Isn't One-Size-Fits-All
Przeprowadzenie edycji genów za pomocą CRISPR/Cas9 jest złożone i powinno być dostosowane do celów badania.W rezultacie silne kontrole i odpowiednia walidacja zarówno edycji genów, jak i utraty ekspresji mają kluczowe znaczenie dla sukcesu. Liczba próbek, koszt, czas realizacji i rodzaj wyników to czynniki, które będą miały wpływ na wybór metody walidacji (Tabela 2).
Określenie odpowiedniej równowagi wszystkich tych czynników może być wyzwaniem. Jeśli chcesz uzyskać więcej informacji lub omówić opcje, które odpowiadają Twoim potrzebom, porozmawiaj z jednym z naszych ekspertów. Nasz zespół wesprze Cię na każdym etapie eksperymentów związanych z edycją genów.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?