Lipopolisacharydy
Glycobiology Analysis Manual, 2nd Edition
Lipopolisacharyd (LPS) jest głównym składnikiem zewnętrznej błony bakterii Gram-ujemnych. Lipopolisacharyd jest zlokalizowany w zewnętrznej warstwie błony i jest, w szczepach niekapsułkowanych, eksponowany na powierzchni komórki.
Struktura
Nieaktywne lipopolisacharydy bakteryjne są makrocząsteczkami o masie cząsteczkowej 10-20 kDa składającymi się z trzech składników strukturalnych
(Rysunek 1):
- hydrofobowej części lipidowej, lipidu A, która odpowiada za toksyczne właściwości cząsteczki,
- hydrofilowy rdzeniowy łańcuch polisacharydowy oraz
- powtarzający się hydrofilowy O-antygenowy oligosacharydowy łańcuch boczny, który jest specyficzny dla serotypu bakterii.1
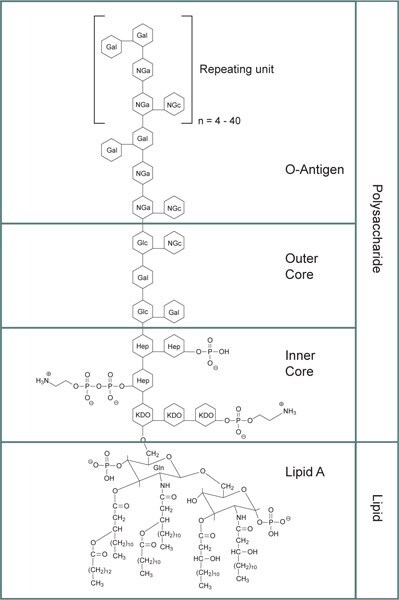
Rysunek 1.Ogólna struktura lipopolisacharydów bakteryjnych. Szczegółowe wyjaśnienia dotyczące każdej sekcji znajdują się w tekście. Skróty: KDO: Kwas 3-deoksy-α-D-mannooktulozonowy; Hep: Heptuloza (ketoheptoza); NGa: Galaktozamina; NGc: Glukozamina.
Rdzeń lipidowy A składa się z zasady β-glukozamino-(1→6)-glukozamino-1-fosforanowej z estrami kwasów tłuszczowych przyłączonymi do obu węglowodanów. Długość łańcucha acylowego i liczba grup acylowych mogą się różnić między gatunkami bakterii, ale są względnie zachowane w obrębie gatunku. Wewnętrzny rdzeń polisacharydowy zawiera zazwyczaj od 1 do 4 cząsteczek KDO (kwas 3-deoksy-α-D-manno-oktulozonowy) przyłączonych do rdzenia disacharydowego. KDO jest specyficznie związany z lipopolisacharydem, a uważano, że biologicznie aktywny lipid A wymaga co najmniej jednej reszty KDO do przeżycia bakterii.2 Jednak szczep supresorowy Escherichia coli K-12 z niedoborem KDO pokazuje, że wymóg KDO nie jest bezwzględny dla żywotności.3
Wewnętrzny rdzeń zawierający KDO jest również modyfikowany monosacharydami heptulozy (ketoheptozy), z których najczęstszym jest L-glicero-α-D-manno-heptopiranoza. Reszty glikanu rdzenia wewnętrznego są zazwyczaj fosforylowane lub modyfikowane grupami zawierającymi fosforany, np. pirofosforanem lub 2-aminoetylofosforanem. Grupy fosforanowe lipopolisacharydów zwiększają ogólny ładunek ujemny błony komórkowej i pomagają stabilizować strukturę.
Zewnętrzny rdzeń lipopolisacharydu zawiera bardziej powszechne heksozy, w tym glukozę, galaktozę i N-acetyloglukozaminę i jest strukturalnie bardziej zróżnicowany niż rdzeń wewnętrzny.
O-antygen jest powtarzającą się jednostką oligosacharydową, zwykle składającą się z dwóch do sześciu cukrów. O-antygen jest podstawowym składnikiem strukturalnym lipopolisacharydu, który różnicuje bakterie. Charakterystyczne struktury antygenu O zostały wykorzystane do identyfikacji i przypisania grup serologicznych do Escherichia coli, Salmonella enterica i Vibrio cholerae.4 Lipopolisacharydy z szorstkich zmutowanych szczepów E. coli brakuje części struktury antygenu O.
Sekcja rdzenia i sekcja lipidowa A lipopolisacharydu mogą mieć pewną zmienność struktury, podczas gdy antygen O ma wysoki stopień zmienności strukturalnej, a także zmienność liczby powtarzających się jednostek. Różnice te powodują znaczną niejednorodność preparatów LPS. Ponieważ LPS jest heterogeniczny i ma tendencję do tworzenia agregatów o różnej wielkości, zgłaszany jest zakres "masy cząsteczkowej" dla tych agregatów wynoszący 1-4 milionów Daltonów lub więcej. Gdy LPS jest traktowany dodecylosiarczanem sodu (SDS) i ciepłem, masa cząsteczkowa wynosi ~50-100 kDa.5
Funkcje i zastosowania
W bakteriach Gram-ujemnych lipopolisacharydy błonowe chronią bakterię przed działaniem soli żółciowych i lipofilowych antybiotyków.6
Lipopolisacharydy są endotoksynami stabilnymi termicznie i od dawna są uznawane za kluczowy czynnik wstrząsu septycznego (posocznicy) u ludzi1,7 oraz, bardziej ogólnie, w indukowaniu silnej odpowiedzi immunologicznej w normalnych komórkach ssaków. Cząsteczka lipidowa A została zidentyfikowana jako krytyczna dla aktywności endotoksyny lipopolisacharydu. Zostało to wykazane przez Galanos i in, stwierdzając identyczne wyniki bioaktywne, w tym aktywność endotoksyczną, między syntetycznymi i pochodzącymi z naturalnych źródeł E. coli preparatami lipidu A.8 Aktywny receptor dla lipopolisacharydu został zidentyfikowany jako kompleks receptora CD14/TLR4/MD2, który promuje wydzielanie cytokin prozapalnych, w tym czynnika martwicy nowotworów-α i interleukiny-1.9 Podczas gdy składnik lipidowy A jest głównie odpowiedzialny za aktywację odpowiedzi immunologicznej, składnik polisacharydowy Salmonella enterica LPS jest również niezbędny do aktywacji NF-κB.10
Preparaty lipopolisacharydowe zostały wykorzystane w badaniach w celu wyjaśnienia struktury LPS,11 metabolizmu,12 immunologii,13 fizjologii,14 toksyczności,15 i biosyntezy.16 Zostały one również wykorzystane do indukowania syntezy i wydzielania czynników promujących wzrost, takich jak interleukiny.17,18 Ze względu na jego związek z posocznicą, lipopolisacharyd został zbadany w celu zidentyfikowania możliwych celów dla przeciwciał i inhibitorów biosyntezy LPS.19,20
Ekstrakcja i oczyszczanie
Lipopolisacharydy można przygotować poprzez ekstrakcję z TCA,21 fenolu,22,23 lub eteru fenolowo-chloroformowo-olejowego (dla szczepów szorstkich).24 Lipopolisacharydy ekstrahowane TCA są strukturalnie podobne do tych ekstrahowanych fenolem, z podobnymi wzorami elektroforetycznymi i endotoksycznością. Podstawowe różnice dotyczą ilości kwasów nukleinowych i zanieczyszczeń białkowych pozostałych po ekstrakcji. Ekstrakty TCA zawierają ~2% RNA i ~10% zdenaturowanych białek, podczas gdy ekstrakty fenolowe zawierają do 60% RNA i <1% białka. Późniejsze oczyszczanie za pomocą chromatografii z filtracją żelową usuwa znaczną część białka obecnego w LPS ekstrahowanym fenolem, ale daje preparat zawierający 10-20% kwasów nukleinowych. Dalsze oczyszczanie przy użyciu chromatografii jonowymiennej daje produkt lipopolisacharydowy, który zawiera <1% białka i <1% RNA.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?