Test immunoprecypitacji RNA (RIP)
Interakcje białko-RNA odgrywają ważną rolę w komórce, w tym funkcje strukturalne, katalityczne i regulacyjne. Podobnie jak immunoprecypitacja chromatyny (ChIP), immunoprecypitacja RNA (RIP) może być stosowana do wykrywania asocjacji poszczególnych białek z określonymi kwasami nukleinowymi, takimi jak mRNA, niekodujące RNA (np. długie niekodujące RNA, wzmacniające RNA, miRNA) i wirusowe RNA. Istnieją dwa główne warianty testu RIP: natywny i usieciowany. Natywny test RIP pozwala na bezpośrednią identyfikację związanych RNA z interesującym białkiem i ich obfitość w immunoprecypitowanej próbce. Natomiast sieciowane testy RIP pozwalają na precyzyjne mapowanie bezpośredniego i pośredniego miejsca wiązania białka i RNA (Rysunek 1). W przypadku usieciowanego RIP, żywe komórki są poddawane działaniu wysoce reaktywnego formaldehydu w celu wytworzenia odwracalnych wiązań krzyżowych białko-RNA między cząsteczkami, które znajdują się w bliskiej odległości (Rysunek 2).
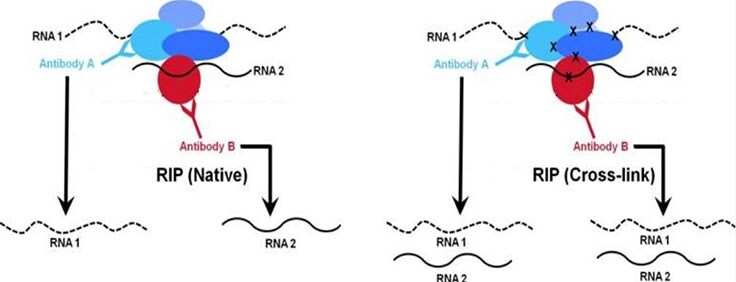
Rysunek 1. Porównanie natywnego i usieciowanegoPodczas gdy zarówno natywne, jak i usieciowane metody są stosowane do analizy RNA związanego z chromatyną, natywne podejście RIP (prawy panel) zazwyczaj pozwala na odzyskanie interakcji białko-RNA o wysokim powinowactwie. I odwrotnie, metoda sieciowania (prawy panel) jest przeznaczona do wychwytywania kompleksów o wyższej masie cząsteczkowej i łatwiejszego wychwytywania słabiej oddziałujących RNA.
Przepływ pracy protokołu testu immunoprecypitacji (RIP)
Protokół wybrany do testu RIP będzie częściowo określony przez miejsce wiązania białka z DNA (cytoplazma lub jądro). Ogólny protokół RIP dla lizatu całej komórki może być najbardziej odpowiedni dla interakcji cytoplazmatycznych, podczas gdy protokół RIP dla jądra może być optymalny dla interakcji zachodzących w jądrze. Jeśli nie ma istniejącej wiedzy na temat lokalizacji wiążącego RNA, ogólny protokół natywnego RIP lizatu całych komórek (Rysunek 2A) (17-700, 17-701) jest zalecany w celu zwiększenia możliwości immunoprecypitacji wiążących RNA niezależnie od ich lokalizacji. Protokół ten działa z różnymi ilościami i rodzajami materiału wyjściowego, takimi jak komórki i próbki tkanek. Jeśli wiadomo, że wiążące RNA występuje w jądrze lub jest związane z chromatyną, natywny jądrowy (17-10522, 17-10523) lub usieciowany protokół RIP (17-10520, 17-10521). Zamiast immunoprecypitacji RNA z lizatu całej komórki, protokoły te przeprowadzają immunoprecypitację na wyizolowanych jądrach, umożliwiając w ten sposób poprawę stosunku sygnału do szumu.
Podczas gdy protokoły te są przeznaczone do immunoprecypitacji wszystkich typów RNA, istnieją dwa dodatkowe protokoły opracowane specjalnie do identyfikacji i analizy regionów chromatyny, które oddziałują z określonymi związanymi z chromatyną długimi niekodującymi RNA / Izolacja chromatyny przez oczyszczanie RNA (ChIRP) (Rysunek 2B) (17-10494, 17-10495) lub profilowanie miejsc metylacji m6A (N6-metyloadenozyna) RNA w całym transkryptomie (Rysunek 2C) (17-10499).

Rysunek 2. Przepływy pracy różnych protokołów RIPA. Schemat postępowania RIP z natywnym lizatem całych komórek (nr produktu 17-700, 17-701) B. Schemat postępowania ChIRP (nr produktu 17-10494, 17-10495) C. Schemat postępowania m6A RIP (nr produktu 17-10499)
RNA wyizolowane po zakończeniu protokołu RIP można analizować kilkoma metodami molekularnymi, w tym końcowym RT-PCR i ilościowym RT-PCR (jeśli znane są cele wiązania RBP), mikromacierzami lub metodami głębokiego sekwencjonowania (Rysunek 3). Biorąc pod uwagę cele RNA o znanej sekwencji, można zaprojektować startery specyficzne dla genów, które umożliwiają walidację (i kwantyfikację) immunoprecypitowanego RNA. Po potwierdzeniu udanego RIP, dalsze badanie populacji RNA w immunoprecypitacji może być prowadzone metodami opartymi na populacji, takimi jak porównawcza hybrydyzacja mikromacierzy powstałych cDNA lub głębokie sekwencjonowanie molekularnie dostosowanych produktów z RIP. Poniżej przedstawiono przykładowe metody wykonywania pomiarów ilościowych w punkcie końcowym lub w czasie rzeczywistym lub analizy NGS eksperymentów RIP.

Rysunek 3.Analiza RIP RNA metodą qRT-PCR
A. Lizat RIP przygotowany z komórek HeLa (2x107 równoważników komórek na IP) poddano immunoprecypitacji przy użyciu 5 µg normalnej mysiej IgG lub przeciwciała anty-PABPC1 i zestawu Magna RIP Kit (nr produktu 17-700). Pomyślna immunoprecypitacja RNA związanego z PABPC1 została zweryfikowana przez qRT-PCR przy użyciu RIP Primers, ACTB.
B.RIP Lizat przygotowany z komórek HeLa (0,5 × 106 lub 1.7 × 106 równoważników komórek na IP) poddano immunoprecypitacji przy użyciu 2,5 μg normalnego króliczego przeciwciała IgG lub przeciwciała anty-HUR i zestawu do immunoprecypitacji Imprint RNA (RIP). Immunoprecypitacja RNA związanego z celem HUR i niezwiązanego z celem została potwierdzona przez qPCR przy użyciu starterów kontrolnych Actin B i JUN.
C. LincSFPQ związany z kompleksem PRC2 (EZH2 i SUZ12) i U1snRNA (negatywny) w komórkach HeLa analizowano za pomocą qRT-PCR z usieciowanym Magna Nuclear RIP (nr produktu 17-10520). Stosunek sygnału do szumu porównano z konkurencyjnym zestawem.
D i E. Magna Nuclear RIP Assay z zaledwie 5 000 (usieciowanych, nr produktu 17-10520) i 100 000 (Native, Product No. 17-10522). Komórki. Dane czułości pokazujące, że wzbogacenie NEAT1 w protokole usieciowania wykazano przy 5000 komórek HeLa, podczas gdy metody natywne wymagały 100 000 komórek (C). Dane dotyczące stosunku sygnału do szumu pokazujące, że ogólny stosunek wyników dodatnich do ujemnych w protokole natywnym był wyższy (D).
F. MeRIP (nr kat.: 17-10499) przeprowadzono przy użyciu całkowitego RNA z komórek HEK293. Oczyszczone RNA analizowano następnie metodą RT-qPCR przy użyciu pozytywnych starterów kontrolnych (MeRIP Primers Human EEF1A1 Positive, część nr CS220017) i negatywnych starterów kontrolnych (MeRIP Primers Human EEF1A1 Negative, nr produktu CS220018).
G. Lewy panel: pomyślne odzyskanie metylowanego RNA za pomocą testu MeRIPTMm6A z mRNA z różnych komórek. MeRIP (nr produktu 17-10499) przeprowadzono przy użyciu mRNA z wolnych od ksenonów ludzkich komórek fibroblastów napletka (HFF, nr produktu SCC058), ludzkich komórek raka płaskonabłonkowego głowy i szyi UM-SCC-104 (nr produktu SCC072) i ludzkich embrionalnych komórek macierzystych (H9). Oczyszczone RNA analizowano następnie metodą RT-qPCR przy użyciu starterów kontroli pozytywnej (MeRIP Primers Human EEF1A1 Positive, Product No. CS220017) i starterów kontroli negatywnej (MeRIP Primers Human EEF1A1 Negative, Product No. CS220018). Panele środkowy i prawy: Analiza poziomów m6A RNA genów reprogramujących w mysich i ludzkich komórkach ES. MeRIP (nr produktu 17-10499) przeprowadzono przy użyciu mRNA z mysich komórek ES PluriStem 129/S6 (nr produktu SCR012) i ludzkich komórek ES H9. Oczyszczony RNA analizowano następnie metodą RT-qPCR przy użyciu starterów MeRIP m6A dla Myc, Sox2, Nanog, Klf4 i Oct4. Sekwencje starterów PCR dla mysich genów reprogramowania zostały dostarczone przez Howarda Changa (Uniwersytet Stanforda). Ludzkie startery PCR zostały zaprojektowane na podstawie opublikowanych danych MeRIP-Seq. Jednoetapowe RT-qPCR przeprowadzono za pomocą urządzenia Bio-Rad qPCR i zestawów iTaq ™ Universal One-Step Kits (Bio-Rad). Procent odzysku wejściowego obliczono na podstawie krzywej standardowej sporządzonej z 0,1% próbki wejściowej lub metod delta delta Ct.
H. ChIRP (nr produktu 17-10494) przeprowadzono przy użyciu lizatu komórek HeLa i zestawu sond Magna ChIRP TERC lncRNA parzyste nieparzyste (nr produktu 03-309) lub NEAT1 lncRNA Probe Set even odd (Product No. 03-308) lub Magna ChIRP Negative Control Probe Set (LacZ) (Product No. 03-307). Oczyszczone RNA analizowano następnie metodą qRT-PCR przy użyciu starterów kontroli pozytywnej RNA (TERC lub NEAT1) i starterów kontroli negatywnej RNA (GAPDH).
RIP RNA analizowane metodą sekwencjonowania nowej generacji (RIP-seq)

Rysunek 4.Analizę RIP-Seq przeprowadzono z EZH2 i SUZ12 w komórkach HeLa przy użyciu zestawu Magna nuclear native RIP (nr produktu 17-10522) lub zestawu Magna nuclear cross-linked RIP (nr produktu 17-10520). Dane wykazały wzbogacone sygnały dla transkryptów NEAT1 (A, górny panel) i HOTAIR lincRNA (B. środkowy i dolny panel).

Rysunek 5.Analiza sekwencjonowania następnej generacji DNA pull-down przy użyciu zestawu Magna ChIRP
ChIRP przeprowadzono na lizacie komórek HeLa. Biotynylowane oligo wychwytujące (Magna ChIRP™ NEAT1 lncRNA Probe Set (odd and even pools) (Product No. 03-308)) i negatywne sondy kontrolne (Magna ChIRP™ Negative Control Probe Set (LacZ) Product No. 03-307) zostały użyte do reakcji pull down. Biblioteki sekwencji zostały zsekwencjonowane na instrumencie HiSeq (Illumina). Odczyty sekwencji zostały dopasowane do genomu referencyjnego (hg19) przy użyciu Bowtie. (a) Piki zostały wywołane oddzielnie przy użyciu danych z parzystych i nieparzystych zestawów sond. Do tego celu można wykorzystać algorytmy takie jak MACS. Te wspólne zostały uznane za prawidłowe piki. (b) Seria etapów przetwarzania i filtrowania po wyrównaniu została przeprowadzona przy użyciu oprogramowania analitycznego dostępnego w laboratorium Howarda Changa (https://changlab.stanford.edu/protocols.html). Dane pokazujące lokalizację RNA NEAT1 do regionu genu NEAT1 i innego przykładowego regionu (c).
RIP FAQs, Troubleshooting Tips and Data Analysis Guide
Podczas przeprowadzania eksperymentów RIP można napotkać pewne wyzwania, takie jak brak immunoprecypitacji RNA, wysokie tło lub niski stosunek sygnału do szumu. Poniższe informacje mają pomóc w uzyskaniu wystarczającej wiedzy na temat tej techniki przed jej rozpoczęciem; zawierają wskazówki, gdy eksperymenty nie działają i prowadzą przez analizę danych.
- Często zadawane pytania dotyczące RIP z lizatu całych komórek
- Wskazówki dotyczące RIP z lizatu całych komórek
- Wskazówki dotyczące izolacji chromatyny przez oczyszczanie RNA (ChIRP)
- Wskazówki dotyczące MeRIP
- Krótki protokół RIP-qPCR
- RIP-qRT-PCR: Data Analysis Calculation Shell
Który zestaw RIP jest odpowiedni dla Ciebie?
Whole cell lysate RIP
Nuklearny RIP (usieciowany i natywny)
Zestawy podstawowe do izolacji chromatyny przez oczyszczanie RNA (ChIRP) i różne zestawy sond lncRNA
meRIP
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?