MBD0012
Wzorzec mikrobiologicznego DNA z Enterococcus faecalis
Suitable for PCR, sequencing and NGS, 10 ng/μL
About This Item
Polecane produkty
Poziom jakości
Formularz
liquid
stężenie
10 ng/μL
metody
DNA extraction: suitable
DNA sequencing: suitable
PCR: suitable
Warunki transportu
ambient
temp. przechowywania
−20°C
Opis ogólny
Przeczytaj tutaj, jak korzystać z naszych standardów, aby zapewnić integralność danych w badaniach nad mikrobiomem.
Zastosowanie
Nadaje się do standardu ilościowego dla PCR, sekwencjonowania i NGS.
Cechy i korzyści
- Indywidualny wzorzec mikrobiologiczny dla mikrobiomu i metagenomiki
- Odpowiedni standard dla PCR, sekwencjonowania i NGS
- Poprawa analiz bioinformatycznych
- Zwiększa odtwarzalność
- Porównywanie wyników między laboratoriami
Postać fizyczna
Kod klasy składowania
12 - Non Combustible Liquids
Klasa zagrożenia wodnego (WGK)
WGK 1
Temperatura zapłonu (°F)
Not applicable
Temperatura zapłonu (°C)
Not applicable
Wybierz jedną z najnowszych wersji:
Certyfikaty analizy (CoA)
Nie widzisz odpowiedniej wersji?
Jeśli potrzebujesz konkretnej wersji, możesz wyszukać konkretny certyfikat według numeru partii lub serii.
Masz już ten produkt?
Dokumenty związane z niedawno zakupionymi produktami zostały zamieszczone w Bibliotece dokumentów.
Produkty
An overview of human microbiome research, workflow challenges, sequencing, library production, data analysis, and available microbiome reagents to support your research.
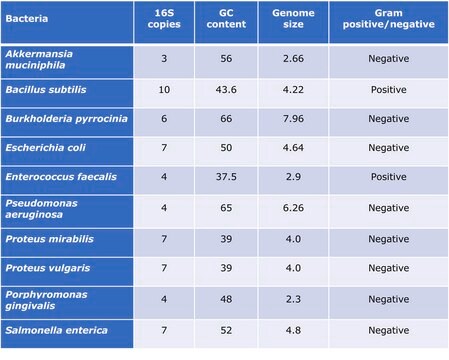
The use of standards is critical to the integrity of metagenomics research. Learn how DNA standards for bacteria, fungi, and viruses are applied to studying the microbiome. Choose standards for E. coli and other key species, as well as mixed community standards.
Nasz zespół naukowców ma doświadczenie we wszystkich obszarach badań, w tym w naukach przyrodniczych, materiałoznawstwie, syntezie chemicznej, chromatografii, analityce i wielu innych dziedzinach.
Skontaktuj się z zespołem ds. pomocy technicznej