

Human Protein Atlasではヒトプロテオームの特性評価を行っています。
Human Protein Atla(HPA)プロジェクトは、2003年に、Knut and Alice Wallenberg Foundationから資金提供を受け、Mathias Uhlén教授を代表とするスウェーデンの研究者によって開始されました1,2。抗体を使用してヒトプロテオームを系統的に探索する世界トップレベルのユニークな取り組みです。
HPAプロジェクトの目的は、 完全なヒトプロテオームの発現マップを提示することです。この目的を達成するため、ヒト遺伝子がコードするすべてのタンパク質に対して特異性の高いポリクローナル抗体が開発され、組織アレイを用いて多数の組織および細胞についてタンパク質プロファイリングが確立されています。使用されているアプリケーションは、免疫組織染色(IHC)、ウェスタンブロッティング(WB)、タンパク質アレイ、および共焦点免疫蛍光顕微鏡法(ICC-IF)です。
2014年11月のHuman Protein Atlas
2014年11月に公開されたHuman Protein Atlasのバージョン13では、組織ベースの完全なヒトプロテオームのマップが示されています。広範なデータはTissue Atlas、Cancer Atlas、Subcell Atlas、Cell Line Atlasの4つの「サブアトラス」に分割されています。Tissue Atlasに示されたすべてのタンパク質の発現プロファイルは、多数のヒト組織について行ったIHC分析に基づいています。現在では、各遺伝子についてRNAシーケンシングデータと相関させたタンパク質発現データも掲載されています。Cancer Atlasでは、多数のがんの差次的発現遺伝子を検討することができ、Subcell Atlasでは、共焦点顕微鏡法により細胞内局在性が示されています。 Cell Line Atlasには、一般的な細胞株でのタンパク質発現に関する追加情報が含められており、それは、研究のための有用な情報源として高く評価されています。
本プロジェクトでは、48の正常なヒト組織、20のがん型、および44のヒト細胞株のサンプルが含まれる組織マイクロアレイが使用されています。48の正常組織データは各3試料で表示されており、82の細胞型が示されています。正常組織の画像データにはすべて、病理検査に基づく発現量のアノテーションが付与されており、そのデータは正常なTissue Atlas上に表示され、mRNAおよびタンパク質レベルでヒト遺伝子の発現プロファイルの情報を得ることができます。 mRNA発現データは27の正常な主要組織型のRNAディープシーケンシグ(RNA-Seq)から得られたものです。
Cancer Atlasには、多数のヒトがん標本のタンパク質発現パターンに基づいた遺伝子発現データが含まれています。もっともよくみられる20のヒトがん型に対応する、全体で216のがんサンプルが、含められた全遺伝子について分析されています。すべてのがん組織画像データは病理学者が手動でアノテーションを付与しており、標準Tissue Atlasに限っても、タンパク質データには、16,621個の遺伝子に対応するタンパク質発現量データが含まれており、それぞれについて適切な抗体が提供されています。
乳房組織サンプルおよび細胞株を用いたバリデーション
標準Tissue Atlasの各抗体について、3個体から得られた正常乳房サンプルのIHC画像データが提供されています。さらに、各抗体について、最高12例の患者から各2試料採取した乳がんサンプルのデータがCancer Atlasに提示されており、大半の抗体について、MCF-7およびSK-BR-3乳房細胞株から得られた画像データもCell Line Atlasに提示されています。
参考文献
Atlas Antibodies社のPrestige Antibodies®(Prestige抗体)
Prestige抗体 – HPAの構成要素
Prestigeポリクローナル抗体が他のタンパク質に対して独自性があり、交差反応性が低いのは、抗原領域の綿密な選択、リコンビナント抗原のアフィニティー精製、複数の方法と厳格な承認プロセスを使用したバリデーションを行っているためです。
製品開発
Prestige抗体は、約50~150個のアミノ酸からなるリコンビナントヒトタンパク質エピトープタグ(Protein Epitope Signature Tag:PrEST)に対して作製します。特異性の高い抗体の産生に適した、天然タンパク質に存在する独特のエピトープを含めるため、独自のソフトウェアを用いてこれらのタンパク質フラグメントを設計しています。他のヒトタンパク質に対する相同性が最低であるPrESTを確実に抗原として使用するため、完全ヒトゲノムスキャニングを行って、このような設計を行います。
承認
Prestige抗体は、IHC、WBまたはICC-IFの試験結果を組み合わせたバリデーション、RNAシーケンシング、ならびにバイオインフォマティクス予測法および文献から得られる情報に基づいて承認しています。文献は決定的な結論がない場合が多いため、HPAプロジェクトの重要な方針として、同じタンパク質ターゲットに対して重複しないエピトープを有するペアの抗体を作製し、1つの抗体の結果とバリデーションをもう1つの抗体のバリデーションに使用できるようにしています。
Prestige抗体のラインナップ
現在、17,000以上のPrestigeポリクローナル抗体がラインナップされており、毎年2,000の新たな抗体が追加されています。
Human Protein Atlasプロジェクトで開発され、特性評価された抗体は、Prestige抗体およびAtlas抗体として提供されています。
モノクローナル抗体の開発
Prestige抗体には、選ばれたマウスモノクローナル抗体も含まれています。 モノクローナル抗体も、毎年新製品が追加され、随時拡大しています。
独自の特徴
独特で重複のないエピトープおよび/またはアイソタイプのみを認識するクローンを提供するために、特別な注意を払っています。モノクローナル抗体は、ポリクローナル抗体と同じ厳格なPrEST製造工程および特性評価手順を経ているため、承認されたアプリケーションで優れた性能を発揮するとともに、明確な特異性を有し、連続性が保証されており、安定供給が可能です。概して、抗体は希釈倍率が低い高濃度でもワークし、標準化された測定法に有用です。
クローンの選択
選択したハイブリドーマのサブクローニングと増殖の前に、多数のELISA陽性の細胞上清について機能特性の評価を行って、各アプリケーションに最適なクローンを選択しています。
エピトープマッピング
オーバーラップする合成ペプチドを用いて、ビーズベースのアレイ形式でエピトープマッピングを行い、重複のないエピトープのみを有するクローンを選択しています。
アイソタイピング
アイソタイプ特異的二次抗体を用いたマルチプレックスを可能にするため、すべてのモノクローナル抗体に対してアイソタイピングを行っています。
ハイブリドーマ細胞の培養
Atlas抗体には、マウスを使用して腹水を産生させる方法に替えて、製造スケールアップ期にはin vitro法を使用します。
抗体の特性評価
Prestigeモノクローナル抗体の特性検討は広範な文献検索から開始し、IHCによる特性評価に最も妥当性があり臨床的に有意な組織を選択します。 IHC適用データには、しばしば各抗体について複数の組織型を表示しています。陽性を示す染色組織に加えて、ネガティブコントロールの染色も表示し、必要に応じて臨床がん組織の染色を表示しています。
ウェスタンブロッティング(WB)による特性評価には、内在性のヒト細胞または組織のタンパク質ライセートの結果、あるいは一部製品ではリコンビナント全長ヒトタンパク質のライセートの結果を含めています。
各モノクローナル抗体は、このように、その特異的ターゲットに関して最も関連性の高い特性評価データを添付して供給します。
Prestigeポリクローナル抗体の製品番号は、「HPA」から始まり、モノクローナル抗体は「AMAB」から始まります。

抗HER2(AMAb90627)を用いた乳がんの免疫組織染色では、HER2陽性乳管がんの腫瘍細胞に(中等度の細胞質陽性とともに)強い膜陽性が認められますが、HER2陰性乳管がんに膜陽性は認められません。HER2はウェスタンブロッティングによって乳がん細胞株K-BR-3で検出されます。
プロゲステロン受容体

正常なヒトの子宮組織を用いた抗PGR抗体(HPA004751)によるIHC染色では、腺細胞に強い核陽性が認められます。 表示されている乳がんサンプルの腫瘍細胞の染色箇所も核です。ICC-IFでは、U-251MG細胞で核染色が認められます。
エストロゲン受容体

抗ESR1抗体(HPA000449)は、IHCにより、ヒト乳房組織の腺細胞および乳がんサンプルの腫瘍細胞で明確な核陽性を示します。

抗ESR1抗体(HPA000450)を用いたIHC染色では、ヒトの子宮組織の腺細胞および間質細胞、ならびに乳がんの腫瘍細胞に強力な核陽性が認められます。
Ki67

抗MKI67抗体(HPA000451)は、IHCにより、ヒトリンパ節の反応中心のわずかな細胞で強力な核陽性を示します。乳がんの腫瘍細胞の染色箇所も核であり、ICC-IFによるヒト細胞株U-2OSの染色では、核での陽性が認められます。

抗MKI67抗体(HPA001164)を用いたヒト扁桃組織のIHC染色では、反応中心細胞に核染色が認められます。乳がんの腫瘍細胞では、染色箇所は主に核であり、U-2OS細胞では、ICC-IFによって強力な核陽性が認められます。
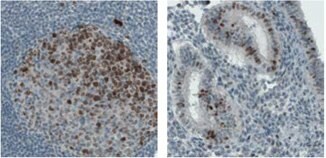
モノクローナル抗MKI67抗体(AMAb90870)を用いたヒト結腸のリンパ節のIHC染色では、反応中心細胞の核と核小体に強力な免疫反応が認められます。子宮では、一部の腺細胞に核陽性が認められます。
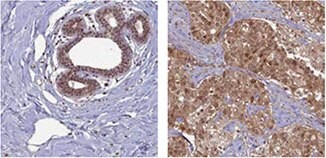
抗BRCA1抗体(HPA034966)はIHCにより、正常ヒト乳房組織の腺細胞および乳がんサンプルの腫瘍細胞で陽性を示します。

抗BRCA2抗体(HPA026815)を用いた正常ヒト乳房組織のIHC染色では、腺細胞で陽性が認められます。乳がんでは、腫瘍細胞の核染色が認められます。
ACAT1

抗ACAT1抗体(HPA004428)を用いたヒト肝組織のIHC染色では肝細胞に強力な細胞質陽性が認められます。ウェスタンブロッティングによって、ACAT1は、ヒト細胞株RT-4およびU251-MGならびに肝および扁桃組織ライセート中で検出されます。ヒト細胞株A-431ではICC-IFによって、ミトコンドリアで陽性が認められます。
CD44

抗CD44抗体(HPA005785)を用いたヒト食道組織のIHC染色では、扁平上皮細胞に強力な細胞質陽性および膜陽性が認められます。ウェスタンブロッティングによって、CD44はヒト細胞株U-251MGで検出されます。ヒト細胞株U-251MGでは、ICC-IFによって細胞膜に陽性が認められます。
* ヒトとげっ歯類の両サンプルのWB
参考文献
MammaPrint、Oncotype、EndoPredictおよびuPA検査における遺伝子産物に対する抗体
ここでは、診断用のMammaPrint、EndoPredict、OncotypeおよびuPA検査に含まれる遺伝子産物に対するPrestige抗体製品カタログの抗体を提示しています。MammaPrintは、Agendiaが販売するアムステルダム70遺伝子の乳がん遺伝子発現シグネチャーに基づいた遺伝子発現プロファイルの検査です。それは、乳がんが身体の他の部位に転移するリスクを評価する検査です。MammaPrintは、患者を「低リスク」および「高リスク」に層別化することが目的です。Oncotype DX(Genomic Healthが開発)は、米国の臨床の現場で最も頻繁に使用されている遺伝子発現プロファイル検査であり、腫瘍内の21の遺伝子のパネルを分析し、再発スコア(Recurrence Score)を判定します。
BIRC5/Survivin

抗CD68抗体(HPA048982)を使用したヒト肺組織のIHC染色では、マクロファージおよび脾臓のような造血組織に強力な細胞質陽性が認められます。
CD68/Macrosialin

抗CD68抗体(HPA048982)を使用したヒト肺組織のIHC染色では、マクロファージおよび脾臓のような造血組織に強力な細胞質陽性が認められます。
DTL

抗DTL抗体(HPA028016)を用いたヒト骨髄のIHC染色では、骨髄造血細胞に強力な核陽性が認められます。ICC-IFにより、U-251 MG細胞では核の染色が認められます。
GSTM5

抗GSTM5抗体(HPA048652)は、IHCによりヒト直腸の腺細胞で細胞質陽性を示し、RT-4、U-251 MGの細胞ライセートおよび肝組織ライセートを用いたWBでは、予測されるサイズのバンドを検出します。
* ヒトとげっ歯類の両サンプルのWB
MMP9
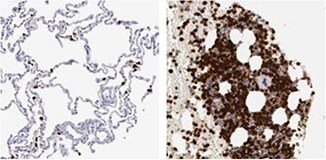
抗MMP9抗体(HPA001238)を使用したヒト肺組織のIHC染色では、マクロファージおよび骨髄組織の骨髄造血細胞に強力な核陽性が認められます。

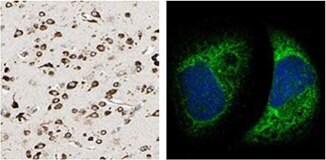
抗MTDH抗体(HPA010932)を使用したIHC染色では、ヒト大脳皮質組織の神経細胞に強力な細胞質陽性が認められます。A-431細胞株を用いたICC-IFでは、本抗体は小胞体を染色します。

モノクローナル抗MTDH抗体(AMAb90762)を使用したIHC染色では、乳がんおよび大腸がんサンプル由来の腫瘍細胞に強力な細胞質反応が認められます。
P5C脱水素酵素/ALDH4A1

抗ALDH4A1抗体(HPA006401)を使用したIHC染色では、ヒト腎臓および肝臓組織に顆粒状パターンを示す強力な細胞質陽性が認められます。
* ヒトとげっ歯類の両サンプルのWB
PITRM1/MP1

抗PITRM1抗体(HPA006753)のIHC染色では、ヒト心筋細胞で強力な細胞質陽性が認められます。ヒト細胞株U-251 MGのICC-IF染色では、ミトコンドリアで陽性が認められます。
PRC1

抗PRC1抗体(HPA034521)を用いたヒト精巣組織のIHC染色では、輸精管細胞に強力な核陽性が認められます。ICC-IFでは、A-431細胞の核、細胞膜および微小管に染色が認められます。
SCOT/OXCT1

抗OXCT1抗体(HPA012047)を用いたヒト心筋および腎臓のIHC染色では、心筋細胞および尿細管の細胞に陽性が認められます。ICC-IFでは、A431細胞のミトコンドリアに染色が認められます。
* ヒトとげっ歯類の両サンプルのWB
抗KLHL26抗体(HPA023074)を使用したIHC染色では、異なる患者由来の乳がんサンプルで膜/細胞質のさまざまな染色パターンが認められます。

抗ACSF2抗体(HPA024693)は、異なる患者由来の乳がん細胞で粒状に染色される強力な陽性から陰性まで強度の異なる細胞質陽性を示します。
抗GCM1抗体(HPA011343)は、乳がん細胞で膜陽性を示しますが、正常乳房組織では陰性です。
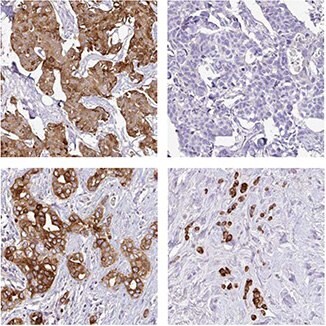
抗AGR3抗体(HPA053942)は、12例中11例の乳がん患者で強力な細胞質陽性を示しますが、1例では完全に陰性です。
* ヒトとげっ歯類の両サンプルのWB
がんバイオマーカーの特定
乳がん
乳がんは二番目によくみられるがんであり、女性では群を抜いて最も発現頻度が高いがんです。乳がんの発生率は、徐々に増加していますが、それに伴った死亡率の増加はみられません。初期に見つかる場合、先進国では予後は比較的良好であり、一般的な5年生存率は約85%です。
乳がんと治療
がんは、しばしば単一疾患として称されますが、実は多種の疾患を指します。このような理解は、長年かけて進展してきましたが、多くの患者が自分に最適ながん治療を受けられていません。がん患者がより個々に適した治療を受けるために、患者を層別化する、より良い方法が引き続き求められています。腫瘍のステージおよびグレードのような従来の予後ファクターは患者の予後を正しく推測するには不十分です。がんバイオマーカーから得られる追加情報は、このような推測を大幅に改善し、最終的には個々の患者に適した治療法を導き出し、治療不十分および過剰治療を回避することができると期待されています。
乳がん患者の主要な治癒的治療は外科手術であり、しばしば、アジュバント療法を併用します。しかし、アジュバント療法は、かなり費用がかかり、重篤な副作用を伴うこともあるため、今日、医師は乳がん治療において主に臨床的に必要なことは過剰治療の低減であると認識しています。したがって、患者をさまざまな予後分類に層別化することは、医師が患者に最も適した治療を選択するための助けとなると考えられるため、非常に重要です。
大半の乳がんは、ホルモン受容体に反応を示します。すなわち、エストロゲン受容体(ER)および/またはプロゲステロン受容体(PR)を発現しています。これらの受容体を発現する腫瘍を有する患者は、タモキシフェンのようなアジュバント内分泌療法を受けることができます。
また、乳がんはHER2タンパク質(ヒト上皮成長因子受容体2)を発現することもあり、このようなタンパク質を発現する腫瘍を有する患者は、トラスツズマブによるアジュバント療法を受けることができます。
アジュバント療法には、化学療法または放射線療法も含まれることがあります。
RBM3
RNA結合モチーフタンパク質3(RBM3)は、RNAおよびDNA結合タンパク質です。これらの機能は完全には解明されていません。軽度低体温、およびグルコース欠乏や低酸素症のような細胞ストレスと関連するその他の状態の初期事象として、発現することが明らかになっています1。ストレスがかかるときに、RBM3は、生存に必要なタンパク質合成の維持を促進して細胞を保護すると考えられています1。最近、RBM3は前立腺がん細胞において幹細胞様の特性を弱めることも示されました2。
Human Protein Atlas(HPA)プロジェクトの一環(proteinatlas.org)として検討された複数のがんで認められる差次的発現パターンから、RBM3は、HPAにより腫瘍バイオマーカー候補として特定されています3,4。
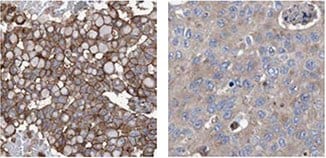
抗RBM3抗体のHPA003624を用いたIHC解析において、正常な乳房組織における発現パターンは弱いものでしたが、乳がん組織では階層的な発現パターンが認められました(図1)。研究者らがさらに大規模な乳がんコホートを対象として詳細に発現を検討したところ、RBM3の発現は、長期生存と関連することが明らかになりました5。

図1. 抗RBM3抗体(HPA003624)を用いた免疫組織染色では、正常乳房組織(A)では弱い発現が認められ、乳がんサンプル(B、C)では弱い~強いまでさまざまな強度の差次的発現が認められています。
乳がんの予後バイオマーカーとしてのRBM3
研究者らは、RBM3を予後バイオマーカー候補として特定した後、 さらに大規模な乳がんコホートを対象にRBM3タンパク質の発現を検討しました1。ステージIIの浸潤性乳がんを認める閉経前の女性500例から成るコホートにおいて、RBM3の発現は、小型で、低グレードの、エストロゲン受容体(ER)陽性の腫瘍と関連していることが明らかになりました。ER陽性患者の一部について解析したところ、RBM3は、無再発生存(RFS)の独立した予測因子でした。図2に示すとおり、RBM3タンパク質の発現量が多い腫瘍の患者の生存は、RBM3発現量が少ない腫瘍の患者よりも良好でした。
RBM3タンパク質の発現は、さまざまながん型の多数の患者コホートを対象にしてさらに詳細に解析されています。RBM3の発現量は、乳がん1、結腸がん2、卵巣がん3,4、精巣がん、尿路上皮がん5、および前立腺がん6、ならびに悪性黒色腫7の患者の生存と有意な関係があることが明らかになっています。
RBM3は乳がんおよびその他の複数のがんにおいて良好な予後を示すマーカーであると結論されます。

図2.ER陽性乳がん患者におけるRBM3発現量で分類した無再発生存(RFS)についてのKaplan-Meier(生存)解析。患者をRBM3発現量の高低に基づいて2群に分けています。
RBM3抗体
Atlas抗体製品では、ポリクローナルHPA003624およびPrecisAモノクローナルAMAb90655の2種の抗RBM3抗体を取り揃えています。モノクローナル抗RBM3抗体AMAb90655は、ヒト細胞株のウェスタンブロッティングにおいて優れた特異性が認められており、IHCのホルマリン固定パラフィン包埋組織の染色に一般的に使用されています(図3)。

図3.乳がん(左)および前立腺がん(右)についてのAMAb90655を使用したRBM3発現のIHC分析では、腫瘍細胞で核陽性が認められます。AMAb90655を用いて行ったヒト細胞株RT4ライセートのWBの画像には、17 kDaの予測されるバンドが認められます。
参考文献
Granulin
Granulinは、単一の前駆体タンパク質から開裂して生成される分泌型グリコシル化ペプチドのファミリーです。単一ペプチドの開裂によって、成熟Granulinが産生されますが、それはさらに開裂を受けてさまざまな活性型ペプチドになる可能性もあります。これらの開裂産物は、Granulin A、Granulin B、Granulin Cなどと呼ばれます。ペプチドと開裂していないGranulinタンパク質はどちらも、細胞増殖を制御します。Granulinタンパク質ファミリーに属する種々のメンバーは、細胞増殖に対して阻害因子、刺激因子として働く場合もあり、両作用を持っていることもあります。Granulinファミリーメンバーは、正常な成長、創傷治癒および腫瘍形成において重要です[2008年7月、RefSeq提供]。
Elkabetsらの論文では、GRNを発現させた骨髄細胞をマウスの応答腫瘍へリクルートすることによって、応答腫瘍の発生におけるGRN発現の役割を研究しています1。特定の腫瘍は他の腫瘍増殖または解剖学的に遠位に位置する転移細胞の増殖を助長することができ、それは腫瘍の扇動(tumor instigation)と呼ばれています。この研究において、激しく増殖するヒト乳がん細胞をマウスに移植したところ、これらの細胞は、移植しなければ、腫瘍形成性が低く不活性である形質転換細胞を刺激して増殖させることが明らかになりました。GRNは、扇動している骨髄細胞の中で最も増加させる遺伝子と特定されました。GRN発現細胞は、常在の線維芽細胞を誘導して、悪性腫瘍の増殖を促進する遺伝子を発現させました。 抗がん療法において、GRNまたは活性化されたGRN発現細胞をターゲットにすることにより、これらの細胞株によるがん増殖促進の伝達を破壊できるのではないかと推測されました。
抗GRN抗体のHPA028747を使用して乳がん患者のコホート由来の腫瘍組織を解析したところ、高いGRN発現は、悪性度の非常に高いトリプルネガティブ基底細胞様腫瘍のサブタイプと相関し、患者生存率の低下と関連することが明らかになりました(図4)。
参考文献
![抗体HPA028747により、GRN発現は悪性の腫瘍サブタイプと相関がみられ、乳がん患者の生存率低下と関係することが示されました。左の図は、各カテゴリー[トリプルネガティブ(TN)/基底細胞腫瘍または基底細胞以外の腫瘍]において高いGRN陽性を示す腫瘍の割合を示します。右のKaplan-Meier解析は、GRN発現陽性の場合(緑)またはGRN発現陰性の場合(青)と生存の関係を示しています。 grn-expression](/deepweb/assets/sigmaaldrich/marketing/global/images/technical-documents/articles/research-and-disease-areas/cancer-research/grn-expression/grn-expression.jpg)
図4.抗体HPA028747により、GRN発現は悪性の腫瘍サブタイプと相関がみられ、乳がん患者の生存率低下と関係することが示されました。左の図は、各カテゴリー[トリプルネガティブ(TN)/基底細胞腫瘍または基底細胞以外の腫瘍]において高いGRN陽性を示す腫瘍の割合を示します。右のKaplan-Meier解析は、GRN発現陽性の場合(緑)またはGRN発現陰性の場合(青)と生存の関係を示しています。

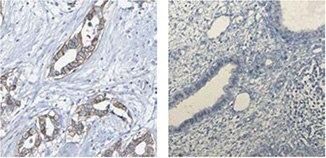
抗GRN抗体(HPA008763)を用いたヒト膵臓組織のIHC染色では外分泌腺細胞に強力な細胞質陽性が認められます。ICC-IFでは、A-431細胞の小胞に陽性が認められます。

抗GRN抗体HPA028747 を用いたIHC分析では、正常十二指腸組織の腺細胞に強力な細胞質陽性が認められ、U-251 MG細胞では小胞陽性が認められます。
Anillin
Anillinは、マイクロフィラメント(細胞骨格成分の1つ)のサブユニットであるアクチン結合タンパク質です。Anillinは、大部分の細胞に発現し、運動性、分裂およびシグナル伝達のような基本的な細胞機能に関与しています。Anillin発現の研究から、いくつかのヒト腫瘍でAnillinが過剰発現していることが明らかになっています。
乳がんにおける治療の予後予測バイオマーカーとしてのAnillin
Anillinの発現は、乳がんと診断された患者から得られた467件のサンプルから成る患者コホートを対象に、抗ANLN抗体のHPA005680により分析されています。Anillinの発現量が高い腫瘍を有する患者の無再発生存率(RFS)は、Anillin発現量の低い患者のRFSよりも低くなりました(図5A)。乳がん特異的生存率(BCSS)を分析したところ、Anillin発現と生存率低下に同様の関係が認められると考えられました(図5B)。O´Learyらによる研究では、Cox回帰分析により、Anillinの予後に対する影響が確認されました。単変量解析、ならびに診断時の腫瘍サイズとグレード、年齢、リンパ節、 ER、PR、HER2およびKi67 の状態について調整した多変量解析では、Anillin発現量が高いとBCSSおよびRFSに低下が見られました。
Anillinは乳がんについて予後不良を示すマーカーであると結論されます。

図5. 乳がん患者におけるAnillin発現量で分類した無再発生存率(A)および乳がん特異的生存率(B)についてのKaplan-Meier(生存)解析患者をAnillin発現量の高低に基づいて2群に分けています。
参考文献
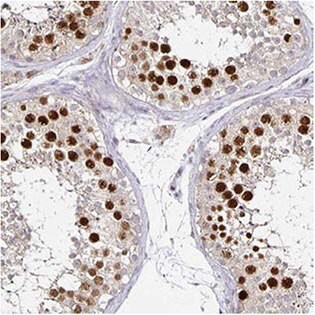

抗ANLN抗体(HPA005680)は、IHCにより、ヒト精巣内の輸精管細胞で強力な核陽性を示します。ICC-IFでは、A-431細胞の核(核小体ではない)は陽性を示します。また、本抗体は、RT-4およびU-251 MGの細胞ライセートを用いたWBで、予測されるサイズのバンドを検出します。

抗ANLN抗体AMAb90660は、肺腺がんの一部の腫瘍細胞で強力な核免疫反応を示し、ヒト細胞株U-251 MGにおいて予測されるサイズのバンドを検出します。

抗ANLN抗体AMAb90662は、大腸がんの一部の腫瘍細胞で強力な核免疫反応を示し、ヒトU-251 MG細胞において予測されるサイズのバンドを検出します。
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?