背景
「制限酵素」という用語は、ヴェルナー・アーバーとマシュー・メセルソンの研究所で行われた腸内細菌ファージλ(ラムダファージ)の研究に由来しています。ファージDNAの酵素的切断によってラムダファージの活性を阻害する特定の大腸菌株の能力が研究され、この成長制限の原因となる酵素に制限酵素という名称が与えられました。1, 2, 3
ヴェルナー・アーバー、ダニエル・ネイサンズ、ハミルトン・O・スミスは、組換えDNA技術の開発につながった制限酵素の発見と特性評価により、1978年にノーベル生理学・医学賞を受賞しました。
はじめに
制限酵素は、制限部位と呼ばれる特定の認識配列またはその近くでDNAを切断することから、「分子のハサミ」とも呼ばれます。これらの酵素は、DNAの2本の鎖のそれぞれに切開を1回行うもので、制限エンドヌクレアーゼとも呼ばれます。4
ウイルスは、DNAを細胞に注入することによって宿主細胞に感染します。このウイルスDNAは、ウイルスの子孫を複製するために宿主細胞の機構をハイジャックし、宿主細胞を死に至らしめます。ウイルス感染を克服するために、多くの細菌や古細菌がいくつかのメカニズムを発展させてきました。主な保護メカニズムとしては、制限酵素を使用して、侵入したウイルスDNAを特定の制限部位で切断することによって分解することが挙げられます。同時に、宿主細胞は、宿主認識配列内のアデニンまたはシトシン塩基をメチル化する、メチラーゼと呼ばれる他の酵素を使用することにより、自身のDNAが切断されるのを防ぎます。宿主細胞は、制限酵素のそれぞれに対応するメチラーゼを生成し、宿主DNAをメチル化して分解から保護します。これらの酵素は、制限修飾(R-M)系を構成します。
制限酵素は、DNAのホスホジエステル骨格における3つの酸素原子とリン原子の間の結合の加水分解を触媒します。制限酵素の活性には、Mg2+等の二価イオンが必要です。
命名法
スミスとネイサンズは、1973年に制限エンドヌクレアーゼの命名ガイドラインを提案しました。これらのガイドラインによると、制限酵素の名称は斜体表記の3文字の頭字語で始まります。最初の文字は、酵素が単離された細菌属の最初の文字を示し、次の2文字は細菌種を表します。これらの後に、血清型または菌株を示す追加の文字または数字が続く場合があります。この後にスペースと、同定された順序を示すローマ数字が続きます。たとえば、Hind IIIは、インフルエンザ菌血清型dから分離された4つの酵素のうちの3番目であったことを示しています。6
制限酵素の型
制限エンドヌクレアーゼは、組成、切断部位の特徴、および補因子の要件に基づいて、I型、II型、III型、およびIV型の4つのグループに分類されます。
この表では、認識部位は赤い矢印で示されています。
制限酵素の活性に影響を与える要因
制限酵素は、基質DNAと反応条件に応じてさまざまな切断活性を示し、スター活性を生じさせる可能性があります。望みの切断を得るためには、以下の要因を制御することが重要となります。
- スター活性:反応条件が至適でない場合、一部の制限酵素は、定義された認識配列とは異なる部位、すなわち非特異的な部位の塩基配列を切断します。この現象をスター活性と呼びます。スター活性を誘発する要因の例としては、高濃度の塩分およびグリセロール、不純物の存在、基質DNAと比較して過剰な酵素、インキュベーション時間の増加、不適合なバッファー条件と補因子などがあります。
- メチル化されたDNA:一部のDNA分子は認識部位でメチル化されており、特定の制限酵素による切断に耐性があります。たとえば、ほとんどの大腸菌株は、特定の認識部位をメチル化してG6mATCまたはC5mCA/TGGを形成する、DamまたはDcmメチルトランスフェラーゼを発現します。G6mATCは、Mbo Iによる切断に耐性があります。
- 温度:ほとんどのエンドヌクレアーゼは、37℃でターゲットDNAを最適に消化します。ただし、至適温度がそれよりも低いまたは高い場合の例外がいくつかあります。たとえば、Taq Iは65℃で最適に分解し、Apa I(カタログ番号10899208001)は25℃で分解します。
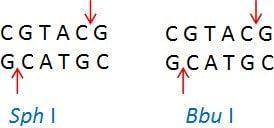
イソシゾマーとネオシゾマー6
イソシゾマーは、同じ認識配列と切断部位を持つ制限酵素です。例:Sph I(CGTAC/G)およびBbu I(CGTAC/G)
ネオシゾマーは、同じ認識配列を持つ制限酵素ですが、その配列内の異なる部位でDNAを切断します。例:Tai I(ACGT/)およびMae II(A/CGT)

制限消化の生成物
二本鎖DNAの制限消化により、突出粘着末端と平滑末端という2種類の末端が生成されます。
平滑末端は、ライゲーションを促進する5-リン酸基を有し、他の平滑末端DNAと普遍的に適合します。
EcoR Vによって生成された平滑末端

粘着末端は、セルフライゲーションまたは別のDNA分子の相補性領域とのライゲーションが可能な一本鎖DNAの短区間です。突出粘着末端は、3’末端または5’末端に1〜4個のヌクレオチドからなるオーバーハングを有します。
Bln I(カタログ番号11558170001)によって生成された5’突出粘着末端

Kpn Iによって生成された3’突出粘着末端

バッファー系
メルクの制限酵素コレクションは、5つの独自バッファーを使用した消化用に最適化されています。単一の酵素を使用してDNAを消化する場合は、その酵素に付属のバッファーを使用します。DNAの二重消化には、両方の酵素が100%の活性を示すバッファーを使用します。または、一般的な二重消化のチャートから至適バッファーを決定することもできます。場合によっては、バッファーの不適合性(組成または温度)のために、順次分解が推奨されます。単一酵素、2つの制限酵素、および順次DNA消化による制限消化のプロトコールについては、「制限酵素消化プロトコール」を参照してください。適切なバッファーを選択することは、両方の酵素で高い活性を得るためだけでなく、スター活性を回避するためにも重要です。5つの選択肢が用意されているため、ユーザーは目的の消化に適したバッファーを選択ことが可能です。ほとんどの消化には1〜2個のバッファーが必要であり、これらのバッファーを使用することで費用効果が高くなります。
アプリケーション
特定の認識部位でDNAを切断する制限エンドヌクレアーゼの能力により、複数の分子生物学技術に不可欠なツールとしてこれらの酵素を幅広く使用できるようになりました。主な用途について以下に説明します。
- 分子クローニング:制限酵素の一般的な用途は、組換えDNA分子の生成です。このプロセスでは、制限酵素によるドナーDNA(通常はプラスミド)とベクターDNA(通常は別の生物の遺伝子)を切断して、適合性のある末端を生成します。これらの末端は、「平滑」または「突出粘着」のいずれかになります。2つの切断されたDNAは、DNAリガーゼと呼ばれる酵素を使用して結合され、組換えDNA分子を生成します。次に、この組換えDNAを宿主生物に導入して複製することができます。詳細については、『制限酵素クローニングマニュアル』を参照してください。
- 制限マッピングとしても知られるDNAマッピングでは、制限エンドヌクレアーゼを使用してDNAフラグメントまたはゲノムの構造情報を取得します。マッピングでは、ゲノム内の制限酵素部位の順序を決定します。構造決定の対象となる目的のDNAは、一連の制限エンドヌクレアーゼで切断され、サイズの異なるDNAフラグメントが生成されます。これらのフラグメントをアガロースゲルで分離して、目的のDNAの構造を決定します。
制限エンドヌクレアーゼを使用すると、特定のDNAフラグメントの既知の制限酵素部位に基づいて、そのDNAフラグメントの同一性を検証できます。 - 制限酵素ランドマークゲノムスキャニングは、制限酵素の組み合わせを利用して、特定の生物のゲノム全体でメチル化レベルの差異を視覚化するゲノム分析方法です。任意のDNAの正常値からの逸脱を特定するための便利な手法であり、腫瘍の高/低メチル化、遺伝子の欠失または増幅、生物の発生過程における遺伝子発現の変化などを検出するのに非常に効果的です。8
- 遺伝子シーケンシング:大きなDNA分子を制限酵素で消化し、得られたフラグメントをDNAシーケンサーで処理することで塩基配列を決定できます。
- 制限酵素断片長多型(RFLP)法では、制限酵素を使用してDNAサンプルを消化し、ゲル電気泳動によって長さに基づいてこれらの断片を分離し、膜に転写します。次に、制限酵素部位で囲まれた特定の配列を標的とする放射性または蛍光標識プローブに、これらのフラグメントを結合します。結果として得られたフラグメントの長さが個体間で異なる場合、RFLPが発生します。各個体には、「生物学的バーコード」と呼ばれる固有のパターンがあります。この手法は、遺伝子マッピング、遺伝性疾患の原因となっている遺伝子の同定、疾患のリスクの判定、親子鑑定で使用された最初のDNA型鑑定技術でした。9
- パルスフィールドゲル電気泳動では、大きなDNAフラグメント(主に細菌ゲノムを切断頻度の低い制限酵素で消化したフラグメント)を分離します。生成された独自のパターンは、細菌のさまざまな菌株を区別するために使用されます。エピデミックの原因として特定の菌株を特定することが有用な場合があります。10, 11
- 遺伝子発現連鎖解析(SAGE)法は、小さなタグの形で多数の転写産物を定量的かつ同時に解析する手法です。この手法では、制限酵素がアンカー型酵素およびタグ付け酵素として使用されます。12
- 制限酵素仲介型挿入変異株作成(REMI)法では、制限酵素を使用して、制限酵素で線形化されたプラスミドDNAの混合物を挿入するための適合性のある突出粘着末端をゲノムに生成します。プラスミドDNAは、染色体の同族の制限部位へのDNAの組み込みを促進する制限酵素とともに宿主細胞に形質転換されます。この手法は、遺伝子スクリーニングに加え、ゲノムの特定のポイントに遺伝子マーカーと分子マーカーを挿入して、変異表現型に基づいて特異な遺伝子を同定する場合に役立ちます。13
参考文献
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?