Metody znakowania digoksygeniną (DIG)
- Znakowanie digoksygeniną (DIG) i przeciwciałem anty-DIG
- Znakowanie DNA DIG metodą PCR
- Znakowanie DNA DIG Random Primed
- Nick Translation Labeling of dsDNA for In Situ Probes
- Transkrypcyjne znakowanie sond RNA
- Znakowanie końca 5' oligonukleotydu DIG, znakowanie końca 3' i znakowanie ogona 3'
- .Oszacowanie wydajności sondy za pomocą procedury bezpośredniego wykrywania
- Pliki do pobrania i zasoby związane z DIG
Znakowanie digoksygeniną (DIG) i przeciwciałem anty-DIG
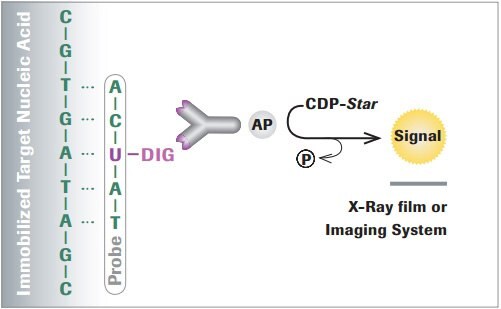
System DIG to nieradioaktywna technologia znakowania i wykrywania kwasów nukleinowych do wielu zastosowań. System oparty jest na steroidzie wyizolowanym z roślin naparstnicy (Digitalis purpurea i Digitalis lanata). Rośliny te są jedynym naturalnym źródłem digoksygeniny, więc przeciwciało anty-DIG nie wiąże się z innym materiałem biologicznym, zapewniając specyficzne znakowanie. Ze względu na tę wysoką specyficzność, potrzeba mniej materiału w porównaniu do znakowania radioaktywnego, dzięki czemu system DIG jest idealny do analizy hybrydyzacji nukleinowej. Unieruchomione kwasy nukleinowe są hybrydyzowane z sondą znakowaną DIG, a następnie wykrywane przy użyciu przeciwciał anty-digoksigeninowych o wysokim powinowactwie, sprzężonych z fosfatazą alkaliczną (AP), peroksydazą chrzanową (HRP), fluoresceiną lub rodaminą do wykrywania kolorymetrycznego, chemiluminescencyjnego lub fluorescencyjnego.

Rysunek 1.Przykład wykrywania kwasów nukleinowych znakowanych DIG przy użyciu substratów chemiluminescencyjnych.
Znakowanie DNA DIG metodą PCR
Znakowanie metodą PCR jest preferowaną metodą przygotowywania sond znakowanych DIG, gdy matryca jest dostępna tylko w ograniczonych ilościach, jest częściowo oczyszczona lub jest bardzo krótka. Wymaga mniej optymalizacji niż inne metody i zapewnia wysoką wydajność znakowanej sondy. W znakowaniu PCR, termostabilna polimeraza zawiera DIG-dUTP podczas amplifikacji określonego regionu matrycowego DNA. Wynikiem jest wysoce znakowana, specyficzna i niezwykle czuła sonda hybrydyzacyjna.
Zasada reakcji
Podczas standardowej reakcji PCR, Digoksygenina-11-dUTP jest włączana do nowo zsyntetyzowanego DNA. Jedynym warunkiem wstępnym jest to, że niektóre informacje o sekwencji docelowej są potrzebne do syntezy odpowiednich starterów. Nieradioaktywny system DIG wykorzystuje digoksygeninę, hapten steroidowy, do znakowania DNA, RNA lub oligonukleotydów w celu hybrydyzacji, a następnie wykrywania koloru lub luminescencji. Digoksygenina jest sprzężona z dUTP poprzez alkaliczne wiązanie estrowe. Znakowany dUTP może być łatwo włączony przez enzymatyczną syntezę kwasu nukleinowego przy użyciu polimeraz DNA. Połączenie nieradioaktywnego znakowania z PCR jest potężnym narzędziem do analizy produktów PCR oraz do przygotowania znakowanych sond z niewielkich ilości odpowiedniej sekwencji docelowej.
Znakowanie DNA DIG metodą PCR cechy i korzyści
Warunki PCR
- Zoptymalizuj parametry amplifikacji PCR (warunki cyklu, stężenie matrycy, sekwencja starterów i stężenie starterów) dla każdej matrycy i zestawu starterów przy braku DIGdUTP przed próbą włączenia DIG.
Szablon
- Aby uzyskać najlepsze wyniki, użyj sklonowanych wstawek jako szablonu. Genomowe DNA może być trudniejsze w użyciu.
- Stężenie szablonu jest kluczowe dla pomyślnej produkcji specyficznych sond.
Znakowanie
Zestaw do syntezy sondy DIG metodą PCR .DIG Probe Synthesis Kit wymaga mniej optymalizacji niż większość metod znakowania, ponieważ zawiera Expand High Fidelity Enzyme Blend. Zalety tej mieszanki enzymów obejmują:
- Może wydajnie wykorzystywać regiony bogate w GC jako szablon
- Dla większości szablonów nie wymaga optymalizacji stężenia MgCl2 a reakcje znakowania będą działać przy standardowych stężeniach 1.5 mM MgCl2
DIG Random Primed DNA Labeling
Metoda "random primed" znakowania DNA opiera się na hybrydyzacji mieszaniny wszystkich możliwych heksanukleotydów do matrycy DNA. Wszystkie kombinacje sekwencji są reprezentowane w mieszaninie primerów heksanukleotydowych, co prowadzi do wiązania primera z matrycą DNA w sposób statystyczny. W ten sposób gwarantowany jest równy stopień znakowania na całej długości matrycowego DNA. Nić komplementarna jest syntetyzowana z końców 3´ OH losowego startera heksanukleotydowego przy użyciu enzymu Klenowa, klasy znakującej. Zmodyfikowane trifosforany dezoksyrybonukleozydów ([32P]-, [35S]-, [3H]-, [125I]-, digoksygenina lub biotyna) obecne w reakcji są włączane do nowo zsyntetyzowanej komplementarnej nici DNA.
Te znakowane sondy są szczególnie odpowiednie do wykrywania pojedynczych kopii genów na genomowych Southern blotach, przesiewowych bibliotekach rekombinowanych, dot/slot blotach i northern blotach. Ponieważ każdy starter ma inną sekwencję sześciu zasad, produkt znakowanej sondy będzie zbiorem fragmentów o zmiennej długości. Tak więc, wyznakowana sonda pojawi się jako rozmaz, a nie unikalne pasmo na żelu. Rozkład wielkości znakowanej sondy zależy od długości oryginalnej matrycy.
Zasada działania
W znakowaniu losowym enzym Klenowa kopiuje matrycę DNA w obecności starterów heksamerowych i alkalilabilnego DIG-11-dUTP. Enzym wstawia średnio jedną cząsteczkę DIG w każdym odcinku 20-25 nukleotydów. Otrzymany znakowany produkt jest jednorodnie znakowaną, czułą sondą hybrydyzacyjną zdolną do wykrycia zaledwie 0,10 - 0,03 pg docelowego DNA.
Nick Translation Labeling of dsDNA for In Situ Probes
Metoda translacji nicków opiera się na zdolności DNazy I do wprowadzania losowo rozmieszczonych nicków do DNA przy niskich stężeniach enzymu w obecności Mg2+.Polimeraza DNA I syntetyzuje DNA komplementarne do nienaruszonej nici w kierunku 5´ - 3´, wykorzystując zakończenia 3´-OH nicka jako starter. Aktywność egzonukleolityczna 5'-3' polimerazy DNA I jednocześnie usuwa nukleotydy w kierunku syntezy. Aktywność polimerazy sekwencyjnie zastępuje usunięte nukleotydy trifosforanami deoksyrybonukleozydów znakowanymi izotopami lub haptenami. W niskiej temperaturze (+15 °C) nieznakowane DNA w reakcji jest zastępowane przez nowo zsyntetyzowane znakowane DNA. W przypadku procedur hybrydyzacji in situ długość znakowanych fragmentów uzyskanych z tej procedury powinna wynosić około 200-500 zasad.
Transkrypcyjne znakowanie sond RNA
Dla niektórych zastosowań, RNA znakowane DIG jest bardziej efektywną sondą hybrydyzacyjną niż DNA znakowane DIG. Na przykład, sondy RNA znakowane DIG mogą wykrywać rzadkie mRNA w nanogramowych ilościach całkowitego RNA. Te znakowane sondy RNA są generowane przez in vitro transkrypcję z matrycy DNA. W metodzie transkrypcji RNA, DNA jest klonowane w miejscu wielokrotnego klonowania wektora transkrypcyjnego pomiędzy promotorami dla różnych polimeraz RNA (takich jak polimeraza RNA T7, SP6 lub T3). Szablon jest następnie linearyzowany przez rozszczepienie wektora w unikalnym miejscu (w pobliżu wstawki). Polimeraza RNA transkrybuje wstawiony DNA do antysensownej kopii RNA w obecności mieszaniny rybonukleotydów (w tym DIG-UTP). Podczas reakcji, DNA może być wielokrotnie transkrybowane (nawet stukrotnie) w celu wygenerowania dużej ilości kopii RNA znakowanych DIG o pełnej długości (10-20 μg RNA z 1 μg DNA w standardowej reakcji). DIG jest włączany do RNA w przybliżeniu co 25-30 nukleotydów.
Zasada działania
Szablon DNA przeznaczony do transkrypcji jest klonowany w miejscu polilinkera odpowiedniego wektora transkrypcyjnego, który zawiera promotory dla polimeraz RNA SP6 lub T3 i T7. Po linearyzacji w odpowiednim miejscu, RNA jest transkrybowany w obecności DIG-11-UTP. W standardowych warunkach, około 10 μg RNA znakowanego DIG o pełnej długości jest transkrybowane z 1 μg matrycy.
Następujące wskazówki są krytyczne dla udanego znakowania sond RNA:
RNazy
RNazy są wszechobecne i nie wymagają żadnych kofaktorów do aktywności. Jeśli chcesz odnieść sukces, podejmij wszelkie możliwe środki ostrożności, aby zapobiec zanieczyszczeniu RNazami. Na przykład:
- Zaleca się stosowanie jednorazowych naczyń plastikowych, naczyń szklanych pieczonych w piecu lub naczyń plastikowych, które zostały odkażone za pomocą RNase ZAP lub podobnych odczynników.
- Przygotuj wszystkie roztwory za pomocą wody, która została potraktowana pirokarbonianem dietylu (DEPC) lub dwuwęglanem dimetylu (DMDC) i autoklawuj roztwory.
- Noś rękawiczki podczas całej procedury.
- Wydajność znakowania zależy w dużej mierze od czystości matrycy DNA. Szablon powinien być wysoce oczyszczony.
- Końcowy szablon musi być linearyzowany, ekstrahowany fenolem/chloroformem i wytrącony etanolem.
Sekwencja szablonu
- Niektóre startery i/lub regiony polilinkerów w szablonach DNA są homologiczne do części sekwencji rybosomalnego 28s i 18s RNA. Dlatego znakowane sondy mogą generować specyficzne, ale niepożądane sygnały w próbkach zawierających te znaczące RNA. Aby zminimalizować ten efekt, należy usunąć jak najwięcej sekwencji polilinkerów z matrycy.
- Jeśli do wykonania matrycy DNA użyto reakcji PCR, produkt reakcji Expand High Fidelity zawiera niektóre fragmenty z pojedynczym nawisem 3´A. Ten nawis może wytwarzać produkty owijające się w reakcji znakowania transkrypcyjnego.
Długość szablonu
- Optymalna długość szablonu wynosi około 1 kb
- Minimalna długość powinna wynosić co najmniej 200 bp
Przechowywanie sondy
- W celu zapewnienia długoterminowej stabilności, sondy RNA powinny być podzielone na porcje i przechowywane w temperaturze -20 °C lub -70 °C<
- Sondy RNA znakowane DIG są stabilne przez co najmniej 1 rok w temperaturze -20 °C lub -70 °C w etanolu
Czułość sondy
- Aby szybko określić czułość antysensownej sondy RNA znakowanej DIG, należy przygotować odpowiedni sensowny RNA (nieznakowany) poprzez transkrypcję in vitro . Następnie użyj oczyszczonego transkryptu sensu w różnych stężeniach jako celu na northern blot. Na podstawie wyniku blot można łatwo określić najniższą ilość celu (transkrypt sensu), która może być wykryta przez znakowaną sondę (transkrypt antysensu).
DIG Oligonucleotide 5' End-Labeling, 3' End-Labeling, and 3' Znakowanie ogonowe
Dla niektórych zastosowań, takich jak hybrydyzacja in situ , syntetyczny oligonukleotyd znakowany DIG jest najlepszą sondą hybrydyzacyjną. Oprócz hybrydyzacji in situ , oligonukleotydy znakowane DIG mogą być używane jako sondy hybrydyzacyjne w wielu zastosowaniach, w tym w dot/slot blotach, badaniach przesiewowych bibliotek, wykrywaniu powtarzających się sekwencji genów na Southern blotach i wykrywaniu obfitych mRNA na northern blotach.
Dostępnych jest kilka metod znakowania oligonukleotydów za pomocą DIG i zostały one podsumowane poniżej.
Oligonukleotyd 5´ znakowany końcowo estrem DIG-NHS
Oligonukleotydowe 3´ znakowanie końcowe za pomocą DIG-ddUTP
Dodanie 3´ ogona DIG-dUTP i dATP (około 40 - 50 reszt)
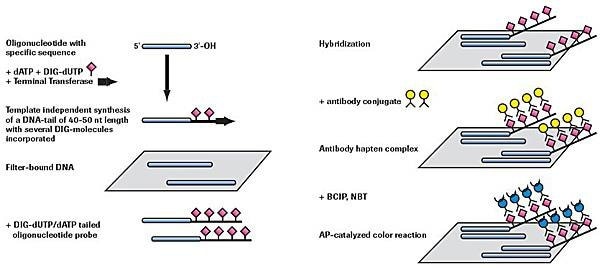
Rysunek 2.Nieradioaktywne ogonowanie i wykrywanie oligonukleotydów.
Estimation of Probe Yield by the Direct Detection Procedure
Aby dodać prawidłową ilość sondy do hybrydyzacji, należy najpierw określić ilość sondy znakowanej DIG wytworzonej w reakcji znakowania. Podana tutaj procedura bezpośredniego wykrywania porównuje ilość etykiety DIG w serii rozcieńczeń przygotowanych ze znakowanej sondy ze znanym stężeniem kontrolnego kwasu nukleinowego znakowanego DIG.
Uwaga: Jeśli znakujesz sondę DNA metodą PCR, nie musisz wykonywać bezpośredniego wykrywania w celu oceny wydajności.
Dla sond znakowanych metodą PCR należy użyć metody oceny elektroforezy żelowej.
Detekcja bezpośrednia obejmuje następujące kroki:
- Przygotowanie seryjnych rozcieńczeń znakowanej sondy i naniesienie ich na nylonową membranę (wymagany czas: 15 minut)
- Wykrywanie DIG w plamkach za pomocą chemiluminescencji; wymagany czas (2-2.5 godzin)
DIG-Related Downloads and Resources
Potrzebujesz pomocy? Sprawdź nasz Przewodnik wyboru produktów DIG.
Więcej informacji można znaleźć w następujących broszurach:
Szczegółowe informacje techniczne można znaleźć w podręcznikach DIG:
- Podręcznik aplikacji DIG dla hybrydyzacji filtrów
- DIG Application Manual for Nonradioactive In Situ Hybridization, 4th edition
Dowiedz się, jak obsługiwać system DIG w naszych poradach technicznych:
Wyłącznie do badań naukowych. Nie do użytku w procedurach diagnostycznych.
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?