10786357001
Roche
Ribonucleasa H (RNasa H)
from Escherichia coli H 560 pol A1
Sinónimos:
rnasa h
About This Item
Productos recomendados
origen biológico
Escherichia coli ( H 560 pol A1)
Nivel de calidad
Análisis
100%
formulario
solution
actividad específica
~40000 units/mg protein
envase
pkg of 100 U
fabricante / nombre comercial
Roche
técnicas
cDNA synthesis: suitable
color
colorless
pH óptimo
7.5-9.1
solubilidad
water: miscible
idoneidad
suitable for molecular biology
Nº de acceso NCBI
aplicaciones
life science and biopharma
actividad extraña
RNase, none detected (up to 10 U with MS- II- RNA)
endonuclease ~10 units, none detected (using lambda-DNA)
nicking activity 10 units, none detected
Condiciones de envío
dry ice
temp. de almacenamiento
−20°C (−15°C to −25°C)
Información sobre el gen
Escherichia coli ... rnhA(946955)
Descripción general
Origen: E. coli H560 pol A1
Tampón de almacenamiento: Tris-HCl 25 mM, KCl 50 mM, ditiotreitol 1 mM, EDTA 0,1 mM, glicerol al 50 % (v/v), pH 8,0 (+4°C)
Volumen de actividad: 1 x 103 U/ml analizadas según Hillenbrand y Staudenbauer.
Aplicación
- Inicio de la síntesis de ADN cebado con ARN in vivo
- Eliminación del ARNm durante la síntesis de la segunda cadena de ADNc
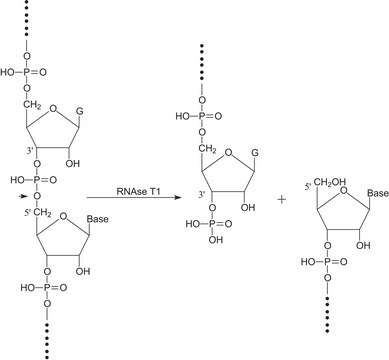
- Escisión específica de sitio del ARN
- Detección de regiones ARN:ADN en el ADN bicatenario de origen natural
- Eliminación de secuencias poli (A) del ARNm si hay oligo (dT) presentes
- Extracción de ARN y reacción en cadena de la polimerasa cuantitativa con transcriptasa inversa (RT-PCR)
Acciones bioquímicas o fisiológicas
Características y beneficios
- Elimina posibles fuentes de errores de la PCR.
- Aumenta la accesibilidad de los cebadores durante la PCR subsiguiente.
Calidad
Definición de unidad
Volumen de actividad: aproximadamente 1 U/μl
Nota de preparación
Almacenamiento y estabilidad
Otras notas
Código de clase de almacenamiento
12 - Non Combustible Liquids
Clase de riesgo para el agua (WGK)
WGK 1
Punto de inflamabilidad (°F)
does not flash
Punto de inflamabilidad (°C)
does not flash
Certificados de análisis (COA)
Busque Certificados de análisis (COA) introduciendo el número de lote del producto. Los números de lote se encuentran en la etiqueta del producto después de las palabras «Lot» o «Batch»
¿Ya tiene este producto?
Encuentre la documentación para los productos que ha comprado recientemente en la Biblioteca de documentos.
Los clientes también vieron
Nuestro equipo de científicos tiene experiencia en todas las áreas de investigación: Ciencias de la vida, Ciencia de los materiales, Síntesis química, Cromatografía, Analítica y muchas otras.
Póngase en contacto con el Servicio técnico