Wichtige Dokumente
R5125
Ribonuklease A aus Rinderpankreas
Type III-A, ≥85% RNase A basis (SDS-PAGE), 85-140 Kunitz units/mg protein
Synonym(e):
Pankreatische Ribonuklease, RNAsea, RNase A, Ribonuclease I
About This Item
Empfohlene Produkte
Biologische Quelle
bovine pancreas
Qualitätsniveau
Typ
Type III-A
Assay
≥85% RNase A basis (SDS-PAGE)
Form
lyophilized powder
Spezifische Aktivität
85-140 Kunitz units/mg protein
Mol-Gew.
~13,700
Methode(n)
cell based assay: suitable
Verunreinigungen
salt, essentially free
Eignung
suitable for molecular biology
Anwendung(en)
diagnostic assay manufacturing
Fremdaktivität
protease, essentially free
Lagertemp.
−20°C
SMILES String
[nH]1cnc(c1)CC(NC(=O)CCN)C(=O)O
InChI
1S/C9H14N4O3/c10-2-1-8(14)13-7(9(15)16)3-6-4-11-5-12-6/h4-5,7H,1-3,10H2,(H,11,12)(H,13,14)(H,15,16)
InChIKey
CQOVPNPJLQNMDC-UHFFFAOYSA-N
Suchen Sie nach ähnlichen Produkten? Aufrufen Leitfaden zum Produktvergleich
Verwandte Kategorien
Allgemeine Beschreibung
Anwendung
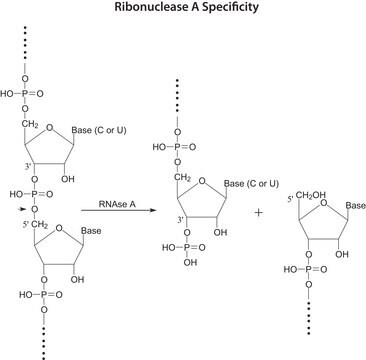
- RNase A wird zur Entfernung von RNA aus DNA-Plasmidpräparationen, aus Präparationen von genomischer DNA und aus Proteinproben verwendet.
- RNase A wird ferner in der RNA-Sequenzanalyse und in Schutzassays eingesetzt.
- RNase A wurde als Hilfsmittel für das computerunterstützte Wirkstoffdesign verwendet.
- RNase A unterstützt die Analyse von RNA-Sequenzen.
- RNase A hydrolysiert in Proteinproben enthaltene RNA.
- Die Aufreinigung von DNA wird durch Rnase A unterstützt.
Biochem./physiol. Wirkung
Leistungsmerkmale und Vorteile
Angaben zur Herstellung
Hinweis zur Analyse
Anwendung
Inhibitor
Signalwort
Danger
H-Sätze
P-Sätze
Gefahreneinstufungen
Resp. Sens. 1
Lagerklassenschlüssel
11 - Combustible Solids
WGK
WGK 3
Flammpunkt (°F)
Not applicable
Flammpunkt (°C)
Not applicable
Persönliche Schutzausrüstung
Eyeshields, Gloves, type N95 (US)
Hier finden Sie alle aktuellen Versionen:
Besitzen Sie dieses Produkt bereits?
In der Dokumentenbibliothek finden Sie die Dokumentation zu den Produkten, die Sie kürzlich erworben haben.
Kunden haben sich ebenfalls angesehen
Protokolle
This procedure may be used for determination of Ribonuclease A (RNase A) activity.
Chromatograms
application for HPLCUnser Team von Wissenschaftlern verfügt über Erfahrung in allen Forschungsbereichen einschließlich Life Science, Materialwissenschaften, chemischer Synthese, Chromatographie, Analytik und vielen mehr..
Setzen Sie sich mit dem technischen Dienst in Verbindung.