Wichtige Dokumente
E1014
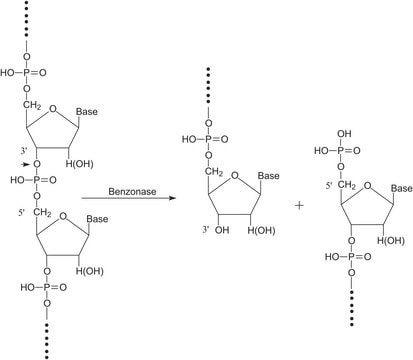
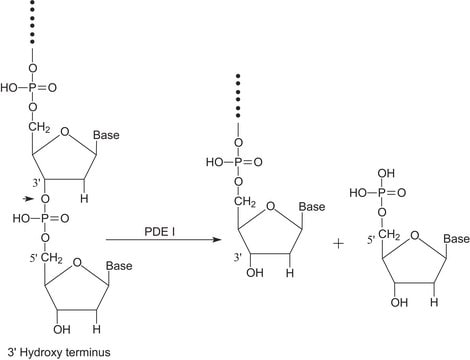
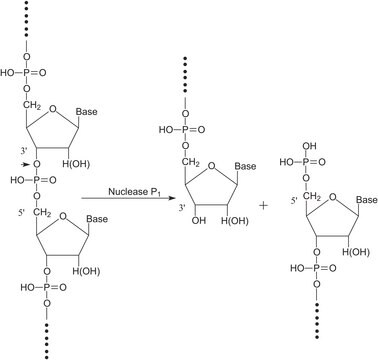
Benzonase® Nuklease
≥250 units/μL, ≥90% (SDS-PAGE), recombinant, expressed in E. coli, buffered aqueous glycerol solution
Synonym(e):
Endonuclease aus Serratia marcescens
About This Item
Empfohlene Produkte
Biologische Quelle
Serratia marcescens
Qualitätsniveau
Rekombinant
expressed in E. coli
Assay
≥90% (SDS-PAGE)
Form
buffered aqueous glycerol solution
Mol-Gew.
30 kDa
Konzentration
≥250 units/μL
Anwendung(en)
research use
Fremdaktivität
protease, essentially free
Versandbedingung
wet ice
Lagertemp.
−20°C
Suchen Sie nach ähnlichen Produkten? Aufrufen Leitfaden zum Produktvergleich
Allgemeine Beschreibung
Anwendung
Biochem./physiol. Wirkung
Leistungsmerkmale und Vorteile
- Depletion der Wirts-DNA in Mikrobiom-Proben.
- Effektiver Nukleinsäureverdau in verschiedenen Arbeitsabläufen.
- Reduktion der Viskosität bei der Proteinextraktion.
Einheitendefinition
Physikalische Form
Sonstige Hinweise
Rechtliche Hinweise
Lagerklassenschlüssel
10 - Combustible liquids
WGK
WGK 1
Flammpunkt (°F)
Not applicable
Flammpunkt (°C)
Not applicable
Persönliche Schutzausrüstung
Eyeshields, Gloves
Analysenzertifikate (COA)
Suchen Sie nach Analysenzertifikate (COA), indem Sie die Lot-/Chargennummer des Produkts eingeben. Lot- und Chargennummern sind auf dem Produktetikett hinter den Wörtern ‘Lot’ oder ‘Batch’ (Lot oder Charge) zu finden.
Besitzen Sie dieses Produkt bereits?
In der Dokumentenbibliothek finden Sie die Dokumentation zu den Produkten, die Sie kürzlich erworben haben.
Kunden haben sich ebenfalls angesehen
Artikel
This page lists nine frequently asked questions and answers about Benzonase® Nuclease.
The field of proteomics is continually looking for new ways to investigate protein dynamics within complex biological samples. Recently, many researchers have begun to use RNA interference (RNAi) as a method of manipulating protein levels within their samples, but the ability to accurately determine these protein amounts remains a challenge. Fortunately, over the past decade, the field of proteomics has witnessed significant advances in the area of mass spectrometry. These advances, both in instrumentation and methodology, are providing researchers with sensitive assays for both identification and quantification of proteins within complex samples. This discussion will highlight some of these methodologies, namely the use of Multiple Reaction Monitoring (MRM) and Protein-AQUA.
Unser Team von Wissenschaftlern verfügt über Erfahrung in allen Forschungsbereichen einschließlich Life Science, Materialwissenschaften, chemischer Synthese, Chromatographie, Analytik und vielen mehr..
Setzen Sie sich mit dem technischen Dienst in Verbindung.