10786357001
Roche
Ribonuklease H (RNase H)
from Escherichia coli H 560 pol A1
Synonym(e):
RNase H
About This Item
Empfohlene Produkte
Biologische Quelle
Escherichia coli ( H 560 pol A1)
Qualitätsniveau
Assay
100%
Form
solution
Spezifische Aktivität
~40000 units/mg protein
Verpackung
pkg of 100 U
Hersteller/Markenname
Roche
Methode(n)
cDNA synthesis: suitable
Farbe
colorless
Optimaler pH-Wert
7.5-9.1
Löslichkeit
water: miscible
Eignung
suitable for molecular biology
NCBI-Hinterlegungsnummer
Anwendung(en)
life science and biopharma
Fremdaktivität
RNase, none detected (up to 10 U with MS- II- RNA)
endonuclease ~10 units, none detected (using lambda-DNA)
nicking activity 10 units, none detected
Versandbedingung
dry ice
Lagertemp.
−20°C (−15°C to −25°C)
Angaben zum Gen
Escherichia coli ... rnhA(946955)
Allgemeine Beschreibung
Quelle: E. coli H560 pol A1
Lagerpuffer: 25 mM Tris-HCl, 50 mM KCl, 1 mM Dithiothreitol, 0,1 mM EDTA, 50 % Glycerin (v/v), pH 8,0 (+4 °C)
Volumenaktivität: 1 x 103 U/mL nach Hillenbrand & Staudenbauer getestet.
Anwendung
- In-vivo-Initiierung der DNA-Synthese mit RNA-Priming
- Beseitigung von mRNA während der Zweitstrang-cDNA-Synthese
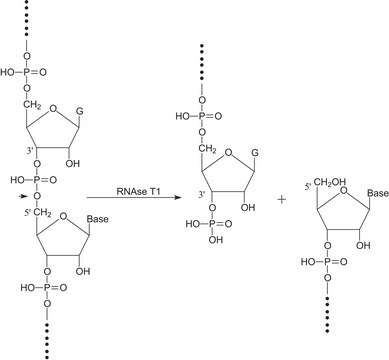
- Stellenspezifische Spaltung von RNA
- Detektion von RNA:DNA-Regionen in doppelsträngiger DNA natürlicher Herkunft
- Entfernung der Poly-(A)-Sequenzen von mRNA bei Vorhandensein von Oligo (dT)
- RNA-Extraktion und quantitative Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR)
Biochem./physiol. Wirkung
Leistungsmerkmale und Vorteile
- Beseitigt potenzielle Quellen von PCR-Fehlern.
- Verbessert die Zugänglichkeit von Primern während einer anschließenden PCR.
Qualität
Einheitendefinition
Volumenaktivität: Ca. 1 U/μL
Angaben zur Herstellung
Lagerung und Haltbarkeit
Sonstige Hinweise
Lagerklassenschlüssel
12 - Non Combustible Liquids
WGK
WGK 1
Flammpunkt (°F)
does not flash
Flammpunkt (°C)
does not flash
Analysenzertifikate (COA)
Suchen Sie nach Analysenzertifikate (COA), indem Sie die Lot-/Chargennummer des Produkts eingeben. Lot- und Chargennummern sind auf dem Produktetikett hinter den Wörtern ‘Lot’ oder ‘Batch’ (Lot oder Charge) zu finden.
Besitzen Sie dieses Produkt bereits?
In der Dokumentenbibliothek finden Sie die Dokumentation zu den Produkten, die Sie kürzlich erworben haben.
Kunden haben sich ebenfalls angesehen
Unser Team von Wissenschaftlern verfügt über Erfahrung in allen Forschungsbereichen einschließlich Life Science, Materialwissenschaften, chemischer Synthese, Chromatographie, Analytik und vielen mehr..
Setzen Sie sich mit dem technischen Dienst in Verbindung.