MISSION™ esiRNAの仕様
カスタム・プレデザインesiRNA
2020年10月下旬/11月初旬から、チュープタイプのesiRNA製品(個別のesiRNA、esiOPENおよびesiSEC)について、溶液ではなく凍結乾燥品として出荷を始めました。なお、プレートタイプのesiRNA製品(esiLibraryおよびesiFLEX)は溶液の状態で引き続き出荷します。次の理由により、チューブタイプの製品(大半のチューブタイプのesiRNA出荷品)に対して、このような変更を行っています。
- 凍結乾燥品は、ドライアイスで凍結せずに常温で出荷できます。このような出荷コストの削減により、製品価格をできるだけ低く抑えることができます。
- 出荷時のドライアイスをなくすことで、包装を削減でき、環境負荷を低減するという私たちのコミットメントにも合致させることができます。esiRNAは、RNAとして室温で3週間以上安定です。esiRNAの納品後は、使用するまで、-20℃で保管してください。製品の技術情報の付録2に再懸濁プロトコルを記載しています。一部のesiRNAは溶液の状態で出荷し、同時に乾燥品も出荷するという移行期間がありますが、できる限り迅速に、すべてのユニットを乾燥フォーマットにシフトさせる予定です。
esiRNA仕様表にすべての注文と見積りのオプションを記載
RNAi研究者は、MISSION™ esiRNA(Eupheria Biotech社のテクノロジーを使用)により、実証済みでコスト効率のよい、シンプルな方法で、タンパク質をコードする遺伝子およびlncRNA(長鎖ノンコーディングRNA)の翻訳後サイレンシングを行うことができます。生物学的に調製されたesiRNAは、すべて同じmRNA配列をターゲットとする不均一なsiRNAプール(天然RNA、修飾なし)で構成されています。 これらのマルチサイレンシングトリガーのオフターゲット効果は、単一の化学合成siRNAと比べて低く、特異性と効力の高い遺伝子ノックダウンをもたらします(図1)。

図1.ターゲットmRNAのノックダウンは、化学合成したsiRNAまたは酵素を使用して合成したsiRNA(esiRNA)により実行可能。A) 化学合成したsiRNAは、ターゲットmRNAに相補的な21 bpの単一のサイレンシングトリガーで構成。トランスフェクション反応液中のsiRNA濃度が高いと、著しいオフターゲット効果をもたらす。B) 対照的に、esiRNAは、ターゲットmRNAの300~600 bp領域をカバーする数百種類のsiRNA(21 bp)のプールで構成。個々のsiRNAはプール中において低濃度であるため、オフターゲット効果を低く抑えつつ、効果的なノックダウンを実行。
製品の利点
- 保証された遺伝子サイレンシング
- 化学合成したsiRNAと比べてオフターゲット効果が低減
- オンターゲット特異性が高いため、効果的な一次スクリーニングツールとして使用可能(図2)
- 手頃な価格のゲノムスケールRNAiスクリーニングツール
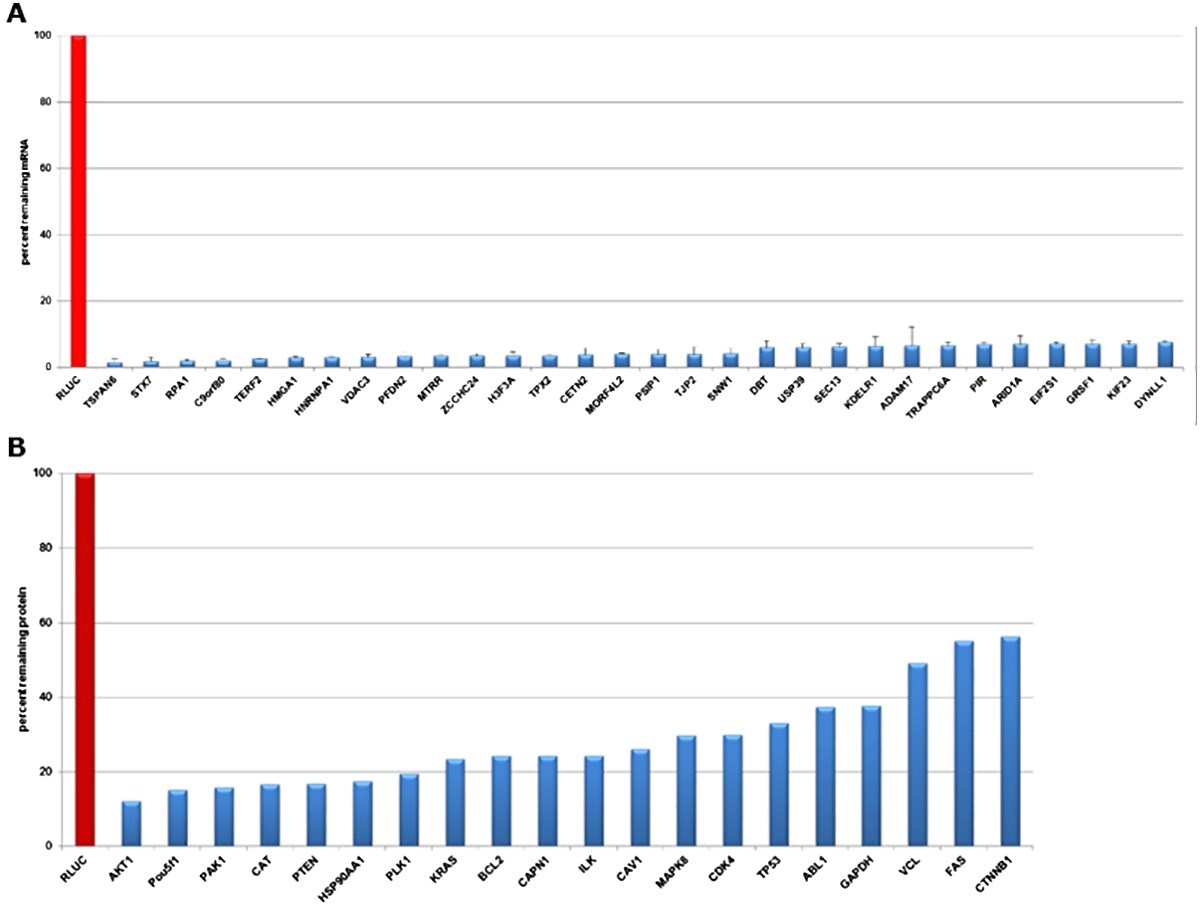
図2.esiRNAをトランスフェクションしたHeLa細胞の遺伝子ノックダウンによる評価。ウミシイタケルシフェラーゼ(ネガティブコントロール)およびさまざまな発現ターゲットを対象としてmRNA濃度とタンパク質濃度について検討した。A) qPCRデータでは、トランスフェクション後24時間でノックダウンが確認された。すべてのトランスフェクションにおいて、mRNA量は90%超で減少した。B) 定量的ウェスタンブロットデータでは、トランスフェクション後72時間で効果的なノックダウンが確認された。タンパク質濃度は45%~90%減少した。
製品の特長
以下は、esiRNA仕様表に示す全製品オプションに適用されます。
- 精製:Q-セファロース分離、イソプロパノール沈殿法およびエタノール洗浄
- 鎖長:二本鎖の平均長21 bpの数百種類のsiRNAからなるプール
- 品質管理:2段階の試験を施
o cDNAクローンのPCR産物を、ゲル電気泳動法とDNAシーケンシングで分析
o ゲル電気泳動法により分解状態を分析 - 安定性:-20℃で保存した場合、2年間安定
「お問い合わせ」のマークがついている製品や一般的な仕様と異なるものをご希望の場合は、customjp.ts@merckgroup.comまでお問い合わせください。
プレデザインesiRNAの製品ページをご覧ください。
esiRNAのコントロール
さまざまなポジティブおよびネガティブコントロールをご用意しています(図3):
- ポジティブコントロール:トランスフェクションと実験条件の最適化を図るための、ヒトEg5 (KIF11)およびマウスEg5 (Kif11) esiRNA、ならびに検証済みの全esiRNA(下表参照)
- ネガティブコントロール:非特異的効果と配列特異的サイレンシングを区別するための、RLUC、FLUCおよびEGFP(EGFPは、EGFPを発現する細胞ではポジティブコントロールとしても使用可能)esiRNA

図3.A) RLUC(ネガティブコントロール)およびB) Eg5/KIF11(ポジティブコントロール)に対するesiRNAをトランスフェクションしたHeLa細胞の表現型解析。RLUCは表現型変化を誘導しないが、Eg5/KIF11は有糸分裂停止(円形の細胞として認められる)を誘導する。
検証済みesiRNA
多数の一般的遺伝子ターゲットにおいて、70%以上のmRNAノックダウンが検証されています(qPCRおよびウェスタンブロットによる検証データ)。多数の実績がある検証済みesiRNAの遺伝子記号別の一覧表をご覧ください。検証済みのesiRNAは、トランスフェクションの最適化やポジティブコントロールに適しています。
lncRNA esiRNA
トランスクリプトーム分析から、最大90%のゲノムがノンコーディングRNAに転写され、いくつかの型の中にIncRNAも含まれることが明らかになっています。lncRNAは、クロマチン修飾、転写因子のコアクチベーション、転写、RNA結合タンパク質との相互作用およびプロモーターの抑制など、さまざまな機能と関連しています。さらに、lncRNAはがんのような疾患にも関与しています。lncRNAがどのように機能するかの詳細については、さらに明らかにする必要があります。esiRNAは、lncRNA機能を研究するための有効なスクリーニングツールです(図4)。

図4.HeLa細胞のlncRNAのノックダウンを検証したqPCRデータ(該当するesiRNAをトランスフェクションした24時間後)。コントロールにはRLUCに対するesiRNAをトランスフェクションし、標準化に使用した。
製品製造
生物学的に調製された(図5)esiRNAは、E. coli RNase IIIエンドヌクレアーゼを用いて長鎖dsRNA(二本鎖RNA)を切断し、製造されます。

図5.単一遺伝子に対するesiRNAの製造概要。A) DEQORを使用して、既知の転写生成物バリアントを網羅し、オフターゲット効果を最小限に抑える、最も効果の高いsiRNAを最大数生成できるような、mRNAターゲット領域を選択。B) そのターゲット領域をcDNAクローンから抽出し、PCRにより増幅(両プライマーを使用してT7プロモーターを導入)。C) PCR産物からRNAポリメラーゼを用いてin vitroで転写。D) アニーリングされた、長鎖の二本鎖RNA(dsRNA)をRNase IIIを用いて分解し、精製によりDNAテンプレート、NTP残留物、不完全分解状態のdsRNAを除去。最終的に、esiRNAは二本鎖の平均長21 bpの数百種類のsiRNAからなるプールとして整えられる。
esiRNAの保証
MISSION™esiRNAは、30 nM以上の濃度でトランスフェクションした場合に、培養細胞中のターゲットmRNAレベルを70%低減させます。esiRNAがターゲット遺伝子を70%ノックダウンしない場合、その遺伝子に対するesiRNAを無料で追加提供します。その遺伝子に対するesiRNAが存在しない場合には、購入金額をお返しします。
この保証には、トランスフェクション効率に関する適切な裏付けデータの提供が必要です。トランスフェクション効率に関する適切な裏付けデータとは、30 nM以上でトランスフェクションした検証済みMISSION™esiRNAのターゲットmRNAレベルと適切なネガティブコントロール(疑似トランスフェクション、RLUC、FLUCまたはeGFP esiRNAなど)を比較して、70%のターゲットmRNAのノックダウンを示すqPCRデータなどを指します。
抗体のばらつきおよびタンパク質の半減期のため、タンパク質ベースの検出方法で得られたデータは認められません。
よくあるお問い合わせ
esiRNAに関する詳細については、よくあるお問い合わせセクションをご覧ください。
チュートリアル動画
esiRNAによる哺乳類細胞のスクリーニングについては、こちらをご覧ください。
さらにサポートが必要な場合は、テクニカルサービス(customjp.ts@merckgroup.com)までご相談ください。
引用文献
単一遺伝子esiRNA
全ゲノム・サブライブラリースクリーニング
lncRNA esiRNA
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?