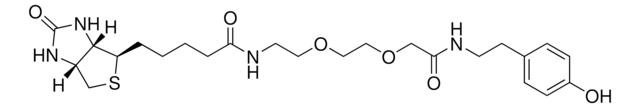
O4387
Oligo(dT)23, verankert
70 μM in H2O
Synonym(e):
Verankerter Oligo(dT)23-Primer
About This Item
Empfohlene Produkte
Biologische Quelle
synthetic (organic)
Form
solution
Verwendung
0.1 mL sufficient for 100 RT-PCR reactions (as described in the Technical Bulletin for Product Codes HSRT100 and HSRT20)
Konzentration
70 μM in H2O
Farbe
colorless
Versandbedingung
wet ice
Lagertemp.
−20°C
Verwandte Kategorien
Allgemeine Beschreibung
Anwendung
Leistungsmerkmale und Vorteile
- Wenn bei der Erzeugung von cDNA-Bibliotheken Sequenzinformationen unvollständig sind oder fehlen oder wenn spezifische Primer nicht geeignet sind, können verankerte Oligo(dT)23-Primer zusammen mit zufälligen Nonameren (Produkt-Nr. R 7647) verwendet werden.
- Diese Primer können bei Aufgaben wie der Erststrang-cDNA-Synthese, der Erzeugung von cDNA-Bibliotheken und verschiedenen anderen Anwendungen als Ersatz für Primer für die reverse Transkription dienen.
- Nach der Transkription zeigen die verankerten Oligo(dT)23-Primer eine verringerte Fähigkeit, bei höheren Temperaturen (bis 65 °C) Priming zu initiieren, sodass sie sich nicht auf die PCR auswirken.
- Verankerte Oligo(dT)23-Primer können bei der Herstellung von cDNA aus poly(A+)-RNA einen Vorteil gegenüber Standard-Oligo(dT)-Primern bieten.
Sonstige Hinweise
Ähnliches Produkt
Lagerklassenschlüssel
10 - Combustible liquids
WGK
WGK 3
Flammpunkt (°F)
Not applicable
Flammpunkt (°C)
Not applicable
Persönliche Schutzausrüstung
Eyeshields, Gloves
Analysenzertifikate (COA)
Suchen Sie nach Analysenzertifikate (COA), indem Sie die Lot-/Chargennummer des Produkts eingeben. Lot- und Chargennummern sind auf dem Produktetikett hinter den Wörtern ‘Lot’ oder ‘Batch’ (Lot oder Charge) zu finden.
Besitzen Sie dieses Produkt bereits?
In der Dokumentenbibliothek finden Sie die Dokumentation zu den Produkten, die Sie kürzlich erworben haben.
Kunden haben sich ebenfalls angesehen
Artikel
One approach to the analysis of gene expression is to measure the concentration of mRNA of a gene. There are several challenges to such analyses, such as the differences in half life between different transcripts, the temporal patterns of transcription and the lack of correlation between mRNA and protein.
Protokolle
The 3’/5’ integrity assay is a potential first step in the identification of RNA degradation. The assay is particularly useful when a large number of samples are to be analyzed or when the degradation is less than that detected by capillary systems but still sufficient to effect qPCR analyses.
Unser Team von Wissenschaftlern verfügt über Erfahrung in allen Forschungsbereichen einschließlich Life Science, Materialwissenschaften, chemischer Synthese, Chromatographie, Analytik und vielen mehr..
Setzen Sie sich mit dem technischen Dienst in Verbindung.