Badanie MSI i IHC
Mike Lacey, M.D.
Medical Director
Testowanie MSI
Historia
Niedobór naprawy niedopasowania
Znaczenie prognostyczne niedoboru Mismatch Repair
w raku jelita grubego
Poradnictwo genetyczne i postępowanie w
dziedzicznym raku jelita grubego niezwiązanym z polipowatością
Rokowanie
Testy MSI
Rak jelita grubego jest powszechnym zjawiskiem wśród mieszkańców większości krajów zachodnich, ustępując jedynie rakowi płuc.1 Pomimo poprawy technik chirurgicznych i schematów chemioterapii, rokowanie w tej chorobie nie poprawiło się znacząco w ciągu ostatnich dziesięcioleci. Obawy te nakazują zwrócenie większej uwagi na etiologię i patogenezę raka jelita grubego.
Chociaż czynniki dietetyczne zostały dobrze zbadane w badaniach etiologicznych2, najbardziej przewidywalny czynnik ryzyka choroby, jej dziedziczna predyspozycja, otrzymał ograniczoną uwagę. Może to wynikać z niedostatecznej wiedzy klinicystów na temat roli czynników genetycznych w etiologii raka.3 Na przykład wielu lekarzy uważa, że rodzinna polipowatość gruczolakowata (FAP) jest jedynym genetycznym czynnikiem ryzyka związanym z etiologią raka jelita grubego. Jednak FAP stanowi około 1% całkowitego obciążenia rakiem jelita grubego, podczas gdy dziedziczne zespoły raka jelita grubego są co najmniej sześć do ośmiu razy częstsze niż FAP.3,4
Hereditary nonpolyposis colorectal carcinoma (HNPCC), lub zespół Lyncha, jest autosomalnym dominującym zespołem stanowiącym od 5 do 10% całkowitego obciążenia rakiem jelita grubego.5 Brak charakterystycznych cech diagnostycznych skłonił do wprowadzenia pewnych kryteriów w celu ustalenia tej diagnozy: (a) histologicznie zweryfikowany rak jelita grubego u co najmniej trzech krewnych (z których jeden jest krewnym pierwszego stopnia pozostałych dwóch), (b) obecność choroby w co najmniej dwóch kolejnych pokoleniach oraz (c) jeden lub więcej przypadków raka jelita grubego zdiagnozowanych przed 50 rokiem życia. Kryteria te, znane jako "Kryteria Amsterdamskie I", zostały zaproponowane w celu identyfikacji rodzin HNPCC z dziedzicznym rakiem jelita grubego.5-7
Typowe cechy HNPCC obejmują rodzinną historię raka jelita grubego (CRC) w stosunkowo młodym wieku, przewagę guzów proksymalnych i tendencję do występowania wielu guzów pierwotnych, synchronicznych lub metachronicznych. Niektóre rodzaje guzów pozakolonowych są również związane z chorobą, takie jak guzy endometrium, jajnika, żołądka, jelita cienkiego, dróg wątrobowo-żółciowych, trzustki, mózgu i dróg moczowych.6,8-10
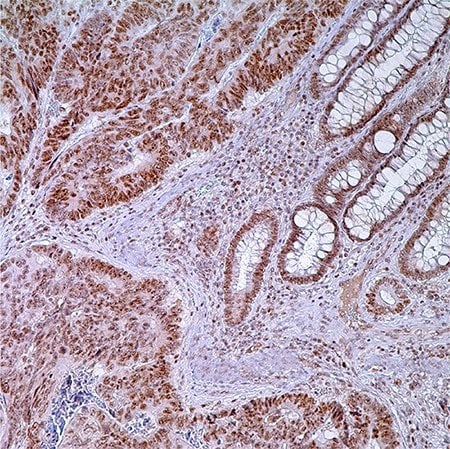
Rysunek 1. PMS2 (EPR3947†) - gruczolakorak okrężnicy. Zwróć uwagę na znakowanie jądrowe komórek gruczolakoraka, normalnych komórek krypt i limfocytów w blaszce właściwej.
Historia
Henry T. Lynch natknął się na chorobę, która ostatecznie nosiła jego imię, konsultując się z pacjentem z silną historią CRC przy braku jawnej polipowatości.29,30 Analiza rodowodowa rodziny tego pacjenta stanowiła podstawę "Czynników dziedzicznych w raku" Lyncha z 1966 roku. Study of 2 large midwestern kindreds", opublikowanej w Arch Intern Med.17 Aktualizacja rodziny Lynch i Krush, opublikowana w 1971 r., zawiera dane dotyczące ponad 650 członków rodziny. Odnotowano wiele istotnych cech zespołu: (1) zwiększoną częstość występowania gruczolakoraków, głównie jelita grubego i endometrium; (2) zwiększone ryzyko wystąpienia wielu nowotworów; (3) dziedziczenie autosomalne dominujące; oraz (4) wczesny początek raka.10 Pomimo wnikliwości Lyncha i Krusha, ich poglądy spotkały się ze znacznym sceptycyzmem ze strony całej społeczności naukowej, ponieważ współczesne myślenie było zdominowane przez pogląd, że czynniki środowiskowe były głównym determinantem raka.29,30

Rysunek 2. Zalecenia dotyczące genetycznych badań przesiewowych w kierunku dziedzicznego raka jelita grubego niezwiązanego z polipowatością (HNPCC).
Pomysły Lyncha powoli zyskiwały na popularności, szczególnie w społeczności międzynarodowej, czego kulminacją było zorganizowanie "International Collaborative Group on Hereditary Non-Polyposis Colorectal Cancer" złożonej z "30 wiodących ekspertów z 8 różnych krajów". Grupa spotkała się w Amsterdamie latem 1990 roku. Głównym wynikiem tego spotkania było sformułowanie szeregu kryteriów klinicznych (obecnie znanych jako kryteria amsterdamskie i potocznie nazywanych zasadą "3-2-1"), które miały służyć jako wspólny punkt wyjścia dla przyszłych badań: (1) co najmniej 3 krewnych z histologicznie potwierdzonym rakiem jelita grubego, z których 1 jest krewnym pierwszego stopnia pozostałych 2; rodzinna polipowatość gruczolakowata powinna być wykluczona; (2) co najmniej 2 kolejne pokolenia zaangażowane; oraz (3) co najmniej 1 z nowotworów zdiagnozowanych przed 50 rokiem życia.19 Kryteria amsterdamskie (AC) zostały zaktualizowane na spotkaniu grupy w 1998 r., uznając znaczenie podzbioru nowotworów pozakomórkowych (tj. endometrium, jelita cienkiego, moczowodu i miedniczki nerkowej). Te zmienione AC-II są przedstawione w Tabeli 1.
Kryteria Amsterdamskie II: Kryteria kliniczne dla rodzin z HNPCC* (Tabela 1)
- ≥3 krewnych z nowotworem związanym z HNPCC**<
- ≥2 kolejne pokolenia dotknięte chorobą
- ≥1 zdiagnozowany przed 50. rokiem życia<
- 1 powinien być krewnym pierwszego stopnia pozostałych 2
- Rodzinna polipowatość gruczolakowata powinna być wykluczona
- Guz powinien być zweryfikowany przez badanie patologiczne
Gastroenterology. 1999;116:1453-1456.2
*Wszystkie kryteria muszą być spełnione.
**Rak jelita grubego, rak endometrium, jelita cienkiego, moczowodu lub miedniczek nerkowych. HNPCC oznacza dziedzicznego raka jelita grubego bez polipowatości.
Molekularne podstawy genetyczne zespołu Lyncha zostały w dużej mierze wyjaśnione w szybkiej serii raportów z lat 1993-1994. Peltomaki i współpracownicy powiązali chorobę (w 2 dużych rasach spełniających pierwotne AC) z locus na chromosomie 2p. Następnie 3 grupy niezależnie zgłosiły osobliwy fenotyp molekularny w CRC charakteryzujący się powszechnymi zmianami długości sekwencji prostych sekwencji powtarzalnych, zjawisko, które grupy różnie nazywały "błędami replikacyjnymi (RER)", "MSI" i "wszechobecnymi mutacjami somatycznymi w prostych sekwencjach powtarzalnych". Aaltonen i wsp.31 znaleźli fenotyp RER-dodatni w 11/14 (79%) "guzach dziedzicznego raka jelita grubego bez polipowatości (HNPCC)" i 6/46 (13%) sporadycznych CRC. Sporadyczne guzy RER+ charakteryzowały się przewagą prawostronną i prawie diploidalnym statusem z przypadkami HNPCC. Thibodeau i wsp.32 stwierdzili pewien stopień MSI w 25/90 (28%) CRC, łącząc to zjawisko z proksymalną lokalizacją i poprawą przeżycia oraz wykazując odwrotny związek z utratą heterozygotyczności. Grupa Ionova zgłosiła wszechobecne mutacje somatyczne w prostych sekwencjach powtarzalnych w 12% CRC, które były stosunkowo częstsze u kobiet i były związane z prawostronną lokalizacją, słabo zróżnicowaną histologią, mniejszą liczbą mutacji KRAS i p53 oraz niższym stadium zaawansowania nowotworu.142 Do końca 1993 r. sklonowano odpowiedni gen na 2p, MSH2, i zidentyfikowano mutacje linii zarodkowej w rodzinach Lynch.33,34 Drugi locus choroby został powiązany z 3p, a na początku 1994 r. odnotowano mutacje zarodkowe MLH1 w rodzinach Lynch.35-37 Następnie wykazano mutacje powodujące chorobę w PMS2 i MSH6.38-40 W ciągu następnych 15 lat wiedza ta została wykorzystana do lepszego zrozumienia częstości występowania choroby i jej fenotypu oraz do zbadania kwestii klinicznych związanych z wykrywaniem i leczeniem. Szczególnie godne uwagi w tym okresie są 2 warsztaty sponsorowane przez National Cancer Institute (NCI) poświęcone wydaniu wytycznych ("wytyczne Bethesda") w celu identyfikacji pacjentów, którzy odnieśliby korzyści z badań klinicznych w kierunku zespołu Lyncha.11,12 Zmienione wytyczne Bethesda (BG) przedstawiono w Tabeli 2.
Naprawa błędów
.Replikacja DNA wiąże się ze skończoną liczbą błędów, w tym z włączeniem błędnie sparowanych zasad (np. G z A) i poślizgiem nici DNA podczas replikacji (co skutkuje tworzeniem pętli insercji/delecji). Niepowodzenie w naprawie tych błędów skutkuje odpowiednio mutacjami punktowymi i przesunięciami ramki. Aparat MMR DNA rozpoznaje błędy, które wymykają się funkcji korekty polimerazy DNA. System ten jest wysoce konserwatywny od bakterii do ludzi, a ponieważ system ten został wcześniej scharakteryzowany w organizmach jednokomórkowych, związek między niestabilnością mikrosatelitarną (MSI) a wadliwą funkcją naprawy niedopasowania (dMMR) w ludzkim raku został po raz pierwszy rozpoznany przez genetyków drobnoustrojów.42,43 Ludzkie geny MMR zostały nazwane na cześć ich prokariotycznych odpowiedników. Na przykład, MSH2 to "MutS homologue 2" ("mut" odnosi się do uogólnionej hipermutowalności odnotowanej w szczepach bakteryjnych z utratą funkcji MutS). Białka MMR funkcjonują jako heterodimery. Kompleks MSH2-MSH6 rozpoznaje nieprawidłowo sparowane zasady i pętle insercyjne/delecyjne. Rekrutuje MLH1-PMS2, który następnie kieruje pozostałą częścią maszynerii MMR.43 MSH2 i MLH1 są dominującymi (obowiązkowymi) składnikami swoich par. W przypadku braku MSH6, MSH2 może łączyć się w pary z MSH3, a w przypadku braku PMS2, MLH1 może łączyć się w pary z PMS1.
Revised Bethesda Guidelines: Badanie kliniczne guzów jelita grubego pod kątem MSI* (Tabela 2)
- Rak jelita grubego zdiagnozowany u pacjenta w wieku 50 lat
- obecność synchronicznego lub metachronicznego CRC lub innych nowotworów związanych z HNPCC, niezależnie od wieku**
- CRC o histologii MSI-H zdiagnozowany u pacjenta <60 lat***
- CRC zdiagnozowany u krewnych pierwszego stopnia Z1 z nowotworem związanym z HNPCC, przy czym jeden z nowotworów został zdiagnozowany w wieku <50 lat
- CRC zdiagnozowany u krewnych pierwszego lub drugiego stopnia Z2 z nowotworami związanymi z HNPCC, niezależnie od wieku
J Natl Cancer Inst. 2004;96:261-268.3
* Spełnienie tylko 1 kryterium niezbędnego do przeprowadzenia badań klinicznych.
** Nowotwory związane z HNPCC zdefiniowane tutaj jako nowotwory jelita grubego, endometrium, żołądka, jajnika, trzustki, moczowodu i miedniczek nerkowych, dróg żółciowych, mózgu (glejak), skóry (gruczolak łojowy i rogowiak) i jelita cienkiego.
***Limfocyty naciekające guz, reakcja podobna do reakcji Crohna, różnicowanie śluzowate/pierścienia pręcikowego lub wzorzec wzrostu rdzeniastego. CRC oznacza raka jelita grubego; HNPCC, dziedziczny rak jelita grubego bez polipowatości; MSI, niestabilność mikrosatelitarna.
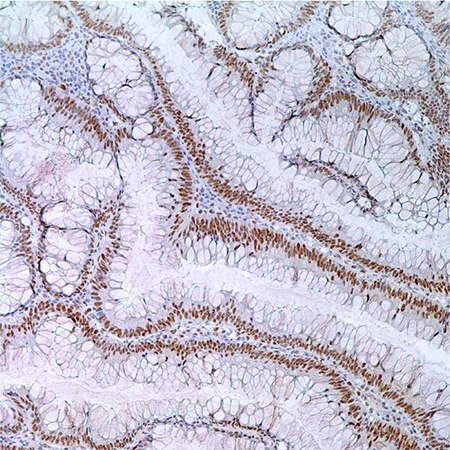
Rysunek 3. MLH1 (G168-728) w gruczolaku okrężnicy u 55-letniego mężczyzny; znakowanie jądrowe komórek gruczolaka.
Białka MSH6 i PMS2 są niestabilne pod nieobecność ich dominującego partnera. Zostało to wykazane za pomocą ilościowej reakcji łańcuchowej odwrotnej transkrypcji-polimerazy i Western blot w serii linii komórkowych dMMR.50 Ponadto większość mutacji Lynch to mutacje nonsensowne lub przesunięcia ramki, prowadzące do skróconych (niestabilnych) białek; mutacje missensowne mogą destabilizować powstałe mRNA lub białko lub zakłócać interakcję białko-białko.51 Te cechy białek MMR mają kliniczne konsekwencje diagnostyczne w odniesieniu do interpretacji immunohistochemii MMR (IHC) ("Badanie kliniczne").
Niestabilność mikrosatelitarna
Mikrosatelity to proste powtarzalne sekwencje DNA rozproszone w całym genomie, składające się z 1 do 6 par zasad, które mogą powtarzać się do 100 razy. Są one z natury hipermutowalne ze względu na ich skłonność do poślizgu nici podczas replikacji DNA. Korekta powstałych pętli insercji/delecji wymaga nienaruszonej funkcji systemu MMR DNA. W przypadku utraty funkcji MMR pętle insercji/delecji nie są naprawiane, co powoduje zmienną ekspansję lub kurczenie się mikrosatelitów. Zjawisko to jest określane jako MSI (wykazane w testach MSI jako typowy wzór prążków na żelu lub pików na analizatorze sekwencji). Geny zawierające proste powtarzające się sekwencje w krytycznych regionach są podatne na to samo zjawisko. Powstałe mutacje z przesunięciem ramki w guzach MSI-H prowadzą do utraty funkcji w dobrze opisanej grupie supresorów nowotworów, w tym TGFbRII, BAX, IGFRII i, co ciekawe, MSH3 i MSH6.52-55 Jako takie, guzy MSI-H zostały opisane jako wykazujące "fenotyp mutatora".
Warsztaty NCI z 1997 roku ustanowiły referencyjny panel mikrosatelitów do badań klinicznych i badawczych oraz zdefiniowały kryteria diagnostyczne dla fenotypów MSI-H, MSI-L i MSS. Podstawowy panel składa się z 2 powtórzeń mononukleotydowych (BAT25, BAT26) i 3 powtórzeń dinukleotydowych (D5S346, D2S123, D17S250). Dostępnych jest również 19 "alternatywnych loci". Analizując 5 loci, MSI-H definiuje się jako niestabilność w 2 loci, a MSI-L jako niestabilność w 1 loci. Biorąc pod uwagę niepewność co do zdolności skończonego panelu do ostatecznego odróżnienia MSI-L od MSS, guzy wykazujące niestabilność w 0 loci są definiowane jako "MSS lub MSI-L". Gdy badanych jest więcej niż 5 loci, MSI-H definiuje się jako niestabilność w >30% do 40% badanych loci, a MSI-L jako niestabilność w <30% do 40% badanych loci.56 Warsztaty NCI z 2002 r. uzupełniły te wytyczne, zalecając testowanie dodatkowych markerów mononukleotydowych w guzach z niestabilnością tylko w loci dinukleotydowych, ponieważ markery mononukleotydowe są bardziej wiarygodne w identyfikacji guzów MSI-H.12
Histologia
Mecklin i wsp.41 zgłosili pierwszą systematyczną ocenę histologiczną raka jelita grubego i gruczolaków u pacjentów z "CFS" (zespół rodzinny raka) w 1986 r., wyprzedzając rozpoznanie MSI i genetycznych podstaw zespołu Lyncha o 7 lat. Zauważyli oni zwiększoną częstość występowania raków śluzowych w przypadkach w porównaniu do sporadycznych kontroli (39% vs. 20%). Słabo zróżnicowane guzy również występowały częściej (24% vs. 12%), ale wynik ten nie osiągnął istotności statystycznej. Chociaż liczba gruczolaków była podobna w obu grupach, gruczolaki pacjentów z CFS zawierały dysplazję wyższego stopnia i bardziej znaczący składnik kosmkowy, co sugeruje, że były bardziej "zaawansowane". Od tego czasu szereg typów nowotworów i cech histologicznych zaczęto wiązać z zespołem Lyncha i MSI-H CRC: rak śluzowy, sygnetowaty i rdzeniasty, limfocyty naciekające guz i okołoguzowe, odpowiedź zapalna "podobna do Crohna", słabe zróżnicowanie, heterogeniczność guza i "pchająca" granica guza.24,26,41,57-63 Greenson i wsp.26,62 dodatkowo podkreślili obecność jakiegokolwiek składnika śluzowego, brak brudnej martwicy i dobrze zróżnicowane guzy jako korelujące ze statusem MSI-H.
Zgodnie z konwencją, gruczolakorak śluzowy jest definiowany jako guz składający się w 50% z mucyny, a gruczolakorak z komórek pierścienia sygnetowego jako guz zawierający w 50% komórki pierścienia sygnetowego. Mucyna w gruczolakorakach śluzowych jest głównie zewnątrzkomórkowa, podczas gdy komórki pierścienia sygnetowego zawierają wyraźną mucynę wewnątrzcytoplazmatyczną, zwykle przesuwającą jądro na jedną stronę komórki.64 Raki rdzeniaste charakteryzują się konstelacją cech cytoarchitektonicznych, w tym dużym rozmiarem komórek, chromatyną pęcherzykową i wyraźnymi jąderkami, wzrostem przypominającym arkusz lub czasami beleczkowatym oraz ogólnym obwodem. Limfocyty naciekające guz (TIL) są szczególnie widoczne w tym typie guza.64
TIL odnoszą się do składnika limfoidalnego ściśle zmieszanego z guzem; wykazano, że składają się one głównie z cytotoksycznych limfocytów T z koekspresją CD3/CD8 [spekuluje się, że ich znaczenie reprezentuje: (1) odpowiedź na obfite tworzenie neoantygenów nowotworowych z powodu "fenotypu mutatora"; oraz (2) możliwą podstawę lepszego rokowania w guzach MSI-H].59 Zgłoszono różne metody (i progi) liczenia TIL, w tym ocenę szkiełek barwionych hematoksyliną i eozyną lub CD3. Jedna z praktycznych metod obejmuje skanowanie slajdu w poszukiwaniu regionu z TIL, zliczanie 5 kolejnych pól 40X i obliczanie średniej liczby TIL / pole o dużej mocy (HPF); badania wykorzystujące tę metodę zdefiniowały wynik pozytywny jako > 2 TIL / HPF.62 Limfocyty okołoguzowe odnoszą się do mankietu limfoidalnego na przedniej krawędzi guza, podczas gdy reakcja "podobna do Crohna" składa się z "wyraźnych" guzowatych agregatów limfoidalnych na naciekającej krawędzi guza, zwykle identyfikowanych na styku mięśniówki właściwej i okołokomórkowej tkanki tłuszczowej. Progi dla pozytywnej reakcji "podobnej do Crohna" obejmowały "2 lub więcej dużych agregatów limfoidalnych w przekroju", "pojedyncze pole 4X z co najmniej 3 guzkowymi agregatami limfocytów", "co najmniej 3 agregaty limfoidalne na sekcję" i "co najmniej 4 agregaty guzkowe w polu o niskiej mocy (4X)."26,61,62,65
Guzy są ogólnie klasyfikowane jako dobrze, umiarkowanie lub słabo zróżnicowane. Kryteria Światowej Organizacji Zdrowia stanowią, że guzy o heterogenicznych wzorcach różnicowania powinny być klasyfikowane w oparciu o składnik o najwyższym stopniu zróżnicowania, z ważnym zastrzeżeniem, że ogniska słabego zróżnicowania na przedniej krawędzi guza (tj. pączkowanie guza, przejście nabłonkowo-mezenchymalne) są niewystarczające do zaklasyfikowania guza jako słabo zróżnicowanego.64 Heterogeniczność guza odnosi się do obecności 2 lub więcej różnych wzorców wzrostu w obrębie guza; Alexander i współpracownicy podają przykład guza o mieszanych cechach śluzowych i rdzeniastych.65 Margines guza "wypychający" lub "ekspansywny" odróżnia się od "naciekającego"; oceny tej najlepiej dokonać przy niskiej mocy.
Testy kliniczne
Testy kliniczne funkcji dMMR w CRC mają 2 potencjalne punkty końcowe: (1) identyfikacja zespołu Lyncha; lub (2) identyfikacja wszystkich MSI-H CRC i jest związana z 2 podstawowymi strategiami badań przesiewowych: (1) wybór pacjentów do badania na podstawie kryteriów klinicznych i/lub patologicznych; lub (2) badanie wszystkich CRC. W tym świetle AC i BG zostały ocenione w kohortach populacyjnych pod kątem ich zdolności do identyfikacji pacjentów z LS. Ogólnie rzecz biorąc, AC-II osiągają czułość rzędu od 40% do 50%, podczas gdy zrewidowane BG działają na poziomie około 90%.13-16,20-23,71 Należy podkreślić, że te cechy testu zostały osiągnięte w wyidealizowanych warunkach, z fińskimi badaczami mającymi dostęp do krajowych rejestrów nowotworów i innymi badaczami opisującymi wykorzystanie "szpitalnych genetyków klinicznych" i "szczegółowych kwestionariuszy". Praktyczne bariery w stosowaniu BG obejmują ich złożoność i kaprysy w dokumentowaniu wiarygodnej historii raka w rodzinie.72
Inne ograniczenia obecnych wytycznych klinicznych obejmują ich zmniejszoną czułość w mniejszych rodzinach i populacjach, w których rutynowa kolonoskopia przesiewowa zmienia naturalną historię choroby. Oczywiście strategie te nie są ukierunkowane na większą populację MSI-H CRC.
Jeśli znaczenie predykcyjne statusu MSI-H (tj. znaczenie terapeutyczne) może zostać ostatecznie udowodnione w prospektywnych, randomizowanych, kontrolowanych badaniach klinicznych, wówczas strategia "testowania wszystkich CRC" dla punktu końcowego MSI-H powinna stać się standardem opieki. Badanie MSI jest uważane za "złoty standard" w celu wykazania statusu MSI-H.
Ponieważ nieukierunkowane badanie mutacji linii zarodkowej pod kątem zespołu Lyncha jest zbyt drogie (około 1000 USD/gen), badanie MSI i MMR IHC zostały ocenione jako testy przesiewowe.73 Każdy z tych testów ma związane z nimi zalety i wady. Badanie MSI obejmuje oparte na PCR badanie panelu markerów mikrosatelitarnych w dopasowanej tkance nowotworowej i normalnej (zazwyczaj sąsiadującej nienowotworowej okrężnicy lub krwi obwodowej). Wymaga mikrodysekcji i usług laboratorium diagnostyki molekularnej. Jego niezdolność do zasugerowania konkretnego genu do analizy mutacji jest wyraźną wadą, a jeśli dalsze testy są ograniczone do przypadków MSI-H, może być również nieco mniej czuły niż IHC w identyfikacji LS z powodu mutacji MSH6 (która może występować jako MSI-L CRC).74
IHC jest łatwo dostępny w większości diagnostycznych laboratoriów patologii anatomicznej. Przeciwciała przeciwko 4 białkom zaangażowanym w LS (poprzez mutację germinalną powiązanego genu) są dostępne w handlu. Nieprawidłowym wynikiem jest całkowity brak (utrata) immunoreaktywności jądrowej dla jednego lub więcej białek w guzie. Wszystkie 4 białka ulegają normalnej ekspresji w tkance nienowotworowej, a zatem zręby, limfocyty i krypty nienowotworowe służą jako krytyczne kontrole wewnętrzne. Wykazano, że etap odzyskiwania antygenu jest ważny; w jednym z badań nieodpowiednie odzyskiwanie antygenu wiązało się ze słabym barwieniem i słabą swoistością dla LS (podczas gdy wysokie barwienie tła wiązało się ze słabą czułością).98 Słabe lub niejednorodne wzory barwienia są czasami spotykane i mogą być związane ze zmiennością utrwalania tkanek lub innymi czynnikami technicznymi. W takich przypadkach pomocne może być powtórzenie badania IHC. Kluczową zaletą stosowania IHC w badaniach przesiewowych Lynch jest jego zdolność do bezpośredniego testowania genów. Utrata funkcji MSH2 (z powodu szkodliwej mutacji) objawia się brakiem ekspresji immunohistochemicznej (IHC) MSH2 i MSH6, podczas gdy utrata funkcji MLH1 (z powodu szkodliwej mutacji lub hipermetylacji promotora) jest wykrywalna jako brak ekspresji immunohistochemicznej MLH1 i PMS2. Izolowany brak białka MSH6 lub PMS2 sugeruje mutację w odpowiednim genie.
Wyniki testów MMR IHC i MSI okazały się w dużej mierze zgodne.15,16,22,23,47,48,76-84 W 1 dużym badaniu, łączne zastosowanie MLH1 i MSH2 IHC osiągnęło czułość 92,3% i swoistość 100% dla identyfikacji guzów MSI-H (1144 przebadanych przypadków, z których 302 były MSI-H).77 Dodanie MSH6 i PMS2 do panelu powinno prowadzić do dalszego zwiększenia czułości, ponieważ mutacje MSH6 i PMS2 są coraz częściej uznawane za etiologiczne w znacznej mniejszości przypadków Lynch.44,45,47,48,79 Potencjalną wadą IHC jest jej niezdolność do wykrywania mutacji missense, które nie destabilizują powstałego mRNA lub białka. Z tego powodu Burgart oszacował maksymalną czułość MMR IHC w wykrywaniu LS na 95%.83 Z naszego doświadczenia wynika, że analiza IHC identyfikuje LS i MSI-H CRC w 90% przypadków; badanie MSI jest podobnie czułe w identyfikacji LS.15,16,21-23,82 W guzach wykazujących brak (utratę) ekspresji białka MLH1, przed przystąpieniem do analizy mutacji MLH1 można zastosować dodatkową warstwę testów: Badanie metylacji promotora MLH1 i/lub analiza mutacji BRAF. Każdy z nich wykorzystuje unikalną historię rozwoju sporadycznego MSI-H CRC, jak omówiono wcześniej. W pierwszym przypadku do określenia statusu metylacji promotora MLH1 stosuje się PCR specyficzny dla metylacji. Metylowane przypadki mają prawdopodobnie charakter sporadyczny, a zatem analiza mutacji linii zarodkowej MLH1 może nie być konieczna. Istnieje jedno ważne zastrzeżenie. Chociaż "drugim trafieniem" w większości LS jest utrata heterozygotyczności lub mutacja somatyczna, w rzadkich przypadkach opisano hipermetylację promotora MLH1.27,28,68,85,86 Jest to również droższe niż analiza BRAF, a także trudne technicznie.
Somatyczne mutacje aktywujące w BRAF, składniku szlaku kinazy Ras/Raf/MAP, występują w wielu różnych nowotworach ludzkich, w tym w 15% CRC.143
Są one często spotykane w gruczolakach ząbkowanych i sporadycznych MSI-H CRC (około 75%) i są uważane za dowód ich ewolucyjnego powiązania.28,66-69,70 Za większość mutacji odpowiada mutacja punktowa skutkująca substytucją kwasu glutaminowego na walinę w kodonie 600 (V600E); w jednym badaniu CRC, 60/63 (95%) mutacji było V600E.66 Tak więc, obecność BRAF V600E w CRC wspiera interpretację, że przypadek jest sporadyczny, a analiza mutacji linii zarodkowej MLH1 zazwyczaj nie jest przeprowadzana. Badanie pojedynczej mutacji punktowej (zamiast sekwencjonowania całego genu) sprawia, że analiza mutacji BRAF jest atrakcyjna ekonomicznie (100 USD).73
Okazjonalnie można zostać poproszonym o ocenę gruczolaków u pacjentów z "możliwym zespołem Lyncha", w otoczeniu silnego wywiadu rodzinnego, w którym tkanka nowotworowa nie jest dostępna do analizy. MMR IHC jest dość czułe i wysoce specyficzne w tym przypadku.25,87,88 Halvarsson i wsp.87 zgłosili utratę ekspresji białka MMR w 23/35 (66%) gruczolaków od 26 pacjentów z "HNPCC" (z których 88% miało potwierdzone mutacje germinalne MMR). Brak barwienia był szczególnie częsty w gruczolakach o wielkości 5 mm (88%). Dziewiętnaście gruczolaków wykazujących utratę białka MMR zawierało jedynie typową dysplazję niskiego stopnia. Wzór nieobecnego barwienia przewidywał zaangażowany gen w każdym przypadku.
Nieobecne barwienie w tej sytuacji klinicznej należy odróżnić od tego obserwowanego w bezszypułkowych ząbkowanych gruczolakach z nałożoną dysplazją cytologiczną (SSAD) (ogólnie akceptowany prekursor sporadycznego MSI-H CRC).89 Architektura zmiany i wzór utraty są ważnymi czynnikami. SSAD są ząbkowane (gruczolaki w LS prawie zawsze nie są), mogą wykazywać połączoną utratę MLH1 i PMS2 (nie MSH2 i / lub MSH6), a utrata ekspresji jest ogólnie uważana za późne zdarzenie, często odpowiadające inwazji (podczas gdy utrata ekspresji jest często obserwowana w gruczolakach LS tylko z dysplazją niskiego stopnia).
Wykazanie szkodliwej mutacji germinalnej jest "złotym standardem" w diagnostyce zespołu Lyncha. Pozwala na stosunkowo niedrogą, ukierunkowaną analizę linii germinalnej u członków rodziny. Należy jednak wziąć pod uwagę kilka kwestii. Ogólnie rzecz biorąc, analiza mutacji jest przeprowadzana na podstawie próbki krwi obwodowej, w tym analizy sekwencji eksonów i granic intron-ekson zaangażowanego genu. Większość zgłaszanych mutacji patogennych to mutacje nonsensowne lub z przesunięciem ramki, powodujące skrócenie białka; mutacje missensowne są trudniejsze do sklasyfikowania.51 Duże delecje mogą stanowić do 20% mutacji patogennych i nie są one wykrywane podczas rutynowego sekwencjonowania. Wielokrotna amplifikacja sondy zależna od ligacji (MLPA) zapewnia ilościową miarę dawki eksonu i jest uważana za metodę z wyboru do wykrywania dużych delecji.90-93 Przy setkach opisanych mutacji w LS, analiza mutacji jest złożona, a czułość testów, choć stale się poprawia, jest mniejsza niż 100%. W przypadku przekonującego wywiadu rodzinnego, niezdolność do udokumentowania jednoznacznie patogennej mutacji linii zarodkowej nie powinna wykluczać pacjenta z odpowiedniego dla LS nadzoru. Baza danych mutacji zespołu Lyncha jest prowadzona przez International Society for Gastrointestinal Hereditary Tumors (dawniej International Collaborative Group on Hereditary Non Polyposis Colorectal Cancer).94
.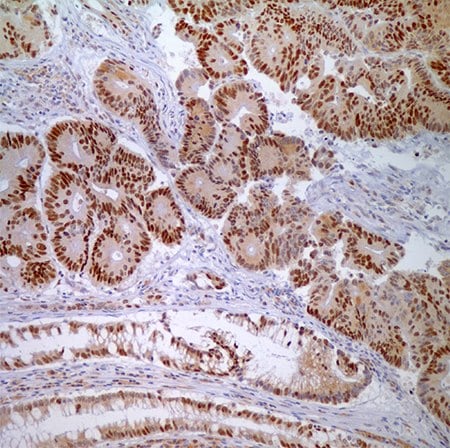
Rysunek 4. MSH6 (44) - znakowanie jądrowe komórek gruczolakoraka i prawidłowych komórek nabłonka krypty.
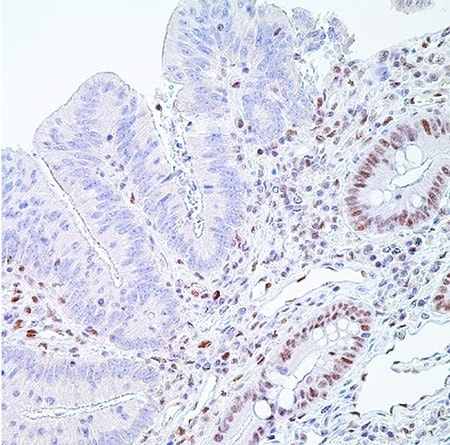
Rysunek 5. PMS2 (EPR3947†) - Zwróć uwagę na normalne znakowanie jądrowe komórek nabłonka krypty (i rozproszonych limfocytów w blaszce właściwej) po prawej stronie i brak znakowania jądrowego w komórkach gruczolakoraka po lewej stronie, co oznacza pozytywny wynik testu (MSI-H).
Znaczenie prognostyczne niedoboru naprawy niedopasowania w raku jelita grubego
Wybieralna korzyść przyznana komórkom nowotworowym przez nabycie niedoboru MMR jest sporna i może, częściowo, odnosić się do zmian w podatności na apoptozę.95,96,97,98 Zauważono nawet przy początkowym opisie MSI, że pacjenci z guzami MSI-H wydawali się mieć lepsze wskaźniki przeżycia niż ci z guzami MSS.21 Od tego czasu przeprowadzono wiele badań dotyczących prognostycznego znaczenia MSI-H w CRC, przy czym większość z nich zgadzała się z początkowymi ustaleniami,99-102 szczególnie u młodych pacjentów, a kilka badań nie zidentyfikowało statusu MSI jako niezależnego czynnika prognostycznego.103,104 Pozornym wsparciem wartości prognostycznej statusu MSI jest wysoka częstość występowania aktywowanych śródnabłonkowych cytotoksycznych limfocytów T i zwiększona apoptoza komórek nowotworowych w guzach MSI-H.99,101 Ta pierwsza cecha może być przypisywana wrodzonej zdolności guzów MSI-H do wytwarzania nowych immunogennych epitopów (jeśli loci B2M i HLA klasy I są nienaruszone i te nowe antygeny mogą być prezentowane), a to może wyjaśniać, dlaczego niektórzy pacjenci z guzami MSI-H mają szczególnie korzystny wynik kliniczny po skutecznych przeciwnowotworowych odpowiedziach immunologicznych.
Znaczenie predykcyjne MSI-H zostało również ocenione w odniesieniu do wyboru pacjentów z CRC, którzy mają otrzymać chemioterapię adiuwantową. Główną wykazaną korelacją jest rozpoznawanie uszkodzeń DNA wywołanych 5-fluorouracylem (5FU) przez nienaruszony system MMR105-107 w wyniku czego komórki z niedoborem MMR są bardziej oporne na 5-FU niż komórki z niedoborem MMR. Co ciekawe, gdy niedobór MMR był spowodowany hipermetylacją MLH1, komórki odzyskiwały wrażliwość na 5-FU po demetylacji MLH1.108 Jednak wyraźna rola MSI-H jako predyktora odpowiedzi na chemioterapię jest nadal kontrowersyjna.109-111
Chociaż testy MSI można stosunkowo łatwo przeprowadzić w laboratoriach diagnostyki molekularnej, istnieją wątpliwości co do klinicznej użyteczności MSI-H jako markera prognostycznego w sporadycznym CRC.112,113 Ważnym czynnikiem wpływającym na brak osiągnięcia konsensusu jest zmienność rodzaju i liczby markerów mikrosatelitarnych, które zostały ocenione w różnych badaniach. Duża metaanaliza 114 z udziałem 7000 pacjentów wykazała, że istnieje wyraźna korelacja między guzami MSI-H a poprawą przeżycia całkowitego. Jednak badanie to zostało przeprowadzone retrospektywnie, a zatem może być podatne na różne czynniki zakłócające. Możliwe jest również, że różnice w podstawowych szlakach molekularnych LS i sporadycznych guzów MSI-H, jak pokazują dane dotyczące częstotliwości mutacji w Tabeli 1, również odgrywają rolę, a zatem mogą istnieć istotne różnice między LS i sporadycznymi guzami MSI-H, które są zaciemnione przez połączenie ich danych.
Genetic Counseling and Management of Hereditary Nonpolyposis Colon Cancer
Przy określaniu odpowiedniego czasu na wykonanie badań genetycznych i rozpoczęcie programu nadzoru należy wziąć pod uwagę wiek zachorowania i historię naturalną raka jelita grubego. Początek raka jelita grubego w zespole Lyncha wydaje się występować 15 do 20 lat wcześniej niż w populacji ogólnej.10 W serii Mecklina w Finlandii119 średni wiek wystąpienia raka jelita grubego wynosił 41 lat, z zakresem wieku od 19 do 83 lat; penetrację genu oszacowano na 89%. W serii Lyncha w Nebrasce średni wiek w momencie diagnozy wynosił 44,4 lat.117,120 Większość nowotworów występuje przed 60 rokiem życia, ze szczytem w przedziale wiekowym 40-50 lat. Dlatego celem badań genetycznych i programów nadzoru powinna być grupa wiekowa 25-60 lat.8,117 Większość pacjentów z HNPCC ma mutację w jednym z dwóch genów naprawy niedopasowania DNA, hMSH2 lub hMLH1. Ponad 90% pacjentów z CRC z hMSH2 lub hMLH1 wykazuje wysoką częstotliwość MSI (MSI-H)121,122; wiele z tych guzów ma błędy replikacji.144 Wstępne badanie linii zarodkowej jest dopuszczalne u osób z wysokim ryzykiem nosicielstwa mutacji, takich jak pacjenci spełniający kryteria Amsterdam I lub pierwsze trzy kryteria wytycznych Bethesda.123 Każdy pacjent z CRC z osobistym lub rodzinnym wywiadem dotyczącym innego CRC lub raka endometrium, lub u którego zdiagnozowano raka przed 50 rokiem życia, niezależnie od wywiadu osobistego lub rodzinnego, spełnia zmodyfikowane wytyczne.
W dużych badaniach12,29 porównujących efektywność kosztową kilku strategii badań genetycznych u pacjentów z HNPCC, wykazano, że badanie linii germinalnej u probantów z CRC, którzy spełniają kryteria amsterdamskie, wykrywa najmniej nosicieli genów i ma niski koszt, podczas gdy badanie MSI guza u wszystkich pacjentów z CRC ma najwyższy koszt i wykrywa najwięcej nosicieli genów. Wykazano również, że badanie linii zarodkowej hMSH2/ hMLH1 u pacjentów spełniających kryteria amsterdamskie i analiza MSI guza u pozostałych pacjentów, którzy spełniają zmodyfikowane kryteria, z późniejszym badaniem linii zarodkowej u osób z guzem MSI-H, jest najlepszą strategią pod względem efektywności kosztowej.122 Jeśli pacjent spełnia kryteria amsterdamskie I lub podejrzewa się, że należy do rodziny HNPCC, pierwszym badaniem przesiewowym powinna być immunohistochemia w celu wykrycia białek hMLH1 i hMSH2.123
Immunohistochemia jest wykonywana przy użyciu bloków tkankowych zatopionych w parafinie, które są barwione hematoksyliną Mayera. Białka hMLH1 i hMSH2 wybarwiają się pozytywnie w jądrach komórkowych, gdy ulegają ekspresji. Analiza mikrosatelitarna jest zwykle wykonywana za pomocą analizy DNA PCR. Ocenianych jest kilka loci, takich jak BAT26, TGF-RII, D2S119, D3S1612, D5S404, D17S261, BAT25, D2S123, D5S346 i D17S250. Nici DNA guza i normalne DNA są porównywane w sąsiednich pasach. Guzy są uważane za MSI-H (wysoka niestabilność), jeśli dwa lub więcej markerów MS wykazuje niestabilność, MSI-L (niska niestabilność), jeśli jeden marker wykazuje dodatnią niestabilność i MSS (stabilny), jeśli żaden marker nie wykazuje niestabilności.124 Jeśli immunohistochemia jest pozytywna, należy wykonać bezpośrednie sekwencjonowanie genomowe ekson po eksonie w celu wykrycia mutacji hMSH2 lub hMLH1. Dwie z najczęściej występujących mutacji to mutacja założycielska 1, delecja genomowa MLH1 obejmująca ekson 16 oraz mutacja założycielska 2, mutacja miejsca splicingu eksonu 6 MLH1 IVS5-1G "A w 454-1.125
Jeśli wynik testu jest negatywny, należy przeprowadzić analizę MS. Jeśli ta ostatnia jest negatywna, nie są potrzebne dalsze badania. Jeśli wynik jest pozytywny, należy przeprowadzić analizę mutacji MLH1 i MSH2, którą wykonuje się poprzez sekwencjonowanie kodujących eksonów, w tym flankujących regionów intronowych i regionu promotora124-126 (Rysunek 2). Żadna z tych trzech metod nie może być stosowana indywidualnie do diagnostyki genetycznej HNPCC. Należy stosować kombinację tych metod i wywiadu rodzinnego. Analiza Southern blot powinna być również stosowana jako test uzupełniający w celu ujawnienia poważnych rearanżacji genomu. W przypadku zidentyfikowania mutacji germinalnej u krewnych dotkniętych chorobą w rodzinie HNPCC, ryzyko zachorowania na raka w ciągu całego życia wynosi 80-85%.120 Proces oceny molekularnej obejmuje standardowe i alternatywne techniki wykrywania mutacji (Tabela 3).
Pacjenci powinni zostać poinstruowani o historii naturalnej HNPCC, z analizą zalet dostępnych strategii nadzoru i zarządzania.6 Pacjenci muszą zdawać sobie sprawę, że pozytywny wynik może wywołać strach i niepokój w rodzinie. Niektórzy członkowie rodzin z HNPCC mogą już cierpieć z powodu nierozwiązanego żalu z powodu wielu zgonów z powodu raka w ich rodzinach. Osoby te mogą być narażone na niekorzystną reakcję psychologiczną i mogą skorzystać z konsultacji psychiatrycznej.120
Wszystkie te osoby z grupy ryzyka powinny zostać poinformowane, że wiek zachorowania na raka w przypadku HNPCC jest zwykle wczesny, ale może być zmienny; dlatego nadzór powinien być kontynuowany przez całe życie. Ponieważ ewolucja gruczolaka jelita grubego do raka jest przyspieszona w HNPCC,115,121 pacjenci powinni być poddawani ocenie kolonoskopowej co roku, aby zmniejszyć ryzyko rozwoju bardziej zaawansowanego stadium raka.119
Proces oceny molekularnej (Tabela 3)
Standardowe techniki wykrywania mutacji
- Polimorfizm konformacyjny pojedynczej nici
- Analiza elektroforezy w gradiencie denaturującym
- Sekwencjonowanie DNA
Alternatywne techniki wykrywania mutacji
- Monoalleliczna analiza ekspresji
- analiza Southern
- ilościowa reakcja łańcuchowa polimerazy
Poradnictwo genetyczne dla członków rodzin wysokiego ryzyka HNPCC powinno rozpocząć się w połowie lat siedemdziesiątych. W rodzinach z tendencją do wczesnego średniego wieku zachorowania na raka, nadzór powinien rozpocząć się w wieku 25 lat. Ze względu na przewagę zmian prawostronnych kolonoskopia całego jelita grubego jest podstawą nadzoru nad HNPCC.7,116 W doświadczonych rękach do jelita ślepego można dotrzeć w prawie 95% przypadków, podczas gdy wskaźnik perforacji nie przekracza 0,2%.10 Kolonoskopia oferuje również możliwość pobierania próbek histologicznych i usuwania polipów. Nadzór jest prowadzony do 60. roku życia, a jeśli do tego czasu nie wystąpi fenotypowa ekspresja zespołu, pacjenci są obserwowani zgodnie z zaleceniami American Cancer Society dotyczącymi ryzyka raka jelita grubego w populacji ogólnej. Endoskopowe wyniki ciężkiej dysplazji błony śluzowej lub płaskich gruczolaków wymagają powtórzenia kolonoskopii po 6 miesiącach. Występuje zwiększona częstość synchronicznych i metachronicznych polipów gruczolakowatych, które stanowią zmiany przednowotworowe w tych zespołach Lyncha.127 W rodzinach z historią raka w miejscach pozakolonicznych nadzór obejmuje wymaz cytologiczny, przezpochwowe USG jajników i biopsję aspiracyjną endometrium co 1 do 2 lat, począwszy od 30 roku życia. W przypadku nowotworów układu moczowego badania przesiewowe powinny być wykonywane co 1 do 2 lat z analizą moczu, USG, cystoskopią i cytologią moczu. W przypadku nowotworów żołądka i dróg żółciowych przełyk, badania czynności wątroby i przezbrzuszne USG wątroby i dróg żółciowych powinny być wykonywane co 1 do 2 lat, począwszy od 30 roku życia.5
W przypadku nowo zdiagnozowanego raka jelita grubego u znanego nosiciela genu lub u osoby wysokiego ryzyka na podstawie wywiadu rodzinnego, subtotalna kolektomia z zespoleniem krętniczo-odbytniczym jest opcją z wyboru ze względu na wysokie ryzyko zmian metachronicznych.5,120,128,129 Przy ryzyku raka jelita grubego zbliżającym się do 80-85% przyjęto profilaktyczną kolektomię.77 Należy wprowadzić coroczne badanie endoskopowe pozostałej części odbytnicy. Ryzyko rozwoju raka odbytnicy po kolektomii brzusznej szacuje się na 12% po 12 latach.131 Alternatywnym podejściem może być proktokolektomia, szczególnie w przypadku nosicieli genu hMSH2, u których ryzyko raka odbytnicy jest wyższe niż u nosicieli hMLH1. Resekcja segmentowa jest sugerowana u starszych pacjentów131 lub u pacjentów z chorobami współistniejącymi, których oczekiwana długość życia jest już ograniczona przez istniejącą chorobę.132 W zespole Lyncha II u kobiet preferowaną metodą nadzoru jest przezpochwowe badanie ultrasonograficzne. Jest to nieinwazyjne, łatwo dostępne badanie, które umożliwia pełne zbadanie jamy macicy. Inne metody nadzoru obejmują pobieranie próbek cytologicznych endometrium i biopsję endometrium, gdy jest to wskazane. U kobiet z zespołem Lyncha II, które są podatne na rozwój raka endometrium i ukończyły rodzinę lub są po menopauzie, należy rozważyć całkowitą histerektomię brzuszną z obustronną salpingooforektomią.5,120,127,132,133
Lekarze powinni poświęcić czas na poinformowanie pacjentek o historii naturalnej tego dziedzicznego zespołu nowotworowego, omówienie ich obaw i lęków związanych z rakiem oraz poinformowanie ich o zaletach i wadach testów genetycznych.
Rokowanie
Ogólne doświadczenia kilku ośrodków z długoterminową obserwacją pacjentów z HNPCC leczonych z powodu CRC są zachęcające. W kilku badaniach odnotowano poprawę wskaźnika 5-letniego przeżycia wśród pacjentów z HNPCC z rakiem jelita grubego.118,119,134,135 W 1996 roku Sankila i wsp. przebadali 175 pacjentów z HNPCC i porównali ich pod względem przeżycia z populacją 14 086 pacjentów ze sporadycznym rakiem jelita grubego.136 Rozpoznanie zespołu opierało się na wykryciu mutacji linii zarodkowej w genie naprawy niedopasowania. Wskaźnik 5-letniego przeżycia dla pacjentów z HNPCC wynosił 65%, w porównaniu do 44% dla przypadków sporadycznych. Rok później Watson i wsp.137 dostarczyli dodatkowych danych na temat rokowania u pacjentów z HNPCC. Przebadali oni retrospektywnie kohortę pacjentów z HNPCC i porównali ich z populacją pacjentów ze sporadycznym rakiem jelita grubego. Stwierdzili oni, że w ciągu pierwszych 10 lat po diagnozie śmiertelność w przypadkach HNPCC była prawie o dwie trzecie wyższa niż w przypadku raka sporadycznego. Niedawno rejestr nowotworów Surveillance, Epidemiology, and End Results (SEER) opublikował podobne wyniki; przeżycie skorygowane o wiek było wyższe u nosicieli HNPCC z rakiem jelita grubego niż u pacjentów ze sporadycznym rakiem jelita grubego.138,139 Oczekiwana długość życia była o jedną trzecią wyższa u pacjentów z HNPCC i rakiem we wczesnym stadium niż w sporadycznych przypadkach; jednak oczekiwana długość życia zmniejszała się wraz z bardziej zaawansowanymi stadiami w momencie diagnozy.140
Biologiczne podstawy tych ustaleń nie zostały jeszcze wyjaśnione. Zaproponowano, że mniej agresywne zachowanie nowotworów HNPCC może być paradoksalnym efektem niestabilności genomowej.141 W ten sposób podstawowe funkcje komórek złośliwych, a zwłaszcza ich potencjał przerzutowy, mogą być hamowane przez bardzo znaczące obciążenie mutacyjne. Ponadto interakcje gen-gen i gen-środowisko mogą odgrywać ważną rolę w historii naturalnej HNPCC. Systematyczny nadzór i indywidualnie zaprojektowane leczenie pacjentów dotkniętych chorobą może pomóc wykryć nowotwory na wcześniejszym etapie, a następnie jeszcze bardziej poprawić rokowanie choroby.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?