GenElute™-E 정제 방법이 DNA 양 측정의 정확도와 다운스트림 효소 공정에 미치는 영향
이러한 결합-세척-용리 방법은 번거로우며, 여러 세척 및 스핀 단계를 요구합니다. 반복되는 스핀 단계는 상당한 DNA 전단을 초래할 수 있습니다. 또한 카오트로픽(chaotropic) 염 및 기타 오염물질은 용리된 DNA 또는 RNA 내로 쉽게 이동할 수 있으며, PCR 같은 다운스트림 효소 과정뿐만 아니라 최종 순도 및 양 측정에 영향을 미칩니다.
당사는 고염 결합 및 에탄올 세척 단계의 필요성을 없애는 새로운 단일 스핀 핵산 정제 시스템을 평가하였습니다. GenElute™-E DNA 및 RNA 정제 키트는 크기 배제 기반 네거티브 크로마토그래피를 이용하여 세포, 조직, 혈액 및 기타 시료에서 커다란 DNA 및 RNA 핵산 분자를 더 작은 단백질, 지질, 이온 성분으로부터 분리합니다(그림 1-2).

그림 1.네거티브 크로마토그래피(크기 배제) 기술을 이용한 핵산 정제.

그림 2.마우스 혈액 시료에서 GenElute™-E 단일 스핀 혈액 DNA 고수율 키트를 사용한 정제된 DNA의 용리와 단백질, 지질 및 기타 분자의 체류. A) 정제된 유전체 DNA의 흡광도 스펙트럼 (붉은 선). B) 정제된 DNA 스펙트럼 (붉은 선) 위에 중첩된 GenElute™-E 스핀 컬럼 잔류액의 흡광도 스펙트럼 (검은 선). 잔류액 스펙트럼은 단백질 및 기타 오염물질을 약 280nm 또는 ≤240nm에서의 광학밀도(OD)로 보여줍니다. y축의 스케일 변화에 주목하십시오.
저희는 핵산 정제 방법이 측정에 방해되는 불순물 및 다운스트림 효소 과정에 미치는 영향을 평가했습니다. 이러한 비교 연구를 위해, GenElute™-E 단일 스핀 정제 기술 또는 모 공급업체의 전형적인 실리카 기반 결합-세척-용리 스핀 추출 기술을 사용하여 유전체 DNA가 추출되었습니다.
- UV 분광광도법(A260nm/280nm 및 A260nm/230nm에 의해 측정된 광학밀도비)
- 겔 전기영동
- 정량적 PCR(qPCR)
결과는 GenElute™-E 단일 스핀 DNA 및 RNA 정제 시스템이 통용되는 실리카 스핀 추출 기술보다 더 높은 순도를 제공하며, PCR 같은 다운스트림 효소 과정에서 더 정확하게 계량하고 오염물질로 인한 방해를 감소시킨다는 것을 나타냅니다.
핵산 추출 중 유입되는 불순물 평가
자외선 (UV) 분광분석에 의한 양 측정 방해
단백질, 절편화된 핵산, ssDNA, RNA(DNA를 측정하는 경우), 프라이머, 추출 및 정제 프로토콜에서 유래한 카오트로픽(chaotropic) 염 같은 흔한 생물학적 오염물질 때문에 핵산 농도를 과대 평가할 수 있습니다. 이러한 불순물 중 일부는 자외선(UV) 분광광도법으로 측정될 수 있습니다. 이 방법에서, UV 광원은 정제된 DNA 또는 RNA 시료를 투과합니다. 다양한 파장에서 시료의 흡광도가 측정됩니다.
260nm에서의 흡광도(A260)는 핵산을 측정하기 위해 사용되었습니다. 280nm에서의 흡광도(A280)는 시료 내 오염 단백질을 측정하기 위해 사용되었습니다. A260/A280 비율은 단백질 오염물질에 대한 시료 순도 평가에 사용될 수 있습니다. 고순도 DNA는 A260/A280 비율 ≥ 1.8이 될 것입니다. 낮은 비율은 최종 추출 내 단백질 오염을 나타냅니다.
230nm에서의 흡광도(A230)는 카오트로픽(chaotropic) 염 같은 화학적 오염물질의 존재를 확인하기 위해 사용될 수 있습니다. A260/A230 비율은 PCR 같은 효소 과정에서 화학적 간섭을 최소화하기 위해 이상적으로 2.0보다 더 커야 합니다. 고순도 시료가 요구될 때 이 비율을 평가하는 것이 대단히 중요합니다.
혈액 시료를 사용하여 GenElute™-E 단일 스핀 DNA 및 RNA 정제 시스템에 대해 흡광도 스펙트럼, A260/280 비율, A260/230 비율이 평가되었습니다. 결과는 경쟁 제품을 사용하여 동일한 시료에 대해 실리카 스핀 추출 정제로 얻은 결과와 비교되었습니다(그림 3 및 표 1). 데이터는 GenElute™-E 정제 시스템이 실리카 결합-세척-용리 추출법에 비해 최종 시료 조제에서 생물학적 및 화학적 오염물질이 더 적으면서도 유전체 DNA를 더 높은 순도로 제공한다는 것을 시사합니다.

그림 3.혈액에서 추출된 유전체 DNA의 UV 흡수 스펙트럼. A) 모 공급업체의 실리카 기반 DNA 정제 스핀 추출 키트 또는 B) GenElute™-E 단일 스핀 네거티브 크로마토그래피 DNA 정제 키트. 획득된 분획물의 10배 농도에서 측정한 그래프. 실리카 용리 분획에서만 불순물이 검출됩니다. 검은 선은 지시된 방법을 사용하여 정제한 샘플의 스펙트럼을 보여주며 어떠한 공정 오염 물질의 영향도 없음을 나타냅니다 빨간 선은 공정 오염물질 없이 정제된 샘플의 스펙트럼으로, 베이스라인 컨트롤로 사용됩니다. 주황색 선은 정제 방법에 대한 스펙트럼으로 실제 시료는 사용하지 않았으며, 분광광도법 판독에 대한 공정 오염의 영향을 보여줍니다.
겔 전기영동법으로 평가한 전단된 DNA 및 저분자량 핵산 불순물
겔 전기영동에 의한 크기 분리는 시료 순도에 대해 시각적이고 정량적인 평가를 제공합니다. 더 커다란 유전체 DNA는 더 작은 DNA 및 RNA 절편보다 더 느리게 이동합니다. 결과 밴드는 형광 염료를 이용하여 관찰 및 정량할 수 있습니다.
실리카 기반 스핀 추출 키트(모 공급업체) 또는 크기 배제에 의한 네거티브 크로마토그래피(GenElute™-E 단일 스핀 정제 키트)를 사용하여 마우스 간 조직에서 추출한 정제된 DNA는 아가로스 겔에서 분리되었습니다. 핵산 염색에 SYBR green이 사용되었습니다. 결과를 정규화하기 위해, 정제된 각 시료로부터 동일한 질량의 DNA를 겔의 각 레인에 로딩하였습니다. 트랜스일루미네이터에서 아가로스 겔을 시각화한 결과, 실리카 기반 정제 방법에 비해 네거티브 크로마토그래피(크기 배제)를 사용할 때 유전체 DNA가 더 어둡고 단일한 밴드로 얻어진다는 것을 보여주었습니다(그림 4).
실리카 멤브레인 스핀 컬럼에서 용리된 시료는 겔의 하단에서 더 희미한 밴드를 보여주었으며, 최종 추출에서 오염 small RNA 또는 전단된 DNA 분자와 일치합니다. 겔 로딩이 시료에서 DNA에 대해 정규화되었으므로(260nm에서의 흡광도로 결정됨), 이러한 데이터는 유전체 DNA 수율이 이 방법에 의해 과대 평가된다는 것을 시사하며, 이는 더 작은 오염 핵산 분자가 흡광도 측정에도 영향을 미치기 때문입니다. GenElute™-E 네거티브 크로마토그래피(크기 배제) 방법을 사용하여 획득된 더 높은 강도의 DNA 단일 밴드는 이러한 조직 시료에서 유전체 DNA 순도가 훨씬 더 높다는 것을 나타냅니다. 결과는 형광측정법을 사용하여 정량화되었습니다(그림 5).
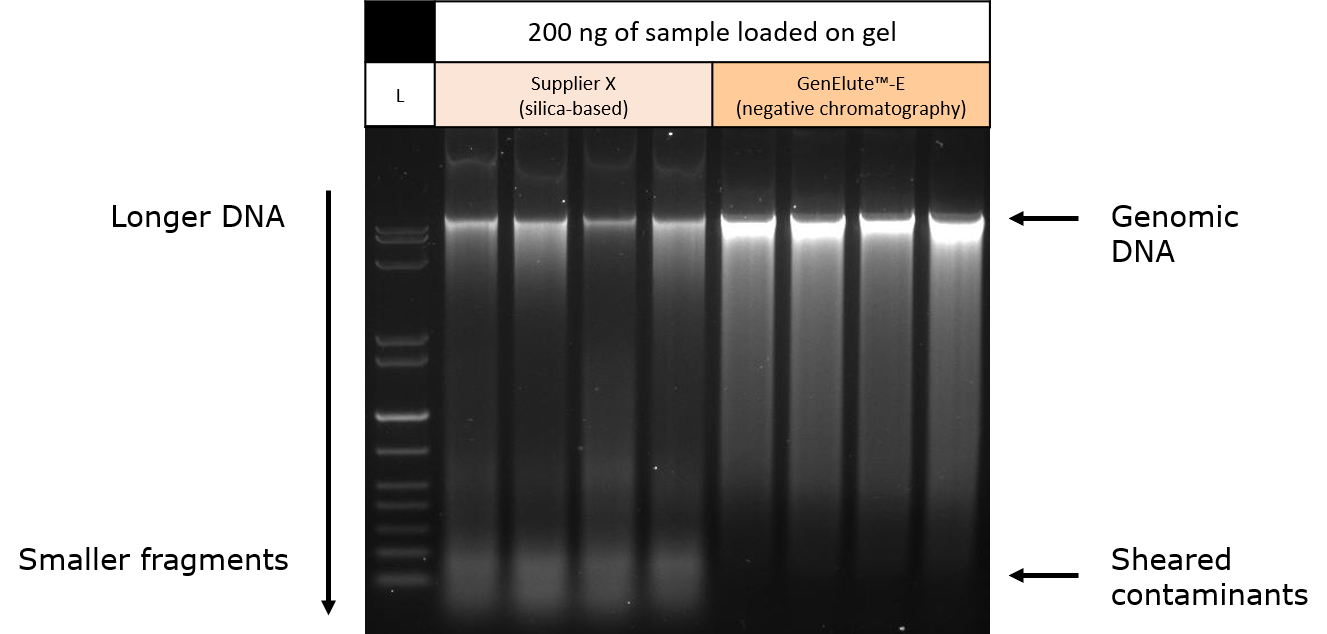
그림 4.마우스 간 조직 시료에서 얻은 정제된 유전체 DNA의 겔 전기영동. 마우스 조직은 실리카 기반 스핀 추출 방법(모 공급업체) 또는 크기 배제를 통한 네거티브 크로마토그래피(GenElute™-E 단일 스핀 DNA 정제 키트)를 활용하여 정제되었습니다. 시료는 아가로스 겔의 각 레인 안에 동일한 양의 DNA를 넣기 위해 조정되었습니다. 결과는 실리카 기반 방법을 사용하여 정제된 시료에서 오염 RNA 또는 전단된 DNA의 존재를 나타냅니다.

그림 5.마우스 간 조직 시료에서 유전체 DNA(gDNA) 수율과 오염 RNA 또는 전단된 DNA의 양을 계산하였습니다. 시료는 실리카 기반 스핀 추출 방법(모 공급업체) 또는 크기 배제를 통한 네거티브 크로마토그래피(GenElute™-E 단일 스핀 DNA 정제 키트)를 사용하여 정제되었습니다. DNA의 양은 SYBR green 염색에서 형광 신호를 정량하여 계산하였습니다. 결과는 실리카 기반 정제 스핀 컬럼을 사용하여 조제한 시료에서 작지만 유의미한 RNA 및 전단된 DNA 오염물질의 양을 보여줍니다.
qPCR에서의 간섭
실리카 기반 정제 방법은 변성 염 및 에탄올 같은 유기 용매를 정제되는 시료에 주입합니다. 이러한 오염물질은 다운스트림 애플리케이션으로 쉽게 옮겨 가서 효소 반응 억제를 초래할 수 있습니다. 이러한 오염물질의 제거는 qPCR 같은 효소 방법을 더욱 민감하고 강력하게 합니다.
효소 과정을 억제하는 오염물질의 존재를 평가하기 위해서, 각각의 스핀 정제 방법을 사용하여 마우스 신장 시료에서 유전체 DNA를 정제하였습니다. 최종 시료는 qPCR 분석 실험에 사용되었습니다(그림 6). 또한 베타 액틴에 해당하는 유전자가 내생 대조군으로서 증폭되었습니다. 데이터는 실리카 정제 시료에 대한 증폭 곡선이 GenElute™-E 네거티브 크로마토그래피(크기 배제 방식) 정제 방법을 사용하여 조제한 시료에 비해 오른쪽으로 이동된 것을 보여줍니다. 이는 실리카 정제 시료에서 간섭 오염물질이 존재하거나 계산된 수율보다 낮다는 것을 나타냅니다.

그림 6.유전체 DNA 추출물에서 간섭 오염물질을 측정하기 위한 qPCR 분석. A) 모 공급업체의 실리카 기반 스핀 추출 키트를 이용하여 마우스 신장 조직에서 추출한 유전체 DNA에 대한 증폭 곡선(파란 곡선)과 GenElute™-E 네거티브 크로마토그래피(크기 배제) 스핀 추출 키트에 대한 증폭 곡선(주황 곡선). 실리카 정제 시료에 대한 곡선은 GenElute™-E 정제 시료에 비해 오른쪽으로 이동되었으며, 이는 실리카 기반 추출물에서 간섭 오염물질의 존재 또는 시작 농도의 과대 평가를 시사합니다. B) 내생 대조군 참조로서 베타 액틴을 사용한 Cq(정량주기) 계산.
계속 읽으시려면 로그인하거나 계정을 생성하세요.
계정이 없으십니까?