プレデザインsiRNA
MISSION®プレデザインsiRNAは、メルクの独占的提携パートナーであるRosetta Inpharmaticsが所有するsiRNA設計アルゴリズムを用いて作成しました。Rosetta siRNA設計アルゴリズムでは、位置特異的スコアリング行列とシード領域の情報を利用して、標的遺伝子の最も特異的かつ有効な配列を予測します。アルゴリズムの規則は、3年間の遺伝子サイレンシング実験で収集した経験的データを用いて作成しました。
製品の利点
- クラス最高レベルの保証付きの遺伝子サイレンシング
- 低発現遺伝子を効率的にノックダウン
- 11種類のポジティブコントロールsiRNAによる簡単なトランスフェクションの最適化
- 8種類のネガティブコントロールsiRNAによる配列特異的サイレンシングと非特異的サイレンシングの識別
- 機能性検証済みのプレデザインsiRNA
製品の特長
- 動物種:ヒト、マウス、ラット
- 保証収量(OD):2 OD(10 nmol)、5 OD(25 nmol)、10 OD(50 nmol)
- 精製:脱塩またはHPLC
- 鎖長:dTdTオーバーハングを有する21 mer duplex
- 修飾:センス鎖上の5' amine
- 品質管理:100%質量分析
- 納品形態:チューブ入り乾燥品
プール製品
プール品は、duplex 5 nmol 4本分を1本のチューブ(20 nmolのプール)にし、さらに同量のduplex 5 nmol 4本分を1本のチューブ(20 nmolの追加プール)にして計2本を納品される場合が多いです。しかし、私たちが開発した高度液体取扱装置は、上記以外の幅広い納品方法が可能です。具体的ニーズの対応可否については、customjp.ts@merckgroup.comにお問い合わせください。
バリデーション済みのsiRNA
mRNAが75%以上ノックダウンされたかどうか、一般的な標的遺伝子に対して多数検証しました(図1データの一例。発注が多い検証済みsiRNAの遺伝子記号順一覧表)。検証済みのsiRNAはトランスフェクション最適化に向いており、ポジティブコントロールに適しています。
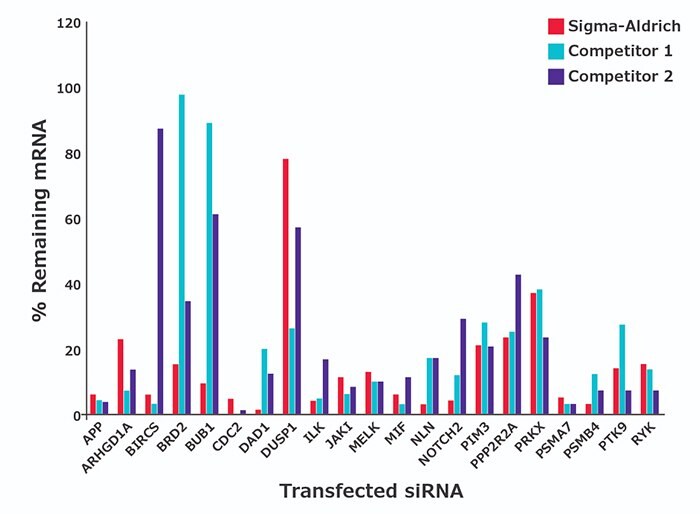
図1.濃度30 nMでプレデザインsiRNAを導入したHeLa細胞。遺伝子導入の48時間後の時点での遺伝子発現残存率をqPCRにより測定した (偽発現との比較)。データは生物学的レプリケート4つの平均を示す。
| 検証したsiRNA(遺伝子記号順) | |||||||
|---|---|---|---|---|---|---|---|
| ABCC1 | CDC7 | DYRK1A | IGFBP4 | MLH1 | PIK3R4 | PRPS1 | SIK2 |
| ACP1 | CDC73 | DYRK1B | IL6ST | MSH2 | PIM2 | PRSS23 | SLC16A1 |
| ACP2 | CDK1 | EEF2K | ILK | MST4 | PIN1 | PSEN1 | SLC25A17 |
| ACVR1 | CDK11B | EIF2AK4 | IMPA2 | MTA2 | PINK1 | PSEN2 | SLC2A1 |
| ACVR1B | CDK17 | EIF4A3 | INPPL1 | MTM1 | PIP4K2B | PSMA7 | SLC30A1 |
| ADAM10 | CDK2 | EMR2 | IP6K1 | MTMR1 | PIP5K1A | PSMB4 | SLK |
| ADAM12 | CDK4 | ENPP1 | IP6K2 | MTMR2 | PKN2 | PSMB5 | SMU1 |
| ADAM15 | CDK5 | EPHA5 | IPMK | MTMR3 | PLAUR | PSMB6 | SNRK |
| ADIPOR1 | CDK6 | EPHB4 | IRAK1 | MTMR6 | PLK1 | PSPH | SORT1 |
| ADIPOR2 | CDK7 | F3 | IRAK4 | MTOR | PLK4 | PTK2 | SRC |
| ADRBK1 | CDK8 | FADD | ITPK1 | NADK | POLK | PTP4A1 | SRPK1 |
| AKT1 | CDK9 | FDFT1 | JAK1 | NCSTN | PPAP2C | PTPLA | ST7 |
| AKT2 | CDKL5 | FER | JUND | NDRG1 | PPAT | PTPN1 | STAT3 |
| ALPL | CDKN1B | FMNL1 | JUP | NEDD8 | PPM1D | PTPN11 | STK16 |
| APP | CDKN3 | FURIN | LANCL1 | NEK2 | PPME1 | PTPN12 | STK24 |
| ARAF | CELSR1 | FYN | LATS1 | NEK6 | PPP1CA | PTPN14 | STK3 |
| ARHGDIA | CERK | FZD4 | LDHA | NEK7 | PPP1CB | PTPN23 | STYX |
| ATF1 | CHEK1 | FZD5 | LEPR | NEK9 | PPP1CC | PTPN9 | TAOK1 |
| ATM | CHUK | FZD6 | LGALS3 | NET1 | PPP1R11 | PTPRE | TAOK3 |
| ATP6V0C | CKS2 | GLTSCR2 | LIMK2 | NF1 | PPP1R12A | PTPRF | TBK1 |
| ATR | CLPP | GPRC5A | LRP5 | NLN | PPP1R2 | PTPRJ | TEK |
| AURKB | CPE | GRB2 | LRP6 | NME1 | PPP1R3C | PTPRK | TESK1 |
| AXIN1 | CPT1A | GRK6 | LTBR | NME1-NME2 | PPP1R7 | PTPRS | TFRC |
| AXL | CRKL | GRN | LYN | NME2 | PPP2CA | RAB22A | TGFBR1 |
| BACE1 | CSK | GSK3B | MAD2L1 | NOTCH2 | PPP2CB | RAF1 | TGM1 |
| BACE2 | CSNK2A1 | HADHB | MANF | NPR1 | PPP2R1A | RAP1B | TGM2 |
| BAD | CTNNB1 | HBEGF | MAP2K2 | NR1H2 | PPP2R2A | RARG | THRA |
| BAG1 | CTSA | HCG 1757335 | MAP2K5 | NR1H3 | PPP2R5A | RB1 | TK1 |
| BCAT1 | CXCR4 | HDAC1 | MAP3K3 | NR2C2 | PPP2R5D | RELA | TKT |
| BIRC5 | CXCR7 | HDAC2 | MAP3K4 | NR2F2 | PPP2R5E | RHOA | TLR4 |
| BMP6 | CYLD | HECTD1 | MAPK14 | NUP85 | PPP3CC | RIOK3 | TM4SF1 |
| BMPR1A | DAD1 | HIPK2 | MAPK3 | OCRL | PPP5C | RIPK1 | TNFRSF10B |
| BRAF | DAPK3 | HIPK3 | MAPK6 | OXSR1 | PPP6C | RIPK2 | TRIM28 |
| BUB1 | DCN | HPRT1 | MAPK8 | PAK2 | PREPL | RNF10 | TWF1 |
| BUB1B | DDR1 | HSPA1A | MAPK9 | PASK | PRKACA | RNF5 | TYRO3 |
| CASK | DGKA | HSPA1B | MAPKAPK5 | PBK | PRKACB | ROCK1 | VEGFC |
| CCL2 | DMBT1 | HSPB8 | MARK3 | PCNA | PRKAG1 | ROCK2 | VIPR1 |
| CCNA2 | DNMT1 | HTRA1 | MASTL | PCSK9 | PRKAR1A | ROR2 | VRK2 |
| CCNC | DUSP10 | HUNK | MBTPS1 | PDE8A | PRKAR2A | RPS6KA3 | WNK1 |
| CCND1 | DUSP11 | ICAM1 | MELK | PDGFRB | PRKCA | RPS6KA4 | YES1 |
| CCT2 | DUSP12 | ICK | MET | PDK1 | PRKCI | RPS6KB1 | ZMPSTE24 |
| CD63 | DVL2 | IGF1R | MIF | PGK1 | PRKCZ | RYK | |
| CD82 | DVL3 | IGF2R | MINPP1 | PHPT1 | PRKDC | SHC1 | |
引用文献の選択
さらなるサポートが必要な場合、テクニカルサービス(customjp.ts@merckgroup.com)にご相談ください。
MISSIONはMerck KGaA(ドイツ)とその関連会社の商標です。ラベルライセンスについて
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?