Alle Fotos(1)
Wichtige Dokumente
T3411
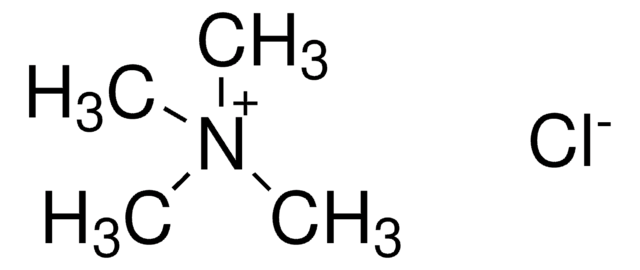
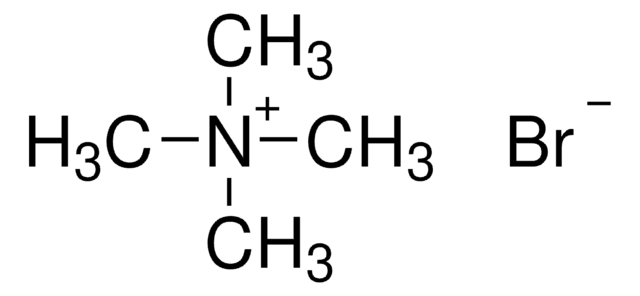
Tetramethylammoniumchlorid -Lösung
for molecular biology
Synonym(e):
N,N,N-Trimethylmethanaminium chloride
Anmeldenzur Ansicht organisationsspezifischer und vertraglich vereinbarter Preise
Alle Fotos(1)
About This Item
CAS-Nummer:
MDL-Nummer:
UNSPSC-Code:
12352107
PubChem Substanz-ID:
NACRES:
NA.31
Empfohlene Produkte
Qualität
Molecular Biology
for molecular biology
Qualitätsniveau
Konzentration
5 M
Fremdaktivität
DNase, RNase, none detected
SMILES String
[Cl-].C[N+](C)(C)C
InChI
1S/C4H12N.ClH/c1-5(2,3)4;/h1-4H3;1H/q+1;/p-1
InChIKey
OKIZCWYLBDKLSU-UHFFFAOYSA-M
Allgemeine Beschreibung
Tetramethylammonium bindet AT-reiche DNA-Polymere und hebt gleichzeitig das bevorzugte Schmelzen von AT- gegenüber GC-Basenpaaren auf. Es wird als 0,2-μm-filtrierte Lösung in 18 Megaohm Wasser bereitgestellt.
Anwendung
Tetramethylammoniumchloridlösung (TMAC) wird wie folgt verwendet:
- bei der Herstellung eines Hybridisierungscocktails für Array-Hybridisierung und -Scannen
- in Next-Generation-Sequencing (NGS) und der genomweiten unvoreingenommenen Identifizierung von Doppelstrangbrüchen, die durch Erstellen einer Sequenzierungsbibliothek (GUIDE-seq) ermöglicht wird
- bei der Herstellung von TMAC-Puffer und Bead-Hybridisierungsmischung für Hybridisierung und Nachweis
Hier finden Sie alle aktuellen Versionen:
Besitzen Sie dieses Produkt bereits?
In der Dokumentenbibliothek finden Sie die Dokumentation zu den Produkten, die Sie kürzlich erworben haben.
Kunden haben sich ebenfalls angesehen
W B Melchior et al.
Proceedings of the National Academy of Sciences of the United States of America, 70(2), 298-302 (1973-02-01)
Several small alkylammonium ions can eliminate, or even reverse, the usual dependence of the DNA transition temperature on base composition. For example, in 3 M tetramethylammonium chloride, or 2.4 M tetraethylammonium chloride, DNAs of different base compositions all melt at
Nikolay L Malinin et al.
Nature protocols, 16(12), 5592-5615 (2021-11-14)
Genome-wide unbiased identification of double-stranded breaks enabled by sequencing (GUIDE-seq) is a sensitive, unbiased, genome-wide method for defining the activity of genome-editing nucleases in living cells. GUIDE-seq is based on the principle of efficient integration of an end-protected double-stranded oligodeoxynucleotide
Hybridization of genomic DNA to oligonucleotide probes in the presence of tetramethylammonium chloride.
A G DiLella et al.
Methods in enzymology, 152, 447-451 (1987-01-01)
Xiao-Yong Li et al.
Methods in molecular biology (Clifton, N.J.), 809, 3-26 (2011-11-25)
Immunoprecipitation of cross-linked chromatin in combination with microarrays (ChIP-chip) or ultra high-throughput sequencing (ChIP-seq) is widely used to map genome-wide in vivo transcription factor binding. Both methods employ initial steps of in vivo cross-linking, chromatin isolation, DNA fragmentation, and immunoprecipitation.
Markus Welcker et al.
Genes & development, 27(23), 2531-2536 (2013-12-04)
The Fbw7 tumor suppressor targets a broad network of proteins for ubiquitylation. Here we show critical functions for Fbw7 dimerization in regulating the specificity and robustness of degradation. Dimerization enables Fbw7 to target substrates through concerted binding to two suboptimal
Unser Team von Wissenschaftlern verfügt über Erfahrung in allen Forschungsbereichen einschließlich Life Science, Materialwissenschaften, chemischer Synthese, Chromatographie, Analytik und vielen mehr..
Setzen Sie sich mit dem technischen Dienst in Verbindung.