S7847
CpGenome Turbo Bisulfit-Modifizierungskit
The CpGenome Turbo Bisulfite Modification Kit is designed to simplify & streamline the bisulfite modification process. In just 90 minutes go from DNA sample to bisulfite converted DNA ready for analysis.
Anmeldenzur Ansicht organisationsspezifischer und vertraglich vereinbarter Preise
Alle Fotos(3)
About This Item
UNSPSC-Code:
12161503
eCl@ss:
32161000
NACRES:
NA.25
Empfohlene Produkte
Qualitätsniveau
Hersteller/Markenname
Chemicon®
CpGenome
Anwendung(en)
genomic analysis
Versandbedingung
ambient
Verwandte Kategorien
Allgemeine Beschreibung
Das CpGenome Turbo Bisulfit-Modifikations-Kit wurde entwickelt, um den Prozess der Bisulfit-Modifikation zu vereinfachen und zu rationalisieren. Dieses Kit enthält alle wichtigen Reagenzien für die Bisulfit-Modifikation, so dass die Wiederherstellung modifizierter DNA in etwa 90 Minuten möglich ist, im Vergleich zu 3–16 Stunden bei ähnlichen Kits. Bei diesem schnellen, aber effektiven Verfahren wird eine firmeneigene Mischung von Modifizierungsreagenzien verwendet, die kürzere Umwandlungszeiten ermöglicht. Diese einzigartige Reagenzienmischung ermöglicht die vollständige Umwandlung unmethylierter Cytosine bei gleichzeitiger Minimierung der Schäden an der Eingangs-DNA. Nach der Modifizierung und Desulfonierung wird die modifizierte DNA gereinigt und mit Hilfe der mitgelieferten Zentrifugationssäulen eluiert. Das Ergebnis ist aufgereinigte, Bisulfit-modifizierte DNA, die für eine Vielzahl von nachgelagerten Anwendungen verwendet werden kann.
Wichtige Eigenschaften
Wichtige Eigenschaften
- Eingangsprobe zu modifizierter DNA in 90 Minuten
- Konversionseffizienz von 99,9 %
- Wirksam mit nur 1 ng Eingangs-DNA
- Gewinnung von modifizierter DNA in nur 25 Mikrolitern
- Bewährte Leistung in mehreren nachgelagerten Anwendungen
Die Methylierung von Cytosinen am 5′ an Guanosin hat eine weitreichende Wirkung auf die Expression vieler eukaryotischer Gene. In gesunden Zellen tritt die Methylierung überwiegend in CG-armen Regionen auf, während CG-reiche Regionen (CpG-Inseln genannt) unmethyliert bleiben. Ausnahmen sind die ausgedehnte Methylierung von CpG-Inseln im Zusammenhang mit der transkriptionellen Inaktivierung von regulatorischen Regionen geprägter Gene und von Genen auf dem inaktiven X-Chromosom von Frauen. Die aberrante Methylierung von normalerweise unmethylierten CpG-Inseln wurde in immortalisierten und transformierten Zellen als relativ häufiges Ereignis dokumentiert und mit der transkriptionalen Inaktivierung von definierten Tumorsupressorgenen in humanen Krebsarten assoziiert.
Es wurden mehrere Methoden zur Bestimmung des Methylierungsstatus von Cytosin entwickelt. Dazu gehören die Verwendung von Antikörpern oder Protein-Methyl-bindenden Domänen, der Verdau mit methylierungsempfindlichen, -unempfindlichen oder -abhängigen Restriktionsenzymen wie beim RLGS (Restriktionslandmark Genomic Scanning), die Oligonukleotid-Array-Hybridisierung, die Bisulfit-Sequenzierung genomischer DNA und die Methylierungs-spezifische PCR (MSP).
Die Sequenzierung genomischer DNA ist zwar zeit- und arbeitsintensiv, bietet aber eine universellere Nachweismethode. MSP ist eine etablierte Technologie zur Überwachung abnormaler Genmethylierung in ausgewählten Gensequenzen. Unter Verwendung geringer DNA-Mengen bietet dieses Verfahren einen empfindlichen und spezifischen Nachweis von 5-Methylcytosin in Promotoren. Sie wird genutzt, um die Funktion von Tumorsuppressorgenen zu bestimmen und eine neue Strategie für die Tumorfrüherkennung durch die Untersuchung von DNA aus Geweben und Körperflüssigkeiten zu entwickeln.
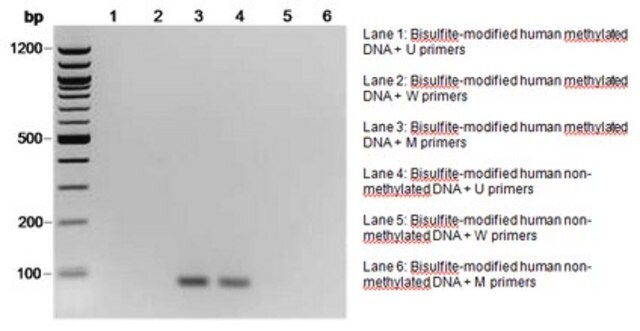
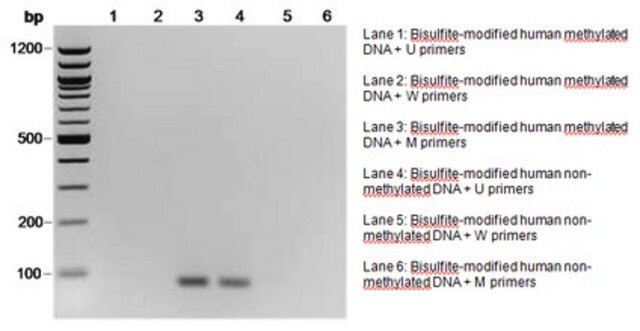
Sowohl bei der genomischen Bisulfit-Sequenzierung als auch bei der MSP wird zunächst eine Bisulfit-Modifikation der DNA-Probe durchgeführt. Bei der Bisulfit-Reaktion werden alle unmethylierten Cytosine desaminiert und sulfoniert, wodurch sie in Uracil umgewandelt werden, während 5-Methylcytosine unverändert bleiben. Die Sequenz der behandelten DNA wird sich also unterscheiden, je nachdem, ob die DNA ursprünglich methyliert oder unmethyliert ist. Auch die ursprünglich komplementären DNA-Stränge sind nach der Cytosin-Umwandlung nicht mehr komplementär. Primer für die MSP können so konzipiert werden, dass sie entweder einen bisulfitempfindlichen, unmethylierten Strang oder einen bisulfitresistenten, methylierten Strang auf der Grundlage dieser chemisch induzierten Unterschiede spezifisch amplifizieren. Millipore bietet eine Auswahl von CpG Wiz MSP-Kits an, die eine genspezifische Analyse mittels MSP ermöglichen. Um mehr über CpG Wiz Kits und die MSP-Technologie zu erfahren, klicken Sie hier.
Es wurden mehrere Methoden zur Bestimmung des Methylierungsstatus von Cytosin entwickelt. Dazu gehören die Verwendung von Antikörpern oder Protein-Methyl-bindenden Domänen, der Verdau mit methylierungsempfindlichen, -unempfindlichen oder -abhängigen Restriktionsenzymen wie beim RLGS (Restriktionslandmark Genomic Scanning), die Oligonukleotid-Array-Hybridisierung, die Bisulfit-Sequenzierung genomischer DNA und die Methylierungs-spezifische PCR (MSP).
Die Sequenzierung genomischer DNA ist zwar zeit- und arbeitsintensiv, bietet aber eine universellere Nachweismethode. MSP ist eine etablierte Technologie zur Überwachung abnormaler Genmethylierung in ausgewählten Gensequenzen. Unter Verwendung geringer DNA-Mengen bietet dieses Verfahren einen empfindlichen und spezifischen Nachweis von 5-Methylcytosin in Promotoren. Sie wird genutzt, um die Funktion von Tumorsuppressorgenen zu bestimmen und eine neue Strategie für die Tumorfrüherkennung durch die Untersuchung von DNA aus Geweben und Körperflüssigkeiten zu entwickeln.
Sowohl bei der genomischen Bisulfit-Sequenzierung als auch bei der MSP wird zunächst eine Bisulfit-Modifikation der DNA-Probe durchgeführt. Bei der Bisulfit-Reaktion werden alle unmethylierten Cytosine desaminiert und sulfoniert, wodurch sie in Uracil umgewandelt werden, während 5-Methylcytosine unverändert bleiben. Die Sequenz der behandelten DNA wird sich also unterscheiden, je nachdem, ob die DNA ursprünglich methyliert oder unmethyliert ist. Auch die ursprünglich komplementären DNA-Stränge sind nach der Cytosin-Umwandlung nicht mehr komplementär. Primer für die MSP können so konzipiert werden, dass sie entweder einen bisulfitempfindlichen, unmethylierten Strang oder einen bisulfitresistenten, methylierten Strang auf der Grundlage dieser chemisch induzierten Unterschiede spezifisch amplifizieren. Millipore bietet eine Auswahl von CpG Wiz MSP-Kits an, die eine genspezifische Analyse mittels MSP ermöglichen. Um mehr über CpG Wiz Kits und die MSP-Technologie zu erfahren, klicken Sie hier.
Anwendung
Forschungskategorie
Epigenetik und Zellkernfunktion
Epigenetik und Zellkernfunktion
Für das Design von MSP-Primern verwenden Sie bitte das Softwarepaket MethPrime. Hier klicken
Komponenten
Bisulfit-Konvertierungsreagenz 1
Bisulfit-Konvertierungsreagenz 2
Bisulfit-Reagenz-Verdünnungsmittel
DNA-Bindungspuffer
Waschpuffer NT3 (Konzentrat)*
Elutionspuffer NE
Modifizierte DNA-Aufreinigungssäulen
2-ml-Sammelröhrchen
*Der DNA-Waschpuffer erfordert die Zugabe von 100 % Ethanol.
Bisulfit-Konvertierungsreagenz 2
Bisulfit-Reagenz-Verdünnungsmittel
DNA-Bindungspuffer
Waschpuffer NT3 (Konzentrat)*
Elutionspuffer NE
Modifizierte DNA-Aufreinigungssäulen
2-ml-Sammelröhrchen
*Der DNA-Waschpuffer erfordert die Zugabe von 100 % Ethanol.
Rechtliche Hinweise
CHEMICON is a registered trademark of Merck KGaA, Darmstadt, Germany
Haftungsausschluss
Sofern in unserem Katalog oder anderen Begleitdokumenten unserer Produkte nicht anders angegeben, sind unsere Produkte nur für Forschungszwecke vorgesehen und nicht für andere Zwecke zu verwenden, einschließlich u. a. zur unautorisierten kommerziellen Verwendung, zur In-vitro-Diagnostik, für Ex-vivo- oder In-vivo-Therapiezwecke oder jegliche Art der Einnahme oder Anwendung bei Menschen oder Tieren.
Signalwort
Danger
H-Sätze
Gefahreneinstufungen
Acute Tox. 4 Inhalation - Acute Tox. 4 Oral - Aquatic Chronic 3 - Eye Dam. 1 - Met. Corr. 1 - Skin Corr. 1C
Zusätzliche Gefahrenhinweise
Lagerklassenschlüssel
8A - Combustible corrosive hazardous materials
Analysenzertifikate (COA)
Suchen Sie nach Analysenzertifikate (COA), indem Sie die Lot-/Chargennummer des Produkts eingeben. Lot- und Chargennummern sind auf dem Produktetikett hinter den Wörtern ‘Lot’ oder ‘Batch’ (Lot oder Charge) zu finden.
Besitzen Sie dieses Produkt bereits?
In der Dokumentenbibliothek finden Sie die Dokumentation zu den Produkten, die Sie kürzlich erworben haben.
Zhentao Yang et al.
Cell reports, 19(9), 1846-1857 (2017-06-01)
2-hydroxyglutarate-(2-HG)-mediated inhibition of TET2 activity influences DNA hypermethylation in cells harboring mutations of isocitrate dehydrogenases 1 and 2 (IDH1/2). Here, we show that 2-HG also regulates DNA methylation mediated by DNA methyltransferase 1 (DNMT1). DNMT1-dependent hypermethylation of the RIP3 promoter
Characterization of induced tissue-specific stem cells from pancreas by a synthetic self-replicative RNA.
Miyagi-Shiohira, C; Nakashima, Y; Kobayashi, N; Saitoh, I; Watanabe, M; Noguchi, H
Scientific Reports null
Hernán G Hernández et al.
BioTechniques, 55(4), 181-197 (2013-10-11)
Comprehensive analysis of DNA methylation patterns is critical for understanding the molecular basis of many human diseases. While hundreds of PCR-based DNA methylation studies are published every year, the selection and implementation of appropriate methods for these studies can be
Unser Team von Wissenschaftlern verfügt über Erfahrung in allen Forschungsbereichen einschließlich Life Science, Materialwissenschaften, chemischer Synthese, Chromatographie, Analytik und vielen mehr..
Setzen Sie sich mit dem technischen Dienst in Verbindung.