R5125
Ribonucléase A from bovine pancreas
Type III-A, ≥85% RNase A basis (SDS-PAGE), 85-140 Kunitz units/mg protein
Synonyme(s) :
RNAse a, RNase A, Ribonucléase pancréatique, Ribonucléate 3′-pyrimidino-oligonucléotido-hydrolase
About This Item
Produits recommandés
Source biologique
bovine pancreas
Niveau de qualité
Type
Type III-A
Pureté
≥85% RNase A basis (SDS-PAGE)
Forme
lyophilized powder
Activité spécifique
85-140 Kunitz units/mg protein
Poids mol.
~13,700
Technique(s)
cell based assay: suitable
Impuretés
salt, essentially free
Adéquation
suitable for molecular biology
Application(s)
diagnostic assay manufacturing
Activité étrangère
protease, essentially free
Température de stockage
−20°C
InChI
1S/C9H14N4O3/c10-2-1-8(14)13-7(9(15)16)3-6-4-11-5-12-6/h4-5,7H,1-3,10H2,(H,11,12)(H,13,14)(H,15,16)
Clé InChI
CQOVPNPJLQNMDC-UHFFFAOYSA-N
Vous recherchez des produits similaires ? Visite Guide de comparaison des produits
Catégories apparentées
Description générale
Application
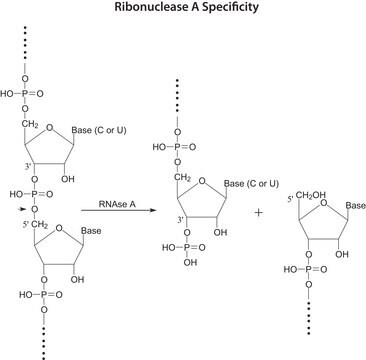
- La RNase A est utilisée pour supprimer l′ARN présent dans les préparations d′ADN plasmidique et d′ADN génomique ainsi que dans les échantillons protéiques.
- Elle est également employée dans l′analyse de séquences d′ARN et les tests de protection.
- Elle a également servi d′outil dans la conception de médicaments assistée par ordinateur.
- La RNase A supporte l′analyse des séquences d′ARN.
- Elle hydrolyse l′ARN contenu dans les échantillons protéiques.
- La purification de l′ADN est supportée par la RNase A.
Actions biochimiques/physiologiques
Caractéristiques et avantages
Notes préparatoires
Remarque sur l'analyse
Application
Inhibiteur
Mention d'avertissement
Danger
Mentions de danger
Conseils de prudence
Classification des risques
Resp. Sens. 1
Code de la classe de stockage
11 - Combustible Solids
Classe de danger pour l'eau (WGK)
WGK 3
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Équipement de protection individuelle
Eyeshields, Gloves, type N95 (US)
Certificats d'analyse (COA)
Recherchez un Certificats d'analyse (COA) en saisissant le numéro de lot du produit. Les numéros de lot figurent sur l'étiquette du produit après les mots "Lot" ou "Batch".
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Les clients ont également consulté
Protocoles
This procedure may be used for determination of Ribonuclease A (RNase A) activity.
Chromatograms
application for HPLCNotre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique