推薦產品
生物源
rabbit
品質等級
抗體表格
serum
抗體產品種類
primary antibodies
無性繁殖
polyclonal
物種活性
Saccharomyces cerevisiae, human, vertebrates
製造商/商標名
Upstate®
技術
ChIP: suitable
dot blot: suitable
western blot: suitable
NCBI登錄號
UniProt登錄號
運輸包裝
wet ice
目標翻譯後修改
acetylation (Lys23)
基因資訊
human ... H3C1(8350)
一般說明
组蛋白H3是参与真核细胞染色质结构的五种主要组蛋白之一。H3具有一个主要的球状结构域和一个长N末端的尾巴,其与核糖体核小体′串珠′结构体的结构有关。
组蛋白H3的N末端尾部从球状核小体核心突起,其可能发生几种不同类型的表观遗传修饰,从而影响细胞过程。这些修饰包括甲基或乙酰基与赖氨酸和精氨酸的共价连接,以及丝氨酸或苏氨酸的磷酸化。
组蛋白H3的N末端尾部从球状核小体核心突起,其可能发生几种不同类型的表观遗传修饰,从而影响细胞过程。这些修饰包括甲基或乙酰基与赖氨酸和精氨酸的共价连接,以及丝氨酸或苏氨酸的磷酸化。
特異性
赖氨酸23乙酰化的组蛋白H3。 未识别未乙酰化的重组组蛋白H3
免疫原
卵清蛋白缀合的合成肽(KQLASAcKAARK-C),对应于在赖氨酸23上乙酰化的酵母组蛋白H3的氨基酸18-27,并添加了C端半胱氨酸便于进行缀合。
應用
使用在ChIP,WB中验证的抗乙酰化组蛋白H3(Lys23)抗体(兔多克隆抗体)检测乙酰化组蛋白H3(Lys23),也称为H3K23Ac,组蛋白H3(乙酰化K23)。
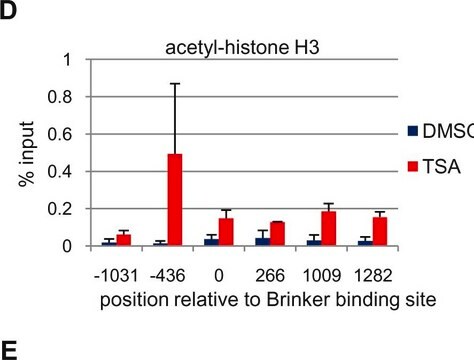
染色质免疫沉淀:
代表性批次数据。
使用2 µL正常兔血清或2 µL抗乙酰组蛋白H3(Lys23)和Magna ChIP A试剂盒(目录号17-610),对从HeLa细胞制备的超声染色质(每IP 1 X 10e6细胞当量)进行染色质免疫沉淀。 使用对人GAPDH启动子区域具有特异性的对照引物作为阳性基因座,并使用MyoD引物作为阴性基因座,通过qPCR验证了乙酰基组蛋白H3(Lys23)相关DNA片段的成功免疫沉淀。数据表示为每个IP样品相对于每个扩增子和ChIP样品的输入染色质的输入百分比。
请参阅EZ-Magna ChIP A(目录号17-408)或EZ-ChIP(目录号17-371)协议以获取实验详细信息。
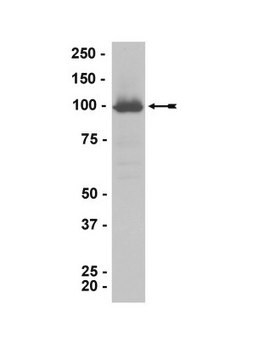
蛋白质印迹分析:
代表性批次数据。
用抗乙酰基组蛋白H3(Lys23)(1:100,000稀释)探测丁酸钠处理的HeLa细胞(泳道1,目录号17-305)和重组组蛋白H3(泳道2,目录号14-494)的酸提取物。
箭头表示乙酰基组蛋白H3(~17 kDa)
斑点印迹:
代表性批次数据。
将40 ng和4 ng量的不同修饰的组蛋白肽(见表1)转移至PVDF膜上,并用抗乙酰基组蛋白H3(Lys23)抗体(1:2000稀释度)进行探测。使用与HRP偶联的山羊抗兔IgG和化学发光检测系统可视化蛋白质。显示了60秒曝光的图像。
代表性批次数据。
使用2 µL正常兔血清或2 µL抗乙酰组蛋白H3(Lys23)和Magna ChIP A试剂盒(目录号17-610),对从HeLa细胞制备的超声染色质(每IP 1 X 10e6细胞当量)进行染色质免疫沉淀。 使用对人GAPDH启动子区域具有特异性的对照引物作为阳性基因座,并使用MyoD引物作为阴性基因座,通过qPCR验证了乙酰基组蛋白H3(Lys23)相关DNA片段的成功免疫沉淀。数据表示为每个IP样品相对于每个扩增子和ChIP样品的输入染色质的输入百分比。
请参阅EZ-Magna ChIP A(目录号17-408)或EZ-ChIP(目录号17-371)协议以获取实验详细信息。
蛋白质印迹分析:
代表性批次数据。
用抗乙酰基组蛋白H3(Lys23)(1:100,000稀释)探测丁酸钠处理的HeLa细胞(泳道1,目录号17-305)和重组组蛋白H3(泳道2,目录号14-494)的酸提取物。
箭头表示乙酰基组蛋白H3(~17 kDa)
斑点印迹:
代表性批次数据。
将40 ng和4 ng量的不同修饰的组蛋白肽(见表1)转移至PVDF膜上,并用抗乙酰基组蛋白H3(Lys23)抗体(1:2000稀释度)进行探测。使用与HRP偶联的山羊抗兔IgG和化学发光检测系统可视化蛋白质。显示了60秒曝光的图像。
研究子类别
组蛋白
组蛋白
研究类别
表观遗传学&核功能
表观遗传学&核功能
品質
通过免疫印迹法对丁酸钠处理过的HeLa细胞的酸提取物进行了常规评估
標靶描述
17kDa
外觀
含0.05%叠氮化钠和30%甘油的兔抗血清。
抗血清
儲存和穩定性
在-20°C下可保存2年
分析報告
对照
经丁酸钠处理过的HeLa细胞的酸提取物
经丁酸钠处理过的HeLa细胞的酸提取物
法律資訊
UPSTATE is a registered trademark of Merck KGaA, Darmstadt, Germany
免責聲明
除非我们的产品目录或产品附带的其他公司文档另有说明,否则我们的产品仅供研究使用,不得用于任何其他目的,包括但不限于未经授权的商业用途、体外诊断用途、离体或体内治疗用途或任何类型的消费或应用于人类或动物。
未找到適合的產品?
試用我們的產品選擇工具.
儲存類別代碼
10 - Combustible liquids
水污染物質分類(WGK)
WGK 1
分析證明 (COA)
輸入產品批次/批號來搜索 分析證明 (COA)。在產品’s標籤上找到批次和批號,寫有 ‘Lot’或‘Batch’.。
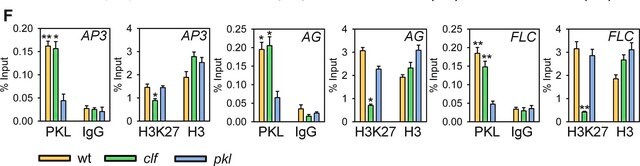
Non-CG methylation patterns shape the epigenetic landscape in Arabidopsis.
Stroud, H; Do, T; Du, J; Zhong, X; Feng, S; Johnson, L; Patel, DJ; Jacobsen, SE
Nature Structural and Molecular Biology null
Alec M Desimone et al.
Molecular and cellular biology, 30(13), 3342-3356 (2010-05-05)
Switching between alternate states of gene transcription is fundamental to a multitude of cellular regulatory pathways, including those that govern differentiation. In spite of the progress in our understanding of such transitions in gene activity, a major unanswered question is
Zhaobo Lang et al.
Molecular cell, 57(6), 971-983 (2015-02-17)
DNA methylation is a conserved epigenetic mark that plays important roles in plant and vertebrate development, genome stability, and gene regulation. Canonical Methyl-CpG-binding domain (MBD) proteins are important interpreters of DNA methylation that recognize methylated CG sites and recruit chromatin
Li Lu et al.
Epigenetics, 10(11), 1044-1053 (2015-12-10)
Histone acetylation and deacetylation are key epigenetic gene regulatory mechanisms that play critical roles in eukaryotes. Acetylation of histone 4 lysine 16 (H4K16ac) is implicated in many cellular processes. However, its biological function and relationship with transcription are largely unexplored
Zain Umer et al.
Epigenetics & chromatin, 12(1), 55-55 (2019-09-25)
Polycomb group (PcG) and trithorax group (trxG) proteins contribute to the specialization of cell types by maintaining differential gene expression patterns. Initially discovered as positive regulators of HOX genes in forward genetic screens, trxG counteracts PcG-mediated repression of cell type-specific
我們的科學家團隊在所有研究領域都有豐富的經驗,包括生命科學、材料科學、化學合成、色譜、分析等.
聯絡技術服務