E1014
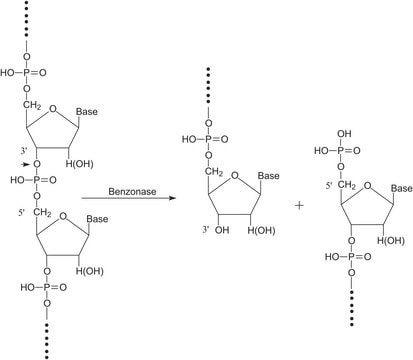
Nucleasas Benzonase®
≥250 units/μL, ≥90% (SDS-PAGE), recombinant, expressed in E. coli, buffered aqueous glycerol solution
Sinónimos:
Endonucleasas from Serratia marcescens
About This Item
Productos recomendados
biological source
Serratia marcescens
Quality Level
recombinant
expressed in E. coli
assay
≥90% (SDS-PAGE)
form
buffered aqueous glycerol solution
mol wt
30 kDa
concentration
≥250 units/μL
application(s)
research use
foreign activity
protease, essentially free
shipped in
wet ice
storage temp.
−20°C
¿Está buscando productos similares? Visita Guía de comparación de productos
General description
Application
Biochem/physiol Actions
Features and Benefits
- Eliminación del ADN del huésped en muestras de microbioma.
- Digestión eficaz del ácido nucleico en una variedad de procedimientos de trabajo.
- Reducción de la viscosidad durante la extracción de proteínas.
Unit Definition
Physical form
Legal Information
Storage Class
10 - Combustible liquids
wgk_germany
WGK 1
flash_point_f
Not applicable
flash_point_c
Not applicable
ppe
Eyeshields, Gloves
Elija entre una de las versiones más recientes:
¿Ya tiene este producto?
Encuentre la documentación para los productos que ha comprado recientemente en la Biblioteca de documentos.
Artículos
The field of proteomics is continually looking for new ways to investigate protein dynamics within complex biological samples. Recently, many researchers have begun to use RNA interference (RNAi) as a method of manipulating protein levels within their samples, but the ability to accurately determine these protein amounts remains a challenge. Fortunately, over the past decade, the field of proteomics has witnessed significant advances in the area of mass spectrometry. These advances, both in instrumentation and methodology, are providing researchers with sensitive assays for both identification and quantification of proteins within complex samples. This discussion will highlight some of these methodologies, namely the use of Multiple Reaction Monitoring (MRM) and Protein-AQUA.
Nuestro equipo de científicos tiene experiencia en todas las áreas de investigación: Ciencias de la vida, Ciencia de los materiales, Síntesis química, Cromatografía, Analítica y muchas otras.
Póngase en contacto con el Servicio técnico