UPS1
Set d′étalon de protéomique universel
Protein Mass Spectrometry Calibration Standard
Synonyme(s) :
Standard Set
About This Item
Produits recommandés
Forme
ready-to-use solution
Niveau de qualité
Qualité
Protein Mass Spectrometry Calibration Standard
Technique(s)
mass spectrometry (MS): suitable
Température de stockage
−20°C
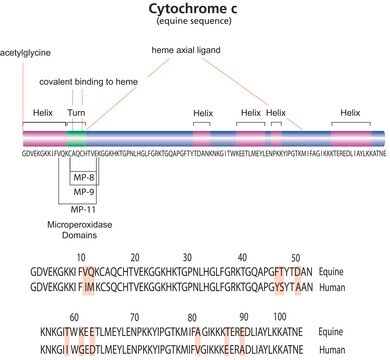
Description générale
Application
- Encadrer des ensembles de données expérimentaux critiques pour confirmer la robustesse des méthodes d'analyse
- Comparer des données de spectrométrie de masse ou d′autres données protéomiques générées dans différents laboratoires avec diverses stratégies et instruments analytiques
- Identifier les limites d′algorithmes de recherche et de systèmes d′analyse en protéomique
- Servir de référence externe pour faciliter l′évaluation de données tirées d′échantillons mal définis
Caractéristiques et avantages
- Testez la force de votre stratégie analytique
- Résolvez les problèmes liés à votre protocole analytique et optimisez-le
- Confirmez la conformité de votre système avant d′analyser des échantillons critiques
- Normalisez les résultats analytiques obtenus à des dates différentes ou dans des laboratoires différents
Composants de kit également disponibles séparément
- T6567Trypsin from porcine pancreas, Proteomics Grade, BioReagent, Dimethylated 20 μgFDS
Produit(s) apparenté(s)
Mention d'avertissement
Danger
Mentions de danger
Conseils de prudence
Classification des risques
Acute Tox. 4 Oral - Eye Dam. 1 - Repr. 1B - Resp. Sens. 1 - Skin Irrit. 2 - STOT SE 3
Organes cibles
Respiratory system
Code de la classe de stockage
6.1C - Combustible acute toxic Cat.3 / toxic compounds or compounds which causing chronic effects
Classe de danger pour l'eau (WGK)
WGK 3
Certificats d'analyse (COA)
Recherchez un Certificats d'analyse (COA) en saisissant le numéro de lot du produit. Les numéros de lot figurent sur l'étiquette du produit après les mots "Lot" ou "Batch".
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Les clients ont également consulté
Articles
The era of high-throughput proteomics has recently blossomed due in large part to advances in the methods by which proteins and proteomes are analyzed. Improved fractionation techniques, combined with advances in mass spectrometry, have decreased concerns of sample complexity, and directed more focus towards high-throughput techniques.
Contenu apparenté
Standardize Your Research. We offer both the Universal Proteomics Standard and the Proteomics Dynamic range Standard as complex, well-defined, well characterized reference standards for mass spectrometry.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique