SAE0045
HRV-3C-Protease
N-Terminal His tagged recombinant protein, aqueous solution, 0.8-1.2 mg/mL
Synonym(e):
3C-Protease des humanen Rhinovirus, PreScission-Protease, Sequenz-LEVLFQGP-Protease
About This Item
Empfohlene Produkte
Biologische Quelle
human (human Rhinovirus Type 14)
Qualitätsniveau
Assay
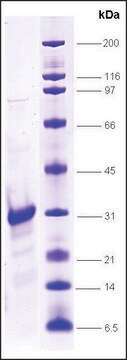
≥90% (SDS-PAGE)
Form
aqueous solution
Spezifische Aktivität
≥5000 U/mg
Mol-Gew.
21 kDa
Konzentration
0.8-1.2 mg/mL
Methode(n)
protein purification: suitable
Eignung
suitable for protein modification
Anwendung(en)
life science and biopharma
Versandbedingung
dry ice
Lagertemp.
−20°C
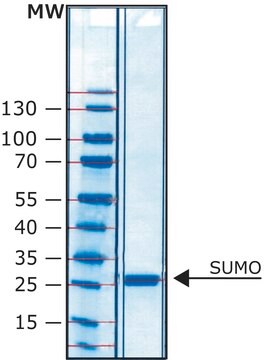
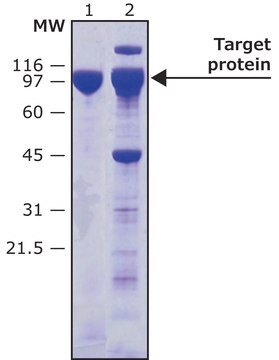
Allgemeine Beschreibung
Die proteolytische Spaltung tritt zwischen dem Gln- und dem Gly-Rest auf.
HRV-3C-Protease ist hilfreiches für die Spaltung rekombinanter Proteine, die als Fusionsprotein mit dieser Sequenz exprimiert werden, zwischen der Transportdomäne und dem betroffenen Protein.
Diese rekombinante Version enthält einen Sechs-Histidin-Tag und kann leicht durch IMAC-Chromatographie entfernt werden.
Anwendung
Einheitendefinition
Physikalische Form
Lagerklassenschlüssel
10 - Combustible liquids
WGK
WGK 2
Analysenzertifikate (COA)
Suchen Sie nach Analysenzertifikate (COA), indem Sie die Lot-/Chargennummer des Produkts eingeben. Lot- und Chargennummern sind auf dem Produktetikett hinter den Wörtern ‘Lot’ oder ‘Batch’ (Lot oder Charge) zu finden.
Besitzen Sie dieses Produkt bereits?
In der Dokumentenbibliothek finden Sie die Dokumentation zu den Produkten, die Sie kürzlich erworben haben.
Kunden haben sich ebenfalls angesehen
Unser Team von Wissenschaftlern verfügt über Erfahrung in allen Forschungsbereichen einschließlich Life Science, Materialwissenschaften, chemischer Synthese, Chromatographie, Analytik und vielen mehr..
Setzen Sie sich mit dem technischen Dienst in Verbindung.